本記事はAI創薬の一つである機械学習を用いたin silico screeningについて書かれた記事です。第5章まであり、すべての内容が理解できると、目的の標的にあった薬物候補化合物を発見することができます。こちらの記事は第4章で第3章で整形したデータ機械学習モデルを行います!AI創薬っぽくなってきました。ぜひ皆さんもトライしてみて下さい!
macOS Ventura(13.2.1), Google colaboratory
今回は直ぐにpythonが実行できるようにGoogle colabを利用していきます。
自宅でできるin silico創薬の技術書を販売中
¥3,200 → ¥1500 今なら50%OFF!!
新薬探索を試したい方必読!
ITエンジニアである著者の視点から、wetな研究者からもdryの創薬研究をわかりやすく身近に感じられるように解説しています
自宅でできるin silico創薬の技術書を販売中
¥3,200 → ¥1,500 今なら50%OFF!!
分子ドッキングやMDシミュレーションなど、
自宅でできるin silico創薬の解析方法を解説したものになります!
AI創薬とは?
AI創薬は、人工知能(AI)技術を利用して新しい薬物を発見、開発するプロセスです。AIは大量のデータを高速に処理し、薬物の候補を予測したり、薬物相互作用を評価したりします。また、AIは薬物の効果や安全性をシミュレートすることも可能で、臨床試験の前の段階でリスクを評価することができます。これにより、薬物開発のコストと時間を大幅に削減することが期待されています。AI創薬は、薬物開発の新しいパラダイムとして注目を集め、製薬企業や研究機関で積極的に研究、導入が進められています。また、バイオインフォマティクス、ケモインフォマティクス、機械学習、ディープラーニングなどの技術が組み合わされ、薬物開発のプロセスを革新しています。さらに、AI創薬は個人化医療の推進にも寄与し、患者にとって最適な治療法を提供する可能性を秘めています。
今回はAI創薬の中でも、in silico screeeningに焦点を当てて解説してきます。
以下の論文、記事をフォローしていきます。
NCBI – WWW Error Blocked Diagnostic
論文と公共データベースを使って無料で始めるAI創薬 – Qiita
この論文では新型コロナウイルスの標的タンパク質である3C-like プロテアーゼ(メインプロテアーゼ:Mpro)に対して、公共データベースにある化合物でスクリーニングを行い、最終的に41個のの化合物を薬物候補化合物としています。
AI創薬によるIn silico screeningの流れ
以下のような流れで行っていきます。全5章となります。
- 公共データベース(ChEMBL)からの機械学習の学習データを収集
- スクレイピングによる公共データベース(PDB)からの機械学習の学習データを収集
- 機械学習の学習データの整形
- 整形データを用いた予測モデルの作成(本記事)
- 予測モデルによる候補化合物のin silico screening
データの準備とモデルの訓練
まずはこれまでのデータを用いて、モデルを作成していきます。
流れは以下の通りです。
- ライブラリのインポート、評価関数の定義
- ハイパーパラメータチューニング
- ランダムフォレストによるモデルの作成
- モデルの保存
Google Colabのファイルに第3章で作成したfingerprint.csvがある状態で始めます。
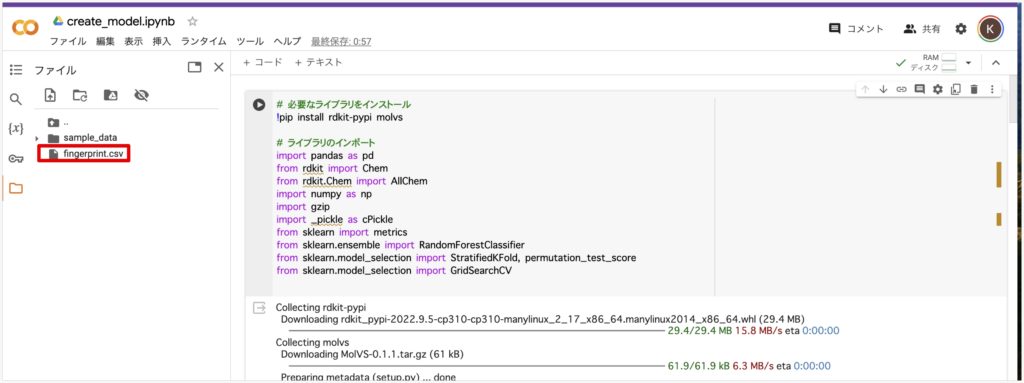
まずはコードを実行し、必要なライブラリをインポートしてください。
# 必要なライブラリをインストール
!pip install rdkit-pypi molvs
# ライブラリのインポート
import pandas as pd
from rdkit import Chem
from rdkit.Chem import AllChem
import numpy as np
import gzip
import _pickle as cPickle
from sklearn import metrics
from sklearn.ensemble import RandomForestClassifier
from sklearn.model_selection import StratifiedKFold, permutation_test_score
from sklearn.model_selection import GridSearchCV
!pip install rdkit-pypi molvs: これは、化学情報学および薬物設計に広く使用されるRDKitライブラリと、化合物の正規化や標準化に使われるMolVSライブラリをインストールするコマンドです。Pythonのパッケージマネージャーであるpipを使用しています。import pandas as pd: データ分析に広く使用されるPandasライブラリをインポートしています。Pandasはデータの操作や分析に便利な関数やデータ構造を提供します。from rdkit import Chemとfrom rdkit.Chem import AllChem: RDKitライブラリから必要なモジュールをインポートしています。これらは化合物の処理や分子間の操作に使用されます。import numpy as np: 数値計算に使われるNumPyライブラリをインポートしています。NumPyは高性能な数学的演算や配列操作を提供します。import gzipとimport _pickle as cPickle: データの圧縮・非圧縮とシリアライズ(オブジェクトの保存・読み込み)に使われる標準ライブラリです。from sklearn import metrics: scikit-learnライブラリからメトリクスモジュールをインポートしています。これはモデルの性能評価に使われる関数を提供します。from sklearn.ensemble import RandomForestClassifier: scikit-learnライブラリからランダムフォレスト分類器をインポートしています。from sklearn.model_selection import StratifiedKFold, permutation_test_score, GridSearchCV: scikit-learnライブラリからモデル選択と評価に関連するモジュールをインポートしています。これには、層化K分割交差検証、パーミュテーションテスト、グリッドサーチなどが含まれます。
このコードセクションは、機械学習モデルの訓練と評価、特に化学情報学の分野で使用されるツールや手法に関連しています。
続いて、以下のコードを実行してください。
def calc_metrics_derived_from_confusion_matrix(metrics_name, y_true, y_predict):
tn, fp, fn, tp = metrics.confusion_matrix(y_true, y_predict).ravel()
# PPV, precision
# TP / TP + FP
if metrics_name in ["PPV", "precision"]:
return tp / (tp + fp)
# NPV
# TN / TN + FN
if metrics_name in ["NPV"]:
return tn / (tn + fn)
# sensitivity, recall, TPR
# TP / TP + FN
if metrics_name in ["sensitivity", "recall", "TPR"]:
return tp / (tp + fn)
# specificity
# TN / TN + FP
if metrics_name in ["specificity"]:
return tn / (tn + fp)
この関数 calc_metrics_derived_from_confusion_matrix は、混同行列(confusion matrix)から派生した様々な性能指標を計算するために使われています。混同行列は分類モデルの性能を評価する際に用いられ、True Positive (TP), False Positive (FP), True Negative (TN), False Negative (FN) の4つの要素から構成されます。関数では、指定された性能指標に基づいて、以下の値を計算します:
- PPV (Positive Predictive Value) / Precision:
- 計算式: TP / (TP + FP)
- 正と予測されたケースの中で実際に正だった割合を示します。
- NPV (Negative Predictive Value):
- 計算式: TN / (TN + FN)
- 負と予測されたケースの中で実際に負だった割合を示します。
- 感度 (Sensitivity) / 再現率 (Recall) / TPR (True Positive Rate):
- 計算式: TP / (TP + FN)
- 実際に正のケースのうち、正と予測された割合を示します。
- 特異性 (Specificity):
- 計算式: TN / (TN + FP)
- 実際に負のケースのうち、負と予測された割合を示します。
この関数は、特に分類モデルの性能を多角的に評価する際に重要で、異なる種類のエラー(例えば、偽陽性と偽陰性)に対するモデルの感度を理解するのに役立ちます。
混合行列について、もっと理解したい方は参考文献をご覧ください。
def calc_metrics(metrics_name, y_true, y_predict):
if metrics_name == "accuracy":
return metrics.accuracy_score(y_true, y_predict)
if metrics_name == "ba":
return metrics.balanced_accuracy_score(y_true, y_predict)
if metrics_name == "roc_auc":
return metrics.roc_auc_score(y_true, y_predict)
if metrics_name == "kappa":
return metrics.cohen_kappa_score(y_true, y_predict)
if metrics_name == "mcc":
return metrics.matthews_corrcoef(y_true, y_predict)
if metrics_name == "precision":
return metrics.precision_score(y_true, y_predict)
if metrics_name == "recall":
return metrics.recall_score(y_true, y_predict)
この calc_metrics 関数は、与えられた名前に基づいて、様々な分類モデルの性能指標を計算します。関数は、予測値 (y_predict) と実際の値 (y_true) を比較し、指定された指標に基づいてスコアを返します。ここで計算される各性能指標の意味は以下の通りです。
- Accuracy (精度):
- モデルが正しく予測したサンプルの割合。全体的な予測の正確性を示します。
- Balanced Accuracy (バランス精度, BA):
- クラスの不均衡を考慮した精度。各クラスのリコールの平均値を示します。
- ROC AUC (Receiver Operating Characteristic Area Under Curve):
- モデルがランダムに選んだ正のサンプルをランダムに選んだ負のサンプルより高い確率でランク付けする能力を示します。値は0から1までで、1に近いほど良い。
- Kappa (カッパ統計量):
- 実際のクラス分布と予測がどれだけ一致するかを示す統計量。ランダムな一致を考慮に入れます。
- MCC (Matthews Correlation Coefficient, マシューズ相関係数):
- 分類の品質を示す指標で、-1(完全に不一致)から+1(完全な一致)までの値を取ります。
- Precision (精度):
- 正と予測されたサンプルの中で実際に正だったサンプルの割合。
- Recall (再現率):
- 実際に正のサンプルの中で正と予測されたサンプルの割合。
これらの指標は、モデルの性能を様々な観点から評価するために重要です。例えば、精度は全体的な性能を示しますが、クラスの不均衡がある場合はバランス精度やマシューズ相関係数の方がより有用です。ROC AUCはモデルの判別能力を示し、カッパ統計量はランダムな予測との比較を提供します。精度と再現率は、特に偽陽性と偽陰性のトレードオフを考慮する際に重要です。
続いて、以下のコードを実行してください。
input_path = "/content/fingerprint.csv"
output_model_path = "/content/output_model.pkl.gz"
output_report_path = "/content/output_report.csv"
df = pd.read_csv(input_path, index_col=0)
print(df.shape)
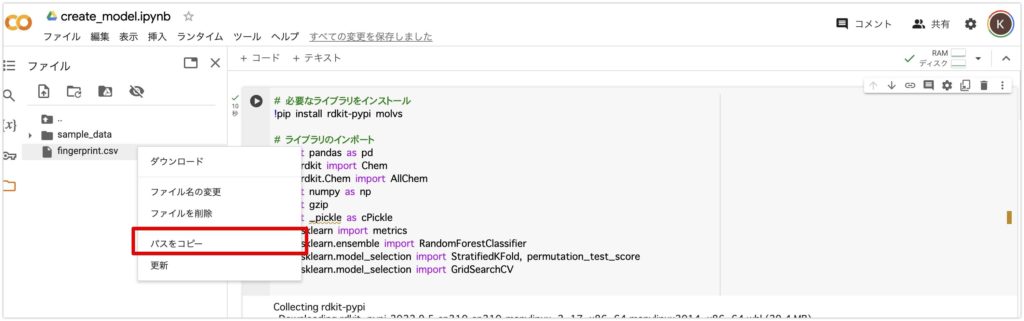
パスは以下でコピーできます。fingerpront.csvを右クリックするとパスをコピーと出てくるので、
これを上のinput_path = “この中に入れる”に入れてください。output_model_path、output_report_pathに関しては、ファイルは勝手に生成されるので、contentの後にファイル名を記載するだけで大丈夫です。
このコードは、CSVファイルからデータを読み込み、そのデータフレームの形状(行と列の数)を表示するためのものです。以下の各ステップについて説明します。
input_path, output_model_path, output_report_path はそれぞれ、入力データファイル、モデルの出力ファイル、レポートの出力ファイルへのパスを保持しています。
df = pd.read_csv(input_path, index_col=0) は、input_path で指定されたCSVファイルをPandasのデータフレームとして読み込んでいます。ここで index_col=0 は、ファイルの最初の列をデータフレームのインデックスとして使用することを指定しています。
print(df.shape) は、データフレーム df の形状を表示します。これにより、データフレームに含まれる行数と列数を知ることができます。
(101, 2049) と出力されると思います。
続いて、次のコードを実行してください。
y_train = df['outcome'].to_numpy()
print(y_train)
X_train = df.iloc[:, 1:]
print(y_train.shape)
print(X_train.shape)
y_train = df['outcome'].to_numpy():
- この行は、DataFrame
dfの'outcome'列を取り出し、NumPy配列に変換しています。
X_train = df.iloc[:, 1:]:
- この行は、DataFrame
dfの最初の列(0番目の列)を除いたすべての列を選択し、**X_train**としています。
これにより、モデルに与える入力(特徴量)と出力(ラベル)が準備と確認をしています。
次に以下を実行してください。終了するのには10分くらいかかります。
# Number of trees in random forest
n_estimators = [100, 250, 500, 750, 1000]
max_features = ['auto', 'sqrt']
criterion = ['gini', 'entropy']
class_weight = [None,'balanced',
{0:.9, 1:.1}, {0:.8, 1:.2}, {0:.7, 1:.3}, {0:.6, 1:.4},
{0:.4, 1:.6}, {0:.3, 1:.7}, {0:.2, 1:.8}, {0:.1, 1:.9}]
random_state = [24]
# Create the random grid
param_grid = {'n_estimators': n_estimators,
'max_features': max_features,
'criterion': criterion,
'random_state': random_state,
'class_weight': class_weight}
# setup model building
rf = GridSearchCV(RandomForestClassifier(), param_grid, n_jobs=-1, cv=5, verbose=1)
rf.fit(X_train, y_train)
print()
print('Best params: %s' % rf.best_params_)
print('Score: %.2f' % rf.best_score_)
このコードセクションは、ランダムフォレスト分類器のハイパーパラメータチューニングを行っています。具体的な内容は以下の通りです。
ランダムフォレストのハイパーパラメータ
n_estimators: これはランダムフォレストにおける決定木の数を指定します。木の数が多いほど、モデルは通常精度が向上しますが、計算時間も長くなり、過学習のリスクも高まる可能性があります。max_features: 各決定木で使用する特徴量の最大数を指定します。これにより、モデルの多様性に影響を与え、過学習を防ぐことができます。criterion: 木を分割する際に使用する品質の基準を指定します。一般的にはジニ不純度(gini)またはエントロピー(entropy)が使用されます。class_weight: クラスの不均衡に対処するために、特定のクラスに重みを付けます。これは、特にデータセットが不均衡な場合に重要です。random_state: モデルのランダムな動作を制御します。同じ値を使用すると、実行のたびに同じ結果が得られるため、結果の再現性が保証されます。
グリッドサーチ
GridSearchCV: scikit-learnのこの機能は、指定されたパラメータの組み合わせに基づいて、最適なパラメータ設定を見つけます。これにより、交差検証を用いてモデルの性能を評価し、最良のパラメータ組み合わせを選択します。param_grid: チューニングするパラメータの組み合わせを指定します。n_jobs=-1: すべてのCPUコアを使用して計算を行います。cv=5: 5回の交差検証を意味します。これは、データセットを5つの部分に分割し、それぞれの部分を一度ずつテストデータとして使用し、残りを訓練データとして使用します。
モデルの訓練と最適パラメータの表示
rf.fit(X_train, y_train): 訓練データセットを用いてモデルを訓練し、最適なパラメータを見つけます。print('Best params: %s' % rf.best_params_): 最適なパラメータの組み合わせを表示します。print('Score: %.2f' % rf.best_score_): 最適なパラメータに基づくモデルのスコア(通常は平均交差検証スコア)を表示します。
このプロセスにより、ランダムフォレストモデルの性能を最大化するためのハイパーパラメータの組み合わせを決定できます。ハイパーパラメータチューニングは、特に複雑なデータセットやモデリングタスクにおいて、モデルの一般化能力と性能を向上させる重要なステップです。
rf_best = RandomForestClassifier(**rf.best_params_, n_jobs=-1)
rf_best.fit(X_train, y_train)
#一旦できたモデルを保存
with gzip.GzipFile(output_model_path, 'w') as f:
cPickle.dump(rf_best, f)
with gzip.GzipFile(output_model_path, 'r') as f:
rf_best = cPickle.load(f)
このコードセクションでは、最適なパラメータを持つランダムフォレストモデルを訓練し、その後でモデルをファイルに保存し、再度読み込んでいます。具体的な手順は以下の通りです。
モデルの訓練
rf_best = RandomForestClassifier(**rf.best_params_, n_jobs=-1):RandomForestClassifierを初期化し、GridSearchCVによって見つかった最適なハイパーパラメータ(rf.best_params_)を使用しています。- **
*rf.best_params_**は、辞書形式のハイパーパラメータをアンパックして分類器に渡しています。 - **
n_jobs=-1**は、モデル訓練に利用可能なすべてのCPUコアを使用することを指定しています。
rf_best.fit(X_train, y_train):- 訓練データセットを使用してランダムフォレストモデルを訓練しています。
モデルの保存と読み込み
with gzip.GzipFile(output_model_path, 'w') as f:- モデルをGzip圧縮ファイルとして保存します。**
output_model_path**は保存するファイルのパスです。 - **
'w'**は書き込みモードを意味します。
- モデルをGzip圧縮ファイルとして保存します。**
cPickle.dump(rf_best, f):- モデル(
rf_best)をcPickleを使ってシリアライズし、開いたファイルに書き込みます。
- モデル(
with gzip.GzipFile(output_model_path, 'r') as f:- 保存されたモデルファイルを読み込みモードで開きます。
rf_best = cPickle.load(f):- ファイルからモデルをデシリアライズして読み込みます。
このプロセスは、訓練されたモデルを保存し、後で必要なときに簡単に再利用できるようにする一般的な方法です。モデルの保存と読み込みは、特に大規模なデータセットや時間がかかる訓練プロセスを持つ場合に便利です。また、保存されたモデルは他の環境で再利用することができ、再現性や共有の面で有用です。
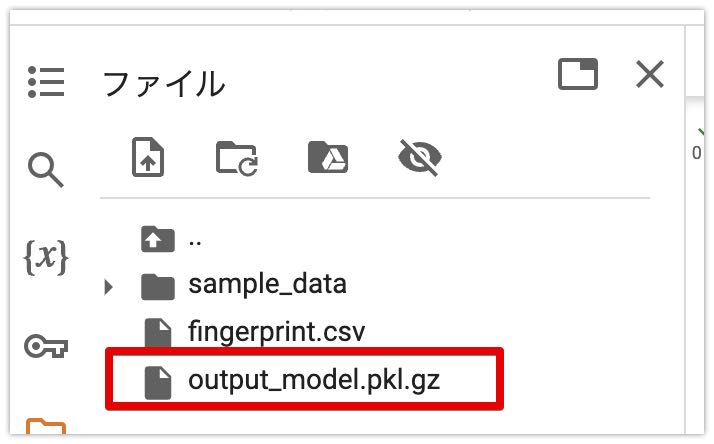
以上でモデルは完成です。ここで完成したモデルを次章以降で使っていきます。
ファイルに新しく**output_model.pkl.gz** が生成していると思います。
モデルの評価と統計指標の計算
以下はモデルの評価なので、飛ばしても次章以降には影響ありません。ですが、ハイパーパラメータを調整した場合は適宜実行すると良いでしょう。
流れは以下の通りです。
- 5分割交差法の実装
- 全データに対する5分割交差法の評価
- より信頼性の高いデータに対する5分割交差法の評価
# Params
pred = []
ad = []
pred_proba = []
index = []
cross_val = StratifiedKFold(n_splits=5)
# Do 5-fold loop
for train_index, test_index in cross_val.split(X_train, y_train):
fold_model = rf_best.fit(X_train.iloc[train_index], y_train[train_index])
fold_pred = rf_best.predict(X_train.iloc[test_index])
fold_ad = rf_best.predict_proba(X_train.iloc[test_index])
pred.append(fold_pred)
ad.append(fold_ad)
pred_proba.append(fold_ad[:, 1])
index.append(test_index)
threshold_ad = 0.70
このコードのセクションは、層化5分割交差検証(Stratified K-Fold Cross Validation)を用いたランダムフォレストモデルの性能評価に関連しています。以下の手順で進められます。
パラメータの初期化
pred,ad,pred_proba,indexはそれぞれ、予測結果、予測確率、特定クラスの予測確率、テストセットのインデックスを保持するための空リストです。cross_val = StratifiedKFold(n_splits=5)は、データセットを5つの異なるサブセットに分割し、それぞれのサブセットが元のデータセットのクラス比率を反映するようにします。
5分割交差検証の実行
for train_index, test_index in cross_val.split(X_train, y_train):このループは、訓練データとテストデータのインデックスを生成し、データを分割します。fold_model = rf_best.fit(X_train.iloc[train_index], y_train[train_index])は、訓練データに基づいてモデルを訓練します。fold_pred = rf_best.predict(X_train.iloc[test_index])は、テストデータに対するモデルの予測を生成します。fold_ad = rf_best.predict_proba(X_train.iloc[test_index])は、テストデータの各サンプルが各クラスに属する確率を計算します。
予測結果の保存
pred.append(fold_pred)は、各分割の予測結果をリストに追加します。ad.append(fold_ad)は、予測確率をリストに追加します。pred_proba.append(fold_ad[:, 1])は、特定クラス(通常はポジティブクラス)の予測確率をリストに追加します。index.append(test_index)は、各分割のテストデータのインデックスをリストに追加します。
予測確率の閾値設定
threshold_ad = 0.70は、活動ドメイン(AD)分析における予測確率の閾値を設定します。この閾値を超える予測確率を持つサンプルは、モデルが自信を持って予測できると判断されます。
このプロセスは、モデルの性能を評価し、異なるデータ分割での予測結果を収集するための一般的な手法を示しています。層化クロスバリデーションは特に、クラスの不均衡があるデータセットに対して適しています。また、予測確率の閾値を設定することで、特定のクラスに対する分類の厳格さを調整できます。
続いて、以下のコードを実行してください。
# Prepare results to export
fold_index = np.concatenate(index)
fold_pred = np.concatenate(pred)
fold_ad = np.concatenate(ad)
fold_pred_proba = np.concatenate(pred_proba)
fold_ad = (np.amax(fold_ad, axis=1) >= threshold_ad).astype(str)
five_fold_morgan = pd.DataFrame({'Prediction': fold_pred, 'AD': fold_ad, 'Proba': fold_pred_proba}, index=list(fold_index))
five_fold_morgan.AD[five_fold_morgan.AD == 'False'] = np.nan
five_fold_morgan.AD[five_fold_morgan.AD == 'True'] = five_fold_morgan.Prediction
five_fold_morgan.sort_index(inplace=True)
five_fold_morgan['y_train'] = pd.DataFrame(y_train)
このコードのセクションでは、交差検証によって得られたランダムフォレストモデルの予測結果を集約し、エクスポート用のデータフレームを作成しています。具体的な手順は以下の通りです。
- 結果の結合:
np.concatenate関数を使って、各分割から得られた予測結果 (pred)、予測確率 (ad)、特定クラスの予測確率 (pred_proba)、テストデータのインデックス (index) を結合します。
- 予測確率に基づく活動ドメイン (AD) の計算:
fold_ad = (np.amax(fold_ad, axis=1) >= threshold_ad).astype(str)では、予測確率が設定された閾値 (threshold_ad) 以上かどうかを判定し、結果を文字列として保存します。
- データフレームの作成:
pd.DataFrame({'Prediction': fold_pred, 'AD': fold_ad, 'Proba': fold_pred_proba}, index=list(fold_index))で、集約された予測結果を含むPandasのデータフレームを作成します。このデータフレームには「Prediction」(予測結果)、「AD」(活動ドメイン)、および「Proba」(予測確率)の列が含まれます。
- 活動ドメイン (AD) の調整:
five_fold_morgan.AD[five_fold_morgan.AD == 'False'] = np.nanでは、活動ドメインが ‘False’ と判定された場合、その値を NaN(Not a Number)に置き換えます。five_fold_morgan.AD[five_fold_morgan.AD == 'True'] = five_fold_morgan.Predictionでは、活動ドメインが ‘True’ と判定された場合、その値を予測結果の値で置き換えます。
- データフレームの整理:
five_fold_morgan.sort_index(inplace=True)で、データフレームをインデックスに基づいてソートします。five_fold_morgan['y_train'] = pd.DataFrame(y_train)で、元の訓練データのラベルをデータフレームに追加します。
このコードは、交差検証の結果を集約し、さらなる分析やレポート作成のために整理する一般的な手法を示しています。活動ドメインの計算は、モデルの予測が信頼できる範囲を示すために用いられ、データフレームにこれらの情報を統合して保存することで、モデルの性能評価や比較が容易になります。
続いて以下のコードを実行してください。
# morgan stats
all_datas = []
data = []
data.append(calc_metrics("accuracy", five_fold_morgan['y_train'], five_fold_morgan['Prediction']))
data.append(calc_metrics("ba", five_fold_morgan['y_train'], five_fold_morgan['Prediction']))
data.append(calc_metrics("roc_auc", five_fold_morgan['y_train'], five_fold_morgan['Proba']))
data.append(calc_metrics("kappa", five_fold_morgan['y_train'], five_fold_morgan['Prediction']))
datas.append(calc_metrics("MCC", five_fold_morgan['y_train'], five_fold_morgan['Prediction']))
datas.append(calc_metrics("precision", five_fold_morgan['y_train'], five_fold_morgan['Prediction']))
datas.append(calc_metrics("recall", five_fold_morgan['y_train'], five_fold_morgan['Prediction']))
datas.append(calc_metrics_derived_from_confusion_matrix("sensitivity", five_fold_morgan['y_train'], five_fold_morgan['Prediction']))
datas.append(calc_metrics_derived_from_confusion_matrix("PPV", five_fold_morgan['y_train'], five_fold_morgan['Prediction']))
datas.append(calc_metrics_derived_from_confusion_matrix("specificity", five_fold_morgan['y_train'], five_fold_morgan['Prediction']))
datas.append(calc_metrics_derived_from_confusion_matrix("NPV", five_fold_morgan['y_train'], five_fold_morgan['Prediction']))
datas.append(1)
all_datas.append(datas)
このコードのセクションは、交差検証によって得られた予測結果に基づいて、ランダムフォレストモデルのさまざまな性能指標を計算しています。具体的には、次のような処理を行っています。
all_datas と datas は、計算された各種指標を格納するためのリストです。
calc_metricsとcalc_metrics_derived_from_confusion_matrix
関数を使用して、以下の指標を計算しています。
- 精度 (Accuracy)
- バランス精度 (Balanced Accuracy, BA)
- ROC AUC (Receiver Operating Characteristic Area Under the Curve)
- カッパ統計量 (Kappa)
- マシューズ相関係数 (Matthews Correlation Coefficient, MCC)
- 精度 (Precision)
- 再現率 (Recall)
- 感度 (Sensitivity)
- PPV (Positive Predictive Value)
- 特異性 (Specificity)
- NPV (Negative Predictive Value)
続いて、以下のコードを実行してください。
# morgan AD stats
morgan_ad = five_fold_morgan.dropna(subset=['AD'])
morgan_ad['AD'] = morgan_ad['AD'].astype(int)
coverage_morgan_ad = len(morgan_ad['AD']) / len(five_fold_morgan['y_train'])
datas = []
datas.append(calc_metrics("accuracy", morgan_ad['y_train'], morgan_ad['AD']))
datas.append(calc_metrics("ba", morgan_ad['y_train'], morgan_ad['AD']))
datas.append(calc_metrics("roc_auc", morgan_ad['y_train'], morgan_ad['Proba']))
datas.append(calc_metrics("kappa", morgan_ad['y_train'], morgan_ad['AD']))
datas.append(calc_metrics("mcc", morgan_ad['y_train'], morgan_ad['AD']))
datas.append(calc_metrics("precision", morgan_ad['y_train'], morgan_ad['AD']))
datas.append(calc_metrics("recall", morgan_ad['y_train'], morgan_ad['AD']))
datas.append(calc_metrics_derived_from_confusion_matrix("sensitivity", morgan_ad['y_train'], morgan_ad['AD']))
datas.append(calc_metrics_derived_from_confusion_matrix("PPV", morgan_ad['y_train'], morgan_ad['AD']))
datas.append(calc_metrics_derived_from_confusion_matrix("specificity", morgan_ad['y_train'], morgan_ad['AD']))
datas.append(calc_metrics_derived_from_confusion_matrix("NPV", morgan_ad['y_train'], morgan_ad['AD']))
datas.append(coverage_morgan_ad)
all_datas.append(datas)
このコードのセクションは、「morgan AD stats」と題され、ランダムフォレストモデルの予測結果に基づいて活動ドメイン(AD)分析と関連する統計指標の計算を行っています。以下の手順で処理が進められています。
- 活動ドメインデータの準備:
morgan_ad = five_fold_morgan.dropna(subset=['AD'])は、’AD’ 列にNaNが含まれている行を除外します。これにより、AD分析に適切なデータのみが選択されます。morgan_ad['AD'] = morgan_ad['AD'].astype(int)は、’AD’ 列のデータタイプを整数に変換します。
- カバレッジの計算:
coverage_morgan_ad = len(morgan_ad['AD']) / len(five_fold_morgan['y_train'])は、活動ドメインに含まれるサンプルの割合を計算します。これは、モデルがどの程度のサンプルを「信頼できる」と判断しているかを示します。
- AD統計の計算:
- さまざまな性能指標(精度、バランス精度、ROC AUC、カッパ統計量、マシューズ相関係数、精度、再現率、感度、PPV、特異性、NPV)を
calc_metricsとcalc_metrics_derived_from_confusion_matrix関数を使って計算しています。
- さまざまな性能指標(精度、バランス精度、ROC AUC、カッパ統計量、マシューズ相関係数、精度、再現率、感度、PPV、特異性、NPV)を
- 結果の保存:
- 計算された指標とカバレッジは
datasリストに追加されます。
- 計算された指標とカバレッジは
- 全データへの追加:
- 最終的に、
datasリストがall_datasリストに追加され、これにより全ての統計データが一つのリストにまとめられます。
- 最終的に、
このコードは、ランダムフォレストモデルの信頼性と性能を評価するために、活動ドメイン分析を行い、さまざまな指標を用いてこれを補完する方法を示しています。これにより、モデルが特定のサンプルに対してどの程度信頼できる予測を行っているか、またその予測の質がどの程度かを評価できます。
続いて、以下を実行してください。
# print stats
print('\\033[1m' + '5-fold External Cross Validation Statistical Characteristcs of QSAR models developed morgan' + '\\n' + '\\033[0m')
morgan_5f_stats = pd.DataFrame(all_datas, columns=["accuracy", "ba", "roc_auc", "kappa", "mcc", "precision", "recall",
"sensitivity", "PPV", "specificity", "NPV", "Coverage"])
morgan_5f_stats.to_csv(output_report_path)
print(morgan_5f_stats)
このコードのセクションでは、ランダムフォレストモデルの性能評価に関連する統計データを表示し、CSVファイルに出力しています。以下の手順で進められています。
- 強調表示のためのエスケープシーケンス:
'\\033[1m'と'\\033[0m'はANSIエスケープシーケンスで、テキストを強調(太字)するために使われています。'5-fold External Cross Validation Statistical Characteristcs of QSAR models developed morgan'というテキストは、計算された統計データの説明を提供します。
- データフレームの作成:
pd.DataFrame(all_datas, columns=["accuracy", "ba", "roc_auc", "kappa", "mcc", "precision", "recall", "sensitivity", "PPV", "specificity", "NPV", "Coverage"])により、計算された統計データを含むPandasのデータフレームが作成されます。- このデータフレームには各種性能指標(精度、バランス精度、ROC AUCなど)とカバレッジが含まれます。
- CSVファイルへの出力:
morgan_5f_stats.to_csv(output_report_path)は、計算された統計データをCSVファイルに保存します。ここでoutput_report_pathは保存するファイルのパスです。
- 統計データのコンソール出力:
print(morgan_5f_stats)で、作成されたデータフレームがコンソールに出力されます。
このコードは、ランダムフォレストモデルの詳細な性能評価を文書化し、共有するための方法を示しています。統計データをCSVファイルに保存することで、後での分析や他の研究者との共有が容易になります。また、コンソールへの出力は、プログラム実行時に即座に結果を確認するために役立ちます。
以下のように出力されます。
5-fold External Cross Validation Statistical Characteristcs of QSAR models developed morgan
accuracy ba roc_auc kappa mcc precision recall \\
0 0.762376 0.697403 0.813853 0.429379 0.448000 0.73913 0.485714
1 0.807692 0.724506 0.828854 0.492628 0.507046 0.75000 0.521739
sensitivity PPV specificity NPV Coverage
0 0.485714 0.73913 0.909091 0.769231 1.000000
1 0.521739 0.75000 0.927273 0.822581 0.772277
0行目のものが、morgan statsであり、全データに対する5分割交差検証を行っています。
1行目のものが、morgan AD statsであり、より信頼性の高いデータに対しての5分割交差検証を行っています。ですので、morgan AD statsの方が少しだけ良い値になっています。
最後に以下を実行してください。
# Y randomization
permutations = 20
score, permutation_scores, pvalue = permutation_test_score(rf_best, X_train, y_train,
cv=5, scoring='balanced_accuracy',
n_permutations=permutations,
n_jobs=-1,
verbose=1,
random_state=24)
print('True score = ', score.round(2),
'\\nY-randomization = ', np.mean(permutation_scores).round(2),
'\\np-value = ', pvalue.round(4))
このコードのセクションでは、Yランダマイゼーションテスト(または順列重要度テスト)を使用して、ランダムフォレストモデルの性能が偶然によるものでないことを確認しています。具体的なプロセスは以下の通りです。
- テストのセットアップ:
permutations = 20は、ランダマイゼーションテストで使用する順列の数を設定しています。ここでは20回の順列を生成します。permutation_test_score関数は、ターゲット変数y_trainをランダムに並べ替え(シャッフル)し、その結果に基づいてモデルのスコアを計算します。これは20回繰り返されます。
- テストの実行:
rf_bestはテストに使用されるランダムフォレストモデルです。X_trainとy_trainはそれぞれ特徴量とターゲット変数です。cv=5は5分割のクロスバリデーションを意味します。scoring='balanced_accuracy'は、バランスの取れた精度をスコアリング基準として使用します。n_jobs=-1は、利用可能なすべてのCPUコアを使用することを指定します。
- 結果の表示:
scoreはモデルの実際のスコアを示します。np.mean(permutation_scores).round(2)はランダマイズされたデータセットで計算されたスコアの平均値です。pvalueは、モデルのスコアがランダムな結果によるものである確率を示します。
次のように出力されます。
True score = 0.7
Y-randomization = 0.48
p-value = 0.0476
結果の解釈は以下の通りです。
- True Score (実際のスコア) = 0.7:
- このスコアは、モデルがテストデータに対して得た実際の性能指標です。この場合、0.7はモデルがそれなりに高い性能を持っていることを示しています(スコアは通常0から1の間で、1に近いほど良い)。しかしまだ改善の余地がある可能性があります。特に、特徴選択、モデルのパラメータチューニング、データの前処理など、さらなる最適化が必要かもしれません。
- Y-randomization Score (Yランダマイゼーションスコア) = 0.48:
- Yランダマイゼーションスコアは、データのラベルをランダムに並べ替えた後のモデルの性能を示しています。0.48はランダムな予測に近い値であり、モデルが単なるランダム推測よりも優れた性能を持っていることを示しています。
- P-value (p値) = 0.0476:
- p値は、モデルの性能がランダムなチャンスによるものかどうかを評価します。一般に、p値が0.05以下の場合、モデルの性能が偶然によるものではないと統計的に有意とみなされます。ここでのp値は0.0476であり、0.05をわずかに下回っているため、モデルの性能は偶然によるものではないと考えられます。
まだまだ改善の余地があるモデルですが、今回はこちらを用いて、スクリーニングをしていこうと思います。
最後に
いかがでしたでしょうか?モデルの学習、評価などようやく機械学習で行うことが出てきました!予測モデルが出来ましたので、いよいよ最終章ではサンプルデータを使って、in silicoスクリーニングをしていきます。
参考文献
混合行列について
【わかりやすく解説】混同行列の概要をかみくだいて説明します – DXコンサルの日進月歩奮闘記
混同行列と評価指標についてわかりやすく解説&Python実装!|スタビジ
謝辞
この記事は以下の論文のフォローとなります。機械学習とAI創薬についてわかりやすく書かれています。このような論文を書いて下った筆者らに感謝を申し上げます。
NCBI – WWW Error Blocked Diagnostic
この記事を書くにあたり、**@kimisyo(晶 公畑)**さんの記事が大変参考になりました。ありがとうございます。





