分子ドッキングは、薬品や生体分子の相互作用を予測するためのコンピュータシミュレーション手法です。この記事では、分子ドッキングの基本原理や手法、具体的な応用例について解説しています。HDockという手法に焦点を当てて紹介しています。
学ぶことによるメリットとしては、新薬開発やタンパク質研究において効果的な薬物スクリーニングや設計が可能となります。また、タンパク質の相互作用解明や薬物の最適化にも役立ちます。さらに、計算科学と生命科学の知識を統合することで、より深い理解と応用が可能となります。
ぜひトライしてみてください!
macOS Ventura(13.2.1)
自宅でできるin silico創薬の技術書を販売中
¥3,200 → ¥1500 今なら50%OFF!!
新薬探索を試したい方必読!
ITエンジニアである著者の視点から、wetな研究者からもdryの創薬研究をわかりやすく身近に感じられるように解説しています
自宅でできるin silico創薬の技術書を販売中
¥3,200 → ¥1,500 今なら50%OFF!!
分子ドッキングやMDシミュレーションなど、
自宅でできるin silico創薬の解析方法を解説したものになります!
本記事を進むにあたって、PyMOLのダウンロードをお願いします。
大阪大学の蛋白研究所からインストールの仕方が解説されています。
分子ドッキングとは?
分子ドッキングとは、薬品や生体分子(例えばタンパク質)がどのように相互作用するかを予測するコンピュータシミュレーションのことです。
特定のタンパク質と薬物がどのように結合するか、どの部分が互いに触れ合うかを計算し、その結果から薬物の効果を予測します。これは新薬開発などに非常に役立ちます。
現在様々なwebで使うことのできる分子ドッキング手法があります。
今回はその中でも、タンパク質-タンパク質ドッキングを扱うHDockについて紹介していきます。
タンパク質の下準備
まず各手法の解析の前にドッキングするタンパク質の下準備をしていきたいと思います。今回はモデルのタンパク質としてProtein Data Bank(PDB)の番号8DHAであるレプチンとその受容体を使います。
レプチンは、脂肪組織に由来するタンパク質ホルモンであり、体重とエネルギーバランスの調節に重要な役割を果たしています。レプチンは脂肪細胞から血液中に放出され、脳の視床下部に存在する特定の受容体に結合することで、食欲を抑制し代謝を調節する作用を持っています。
薬物としてのレプチンの魅力は、肥満治療における可能性です。レプチンの欠乏または抵抗は、肥満の原因となることが知られています。そのため、レプチンの補充や受容体の活性化を促進する薬剤の開発が進められています。
レプチン薬物の魅力は、食欲を抑制する効果と体重減少をもたらす可能性です。これにより、肥満者やメタボリックシンドロームの患者にとって、体重管理や疾患リスクの低減に役立つことが期待されています。さらに、レプチンは食欲を制御する神経回路にも関与しているため、食欲をコントロールする新たな治療法の開発にもつながる可能性があります。
最近ではインスリンを用いずに、レプチンを使って、血糖値を下げることにも成功しており、糖尿病への治療へも期待されています。
(PDB)の番号8DHAであるレプチンとその受容体は結合している箇所のみを実際に取り出したもので、全体は8DH8に登録されています。今回は計算量を少なくするために、番号8DHAであるレプチンとその受容体を使います。
まずPymolのタブのFile→Get PDB…に8DHAと打ち、レプチンとその受容体を表示させます。
続いてDisplay→Sequenceより全体の配列を表示します。
/C/C/1から始まる部位がレプチンに該当するので、この配列を選択します。
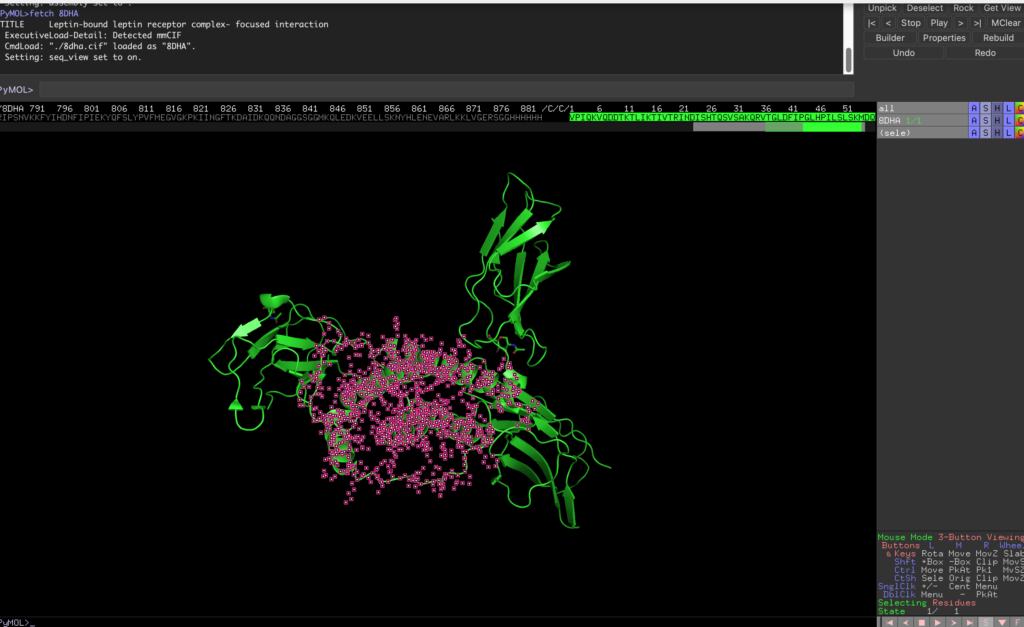
続いて、以下のコードを打ち、実行します。
save leptin.pdb, seleこれによって、選択部位がleptin.pdbとして保存されます。
続いてレプチン以外の表示されているレプチン受容体の部分を選択し、
save focus_leptinR.pdb, seleとして保存します。2-acetamido-2-deoxy-beta-D-glucopyranoseも一緒に結合していますが、こちらの部分は選択しないでください。
これによって、選択部位がfocus_leptinR.pdbとして保存されます。
これらのデータはMacintosh HD/ユーザ/自分のパソコンの名前
の階層に保存されていると思います。
以上でタンパク質の下準備は終了です。
HDockとは?
HDockは、リガンドとレセプターの間の可能な結合を探索し(ドッキング)、その結合の品質を評価する(スコアリング)という2つのステップから成り立っています。これにより、HDockはリガンドとレセプターが最も自然に結合する形を予測することができます。HDockはタンパク質-タンパク質に加え、タンパク質-DNA/RNAドッキングのためのウェブサーバーです。今回はタンパク質-タンパク質ドッキングを試していきます。
では早速HDockを試してみましょう!
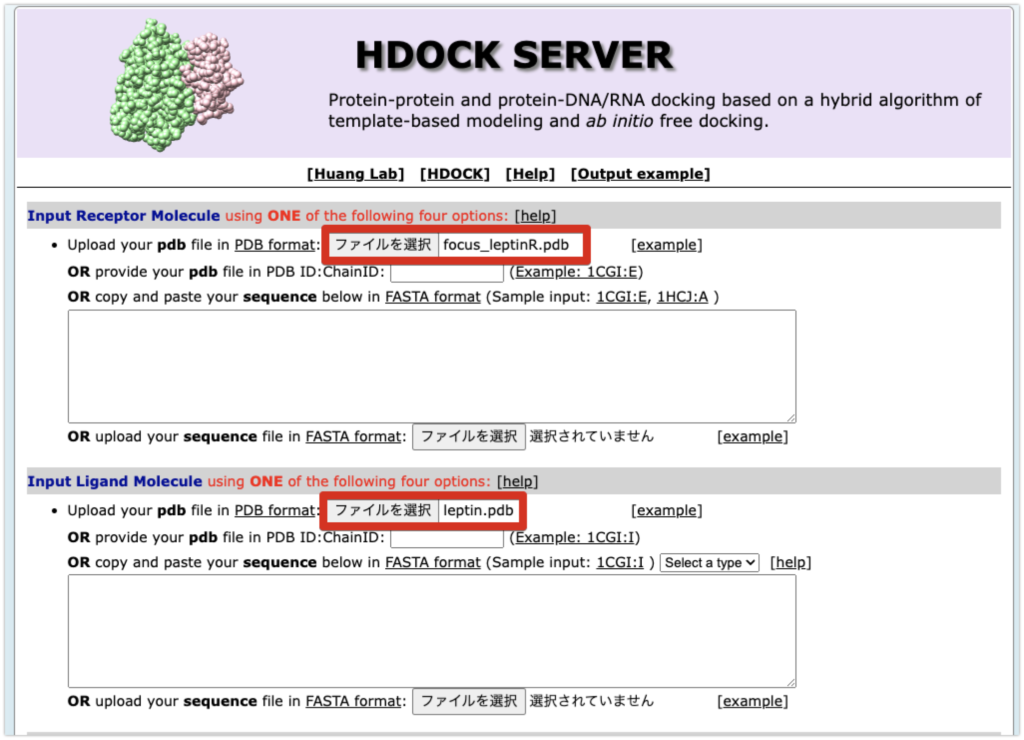
HDockのサイトにいき、以下の項目を入力してください。
- Input Receptor Molecule に
focus_leptinR.pdb - Input Ligand Moleculeに
leptin.pdb - mailアドレスとjobnameを入力してください。
- Submitを押し、しばらく待ちます。2時間ほどで結果が出てきます。
HDockのメカニズム
HDockのメカニズムについて詳しく見てみましょう。HDockは、主に2つのステップで動作します。まず、リガンドとレセプターの間の可能な結合を生成する「ドッキング」フェーズ。次に、生成された結合の品質を評価する「スコアリング」フェーズです。
- ドッキングフェーズ: このフェーズでは、HDockはリガンドとレセプターの間の可能なすべての結合を探索します。リガンドの構造を変えることで、それがレセプターにどのようにフィットするかを見るためのプロセスです。このフェーズでは、多数の可能な結合が生成されます。
- スコアリングフェーズ: 生成された結合の品質が評価されるフェーズです。HDockは、各結合がどれだけ「自然」であるか、つまり、実際の生物学的な環境で起こり得るかを評価します。これは、結合エネルギーを計算することで行われます。最も自然な結合(つまり、最も低い結合エネルギーを持つ結合)が最も良い結合とされます。
以上が、HDockの詳細なメカニズムです。
結果
次のような結果が出てきました。(サーバーでは保持が2週間ほどらしいので、自身のコンピュータにダウンロードしておきましょう。)
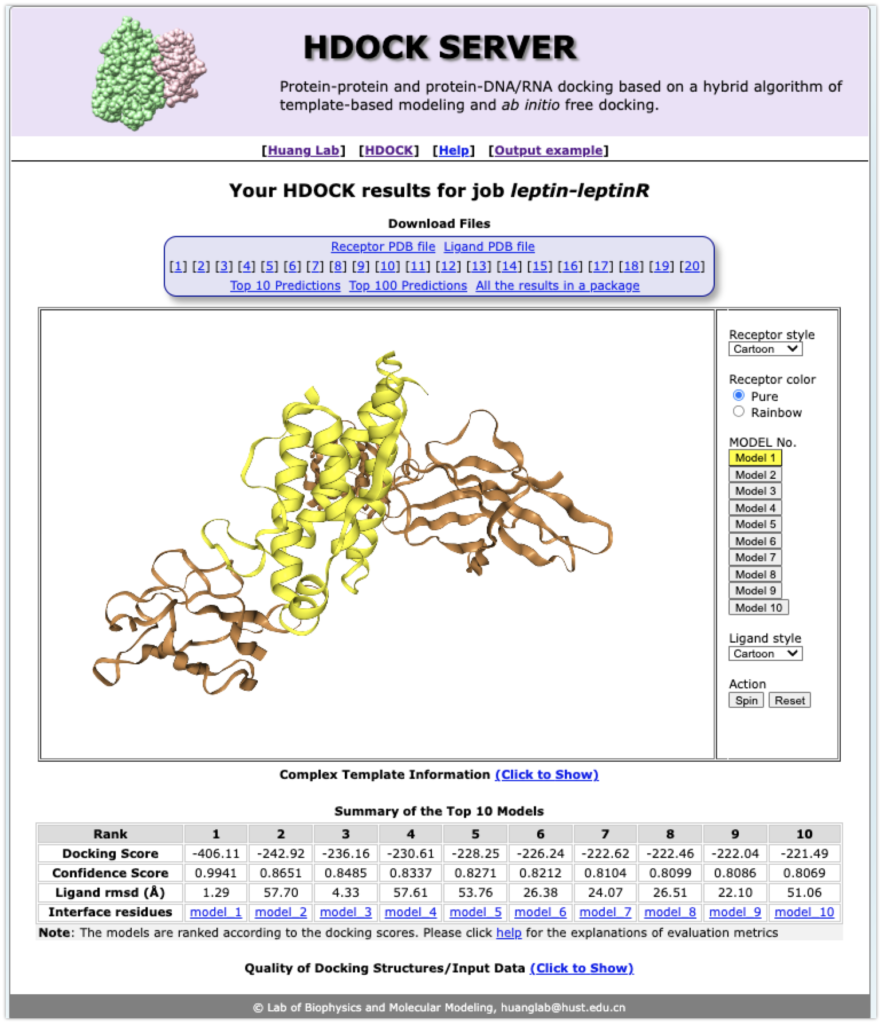
最もドッキングスコアの良いモデル1をpymolで表示します。
色を少し変更して、leptin.pdbを肌色、focus_leptinR.pdbを緑色にしています。
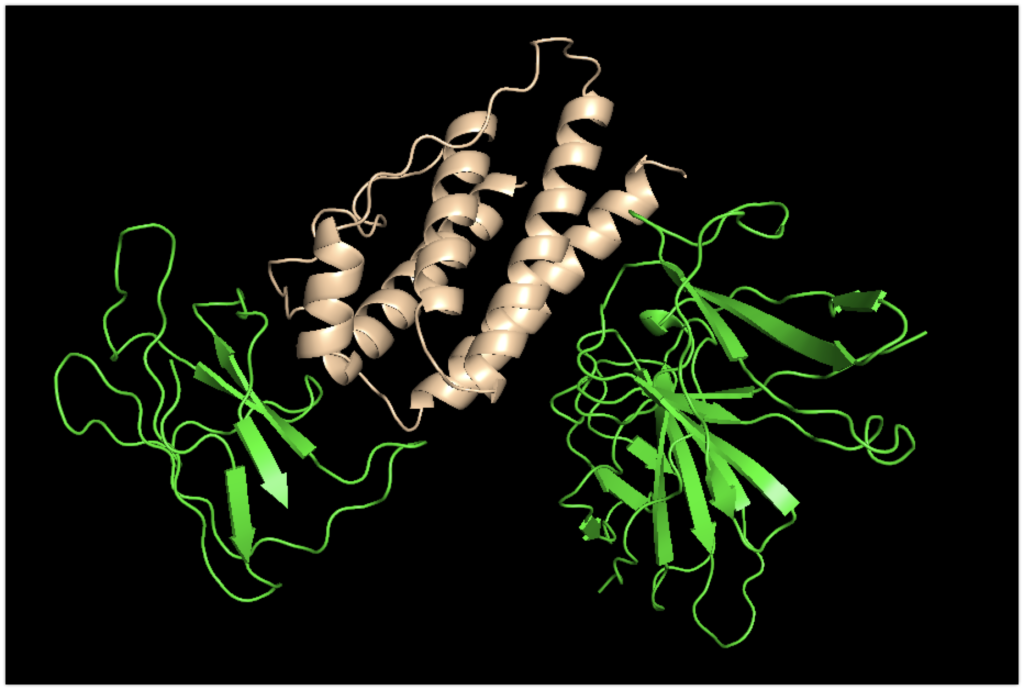
それでは元々の配列と比べてみます。
PymolのタブのFile→Get PDB…に8DHAと打ちます。
マゼンダ色のものが元々PDBに載っていた複合体の構造です。
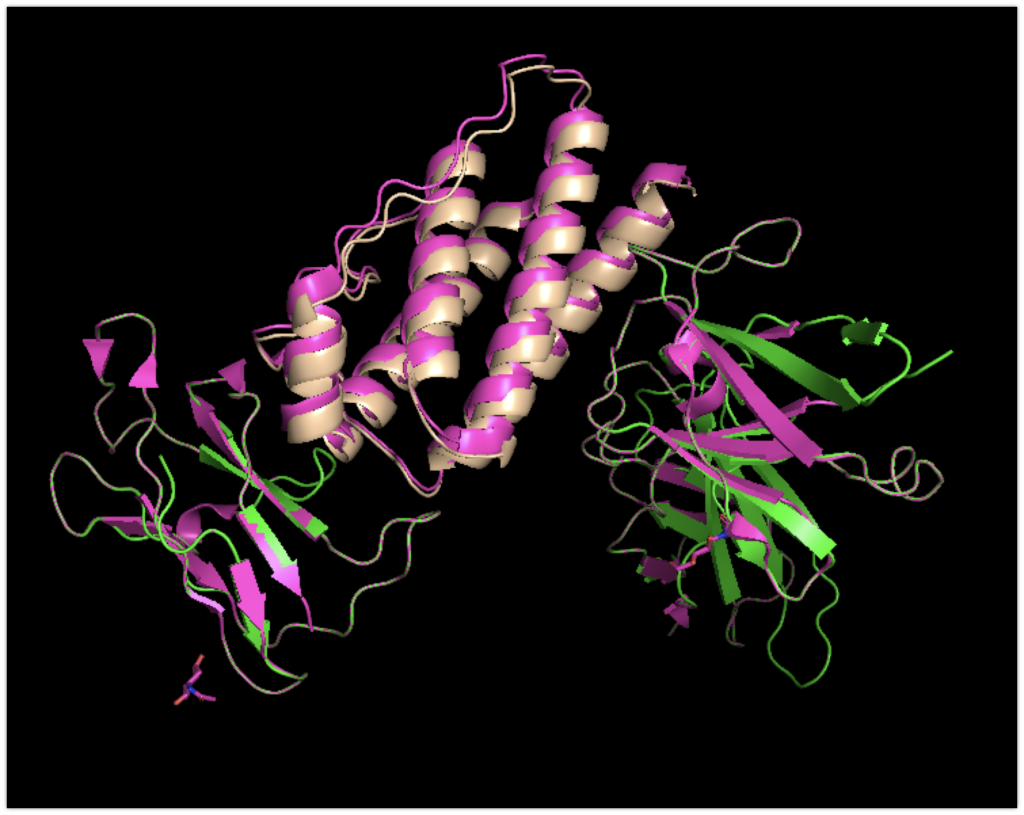
多少はずれているものの、こちらも精度良く結合していることがわかります。
最後に
いかがでしたでしょうか?本日はタンパク質ータンパク質ドッキング法の中でHDockについて取り上げてみました。こちらはサーバーで簡単にドッキングを試すことができるので、ぜひ皆さんも試してみてください。以前の記事ではClusPro、PatchDock、LZerD pairwise dockingを取り上げました。HDockも今回のタンパク質を用いて精度良くドッキングできています。
HDockはさらに、DNAとタンパク質のドッキングをすることができます。
また配列のみを入れたものでもドッキングすることができるので、非常に便利となっています。
ぜひこのような方法も試してみてください!





