本記事はin silico創薬におけるタンパク質のポケット探索の記事です。CASTpと呼ばれるツールを用いて、実際にタンパク質のくぼみ(ポケット)を探索し、薬剤の結合しうる場所を予測します。この記事を学ぶと、タンパク質のポケットが理解でき、分子ドッキングやin silicoスクリーニングに役立ちます。ぜひトライしてみてください。
macOS Ventura(13.2.1), UCSF Chimera-1.17.3-mac64
自宅でできるin silico創薬の技術書を販売中
¥3,200 → ¥1500 今なら50%OFF!!
新薬探索を試したい方必読!
ITエンジニアである著者の視点から、wetな研究者からもdryの創薬研究をわかりやすく身近に感じられるように解説しています
自宅でできるin silico創薬の技術書を販売中
¥3,200 → ¥1,800 今なら45%OFF!!
分子ドッキングやMDシミュレーションなど、
自宅でできるin silico創薬の解析方法を解説したものになります!
目次
CASTpとは
CASTpは「Computationally Automated Sites of Topological Pockets」と呼ばれるシステムで、タンパク質のポケット(タンパク質の表面にあるくぼみや穴のこと)やキャビティ(ンパク質の内部にある閉じた空間で、外部の溶媒がアクセスできない部分)を計算し、識別するためのツールです。
CASTpは、タンパク質の3D構造から特定の部分を抽出し、科学者がタンパク質の機能や反応の仕組みを理解するのを助けます。
詳しい説明についてはこちらをご覧ください。
タンパク質の下準備
今回の扱うタンパク質はsodium glucose transporter 2(SGLT2)(PDB:7VSI)になります。
SGLT2(ナトリウム-グルコース共輸送体2)は、糖尿病治療における魅力的な薬物標的です。この輸送体は、腎臓の尿細管でグルコースの再吸収を促進する役割を持ちます。SGLT2阻害剤はこのプロセスを抑制し、尿中に糖を排出させることで血糖を下げます。このメカニズムにより、インスリンに依存せずに血糖コントロールが可能となるため、特に2型糖尿病患者に有効です。またSGLT2阻害剤の一つであるフォシーがは痩せ薬としても知られています。
以下の順でタンパク質の下準備を行なってください。
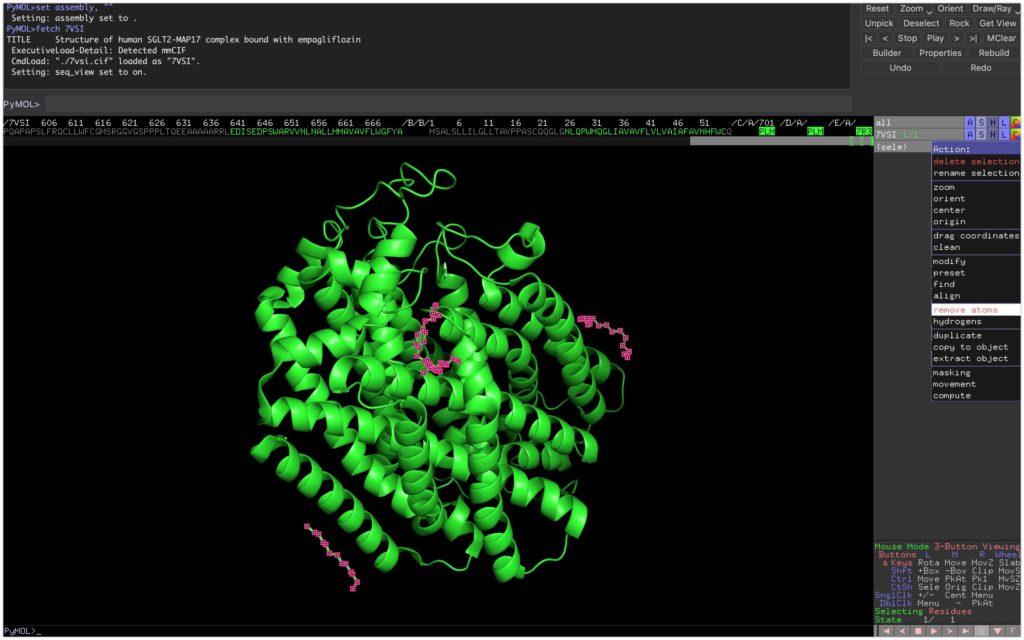
- 既に結合している低分子については消去します。上部タブのDisplay→Sequenceより配列を出します。
- アミノ酸配列の後ろに書いてある低分子を選択し、(sele)→Action→Remove atomsで低分子を消去します。
- 最後にタンパク質をpdbファイルで保存します。
CASTpによるポケット探索
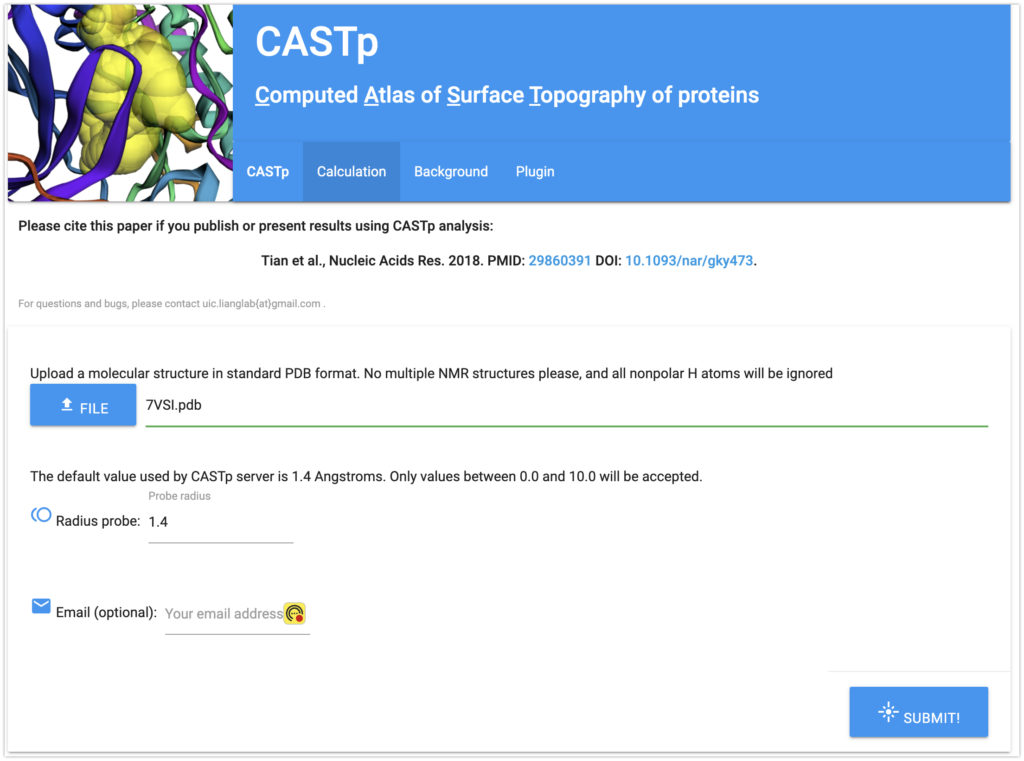
まずはCASTpにアクセスします。
Calculationのタブにいき、先ほど作成したPDBファイルをアップロードします。
そのあと、自身のメールを書いて、SUBMIT!を押してください。
結果
解析が完了すると、メールが来るので、開いてみてください。
なぜか私の場合はメールが来ずに、すぐに結果が見れました。
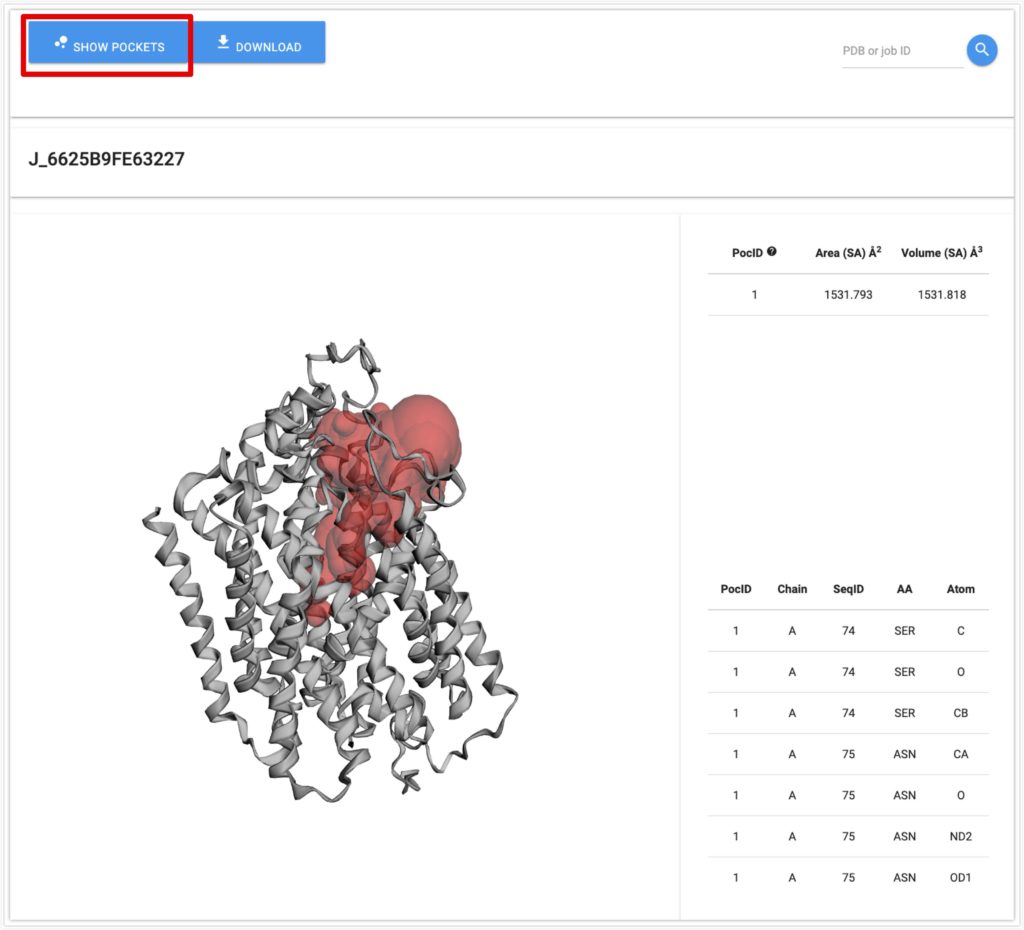
デフォルトで最も大きいポケットが表示されています。
左上のSHOW POCKETSを押すと、タンパク質の様々なポケットが表示されます。
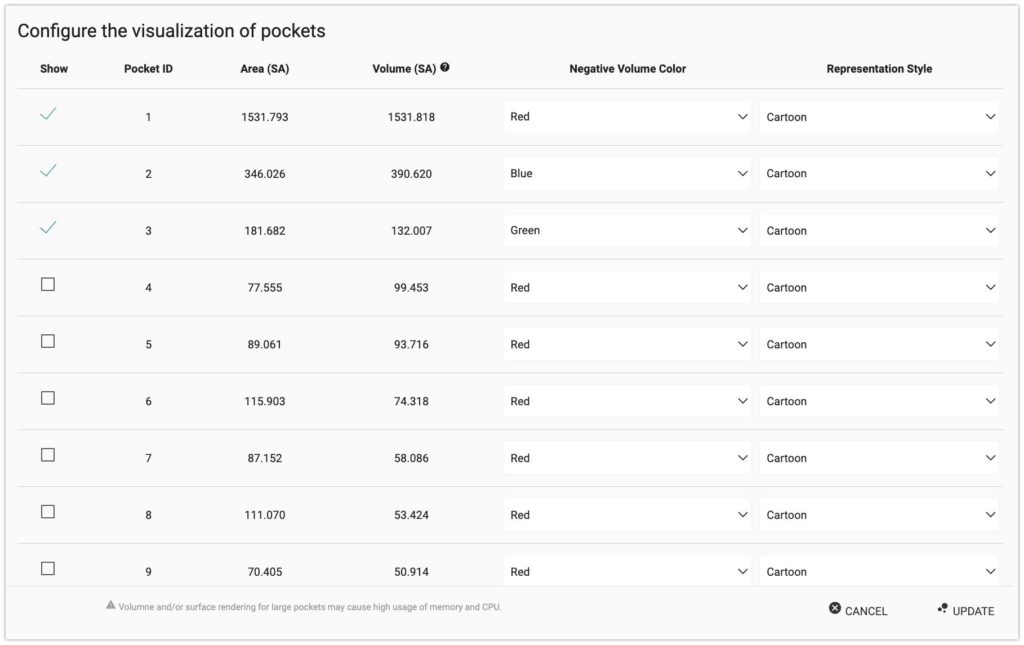
試しに上位三つを選択して、色をつけてみましょう。
赤が一番大きいポケットで、青が二番目に大きいポケット、緑が三番目に大きいポケットです。
Showにチェックを入れて、右下のUpdateを押します。
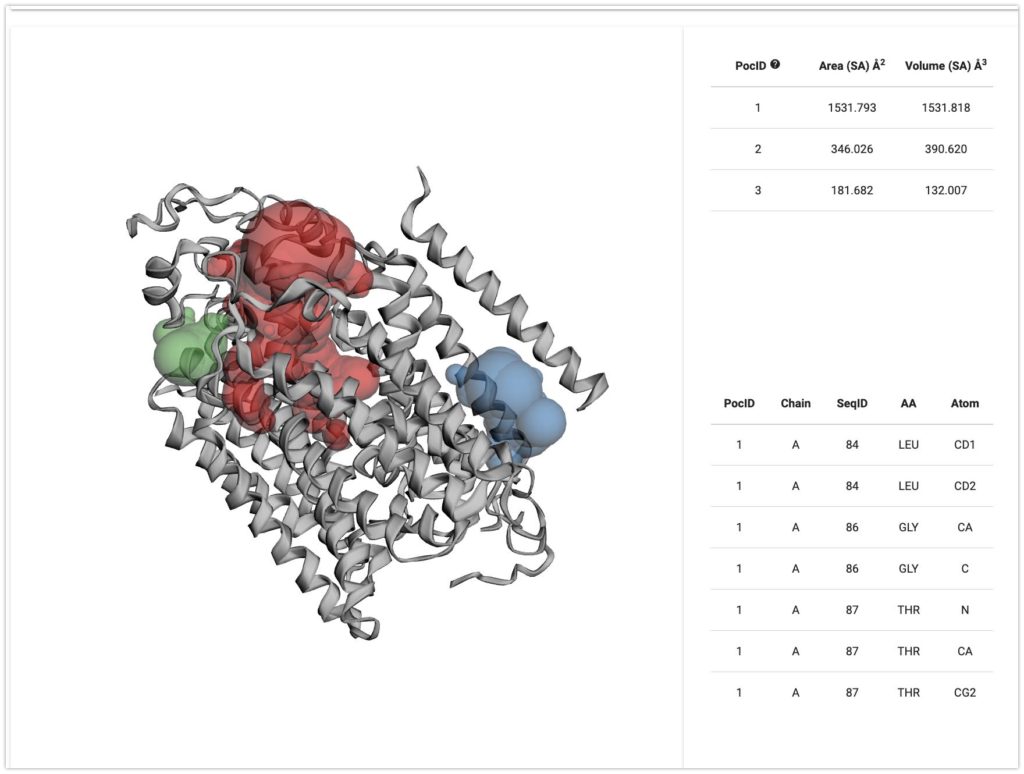
またDownloadを押すと、PymolやChimeraで開けるファイルがダウンロードできます。
ポケットの可視化
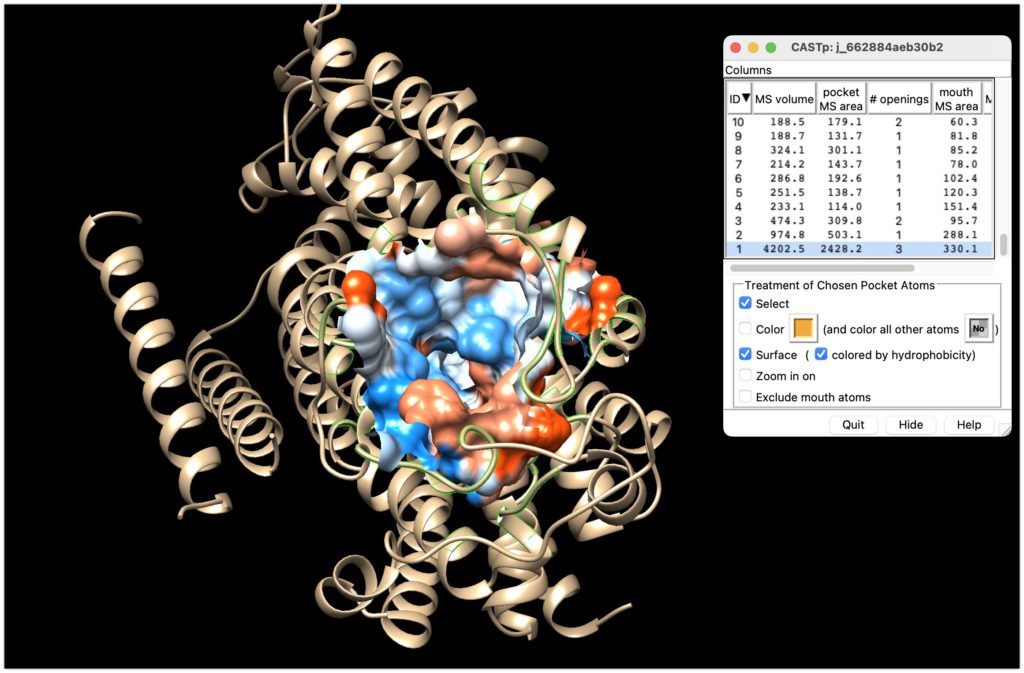
UCSF Chimeraで開いてみます。
試しにポケットを一番大きいポケットを見てみると以下のようになりました。
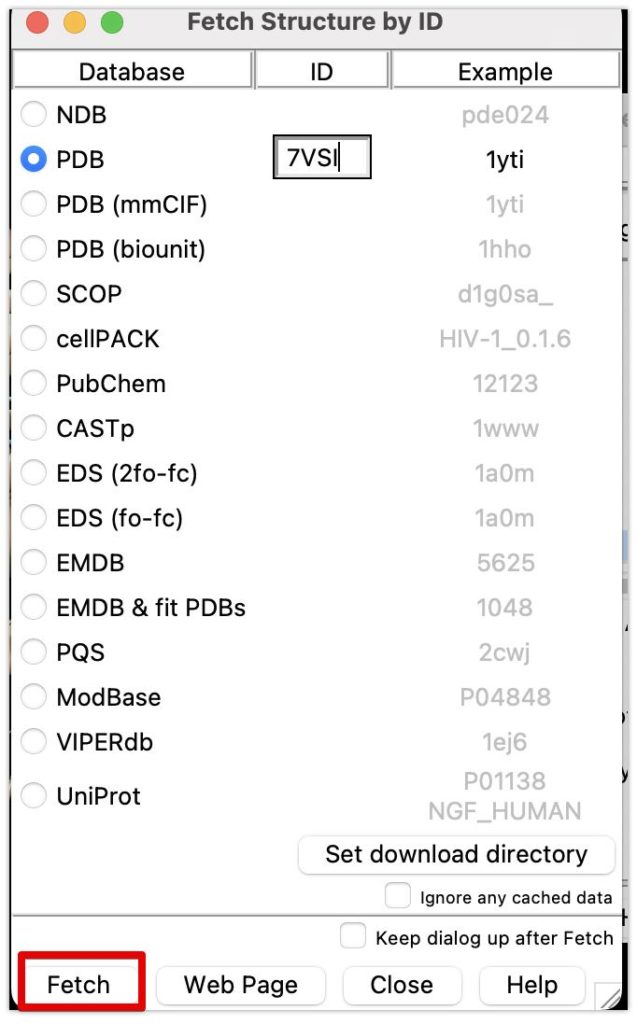
さらに左上のFile→Fetch by ID を押すと、PDB IDからタンパク質を表示することができます。リガンド付きの元のファイル(PDB:7VSI)を記入し、Fetch を押してみましょう。
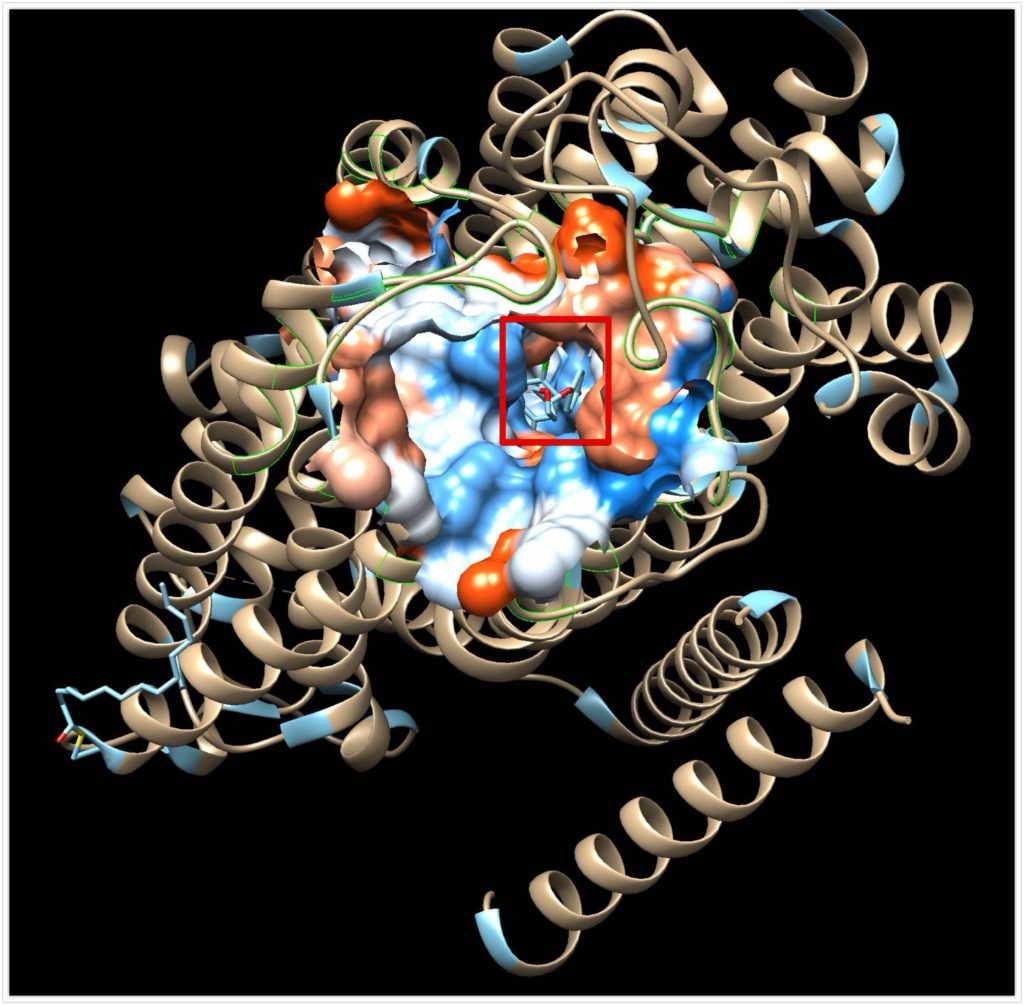
少し見えにくいですが、元々のリガンドがポケットの奥の方にあるのがわかります。
最後に
このポケット探索はPyRxやAuto DockによるスクリーニングのGrid Boxを決める際に役に立ちます。Grid Boxを予め決めることによって、目的の場所に結合する低分子をスクリーニングできます。ぜひこれらもCastpを使って、目的のポケットに結合する低分子を探してみてください!
参考文献
CASTp 3.0: computed atlas of surface topography of proteins
自宅でできるin silico創薬の技術書を販売中
¥3,200 → ¥1500 今なら50%OFF!!
新薬探索を試したい方必読!
ITエンジニアである著者の視点から、wetな研究者からもdryの創薬研究をわかりやすく身近に感じられるように解説しています
自宅でできるin silico創薬の技術書を販売中
¥3,200 → ¥1,800 今なら45%OFF!!
分子ドッキングやMDシミュレーションなど、
自宅でできるin silico創薬の解析方法を解説したものになります!





