本記事はAI創薬の一つである機械学習を用いたin silico screeningについて書かれた記事です。第5章まであり、すべての内容が理解できると、目的の標的にあった薬物候補化合物を発見することができます。こちらの記事は第2章で、スクレイピングによる公共データベース(PDB)からの機械学習の学習データを収集となります。webからの情報収集であるスクレイピングの基礎ができるようになります。ぜひ、トライしてみてください!第1章はこちら。
macOS Ventura(13.2.1), Google colaboratory
今回は直ぐにpythonが実行できるようにGoogle colabを利用していきます。
自宅でできるin silico創薬の技術書を販売中
¥3,200 → ¥1500 今なら50%OFF!!
新薬探索を試したい方必読!
ITエンジニアである著者の視点から、wetな研究者からもdryの創薬研究をわかりやすく身近に感じられるように解説しています
自宅でできるin silico創薬の技術書を販売中
¥3,200 → ¥1,500 今なら50%OFF!!
分子ドッキングやMDシミュレーションなど、
自宅でできるin silico創薬の解析方法を解説したものになります!
AI創薬とは?
AI創薬は、人工知能(AI)技術を利用して新しい薬物を発見、開発するプロセスです。AIは大量のデータを高速に処理し、薬物の候補を予測したり、薬物相互作用を評価したりします。また、AIは薬物の効果や安全性をシミュレートすることも可能で、臨床試験の前の段階でリスクを評価することができます。これにより、薬物開発のコストと時間を大幅に削減することが期待されています。AI創薬は、薬物開発の新しいパラダイムとして注目を集め、製薬企業や研究機関で積極的に研究、導入が進められています。また、バイオインフォマティクス、ケモインフォマティクス、機械学習、ディープラーニングなどの技術が組み合わされ、薬物開発のプロセスを革新しています。さらに、AI創薬は個人化医療の推進にも寄与し、患者にとって最適な治療法を提供する可能性を秘めています。
今回はAI創薬の中でも、in silico screeeningに焦点を当てて解説してきます。
以下の論文、記事をフォローしていきます。
NCBI – WWW Error Blocked Diagnostic
論文と公共データベースを使って無料で始めるAI創薬 – Qiita
この論文では新型コロナウイルスの標的タンパク質である3C-like プロテアーゼ(メインプロテアーゼ:Mpro)に対して、公共データベースにある化合物でスクリーニングを行い、最終的に41個のの化合物を薬物候補化合物としています。
AI創薬によるIn silico screeningの流れ
以下のような流れで行っていきます。全5章となります。
- 公共データベース(ChEMBL)からの機械学習の学習データを収集
- スクレイピングによる公共データベース(PDB)からの機械学習の学習データを収集(本記事)
- 機械学習の学習データの整形
- 整形データを用いた予測モデルの作成
- 予測モデルによる候補化合物のin silico screening
公共データベース(PDB)からのデータを収集
今回作成する機械学習モデルの学習に第1章で集めたデータに加え、PDBからのデータも加えます。そのため、PDBのデータをwebスクレイピングで収集します。
PDB(Protein Data Bank)は、生体高分子、特にタンパク質や核酸の三次元構造データを収集・提供する国際的なデータベースです。X線結晶構造解析、核磁気共鳴(NMR)、電子顕微鏡などの手法によって解析された構造データが含まれています。このデータベースは、生命科学や医薬品開発の分野で基本的なリソースとして利用されており、タンパク質の機能や相互作用の理解、新しい薬剤の設計に不可欠です。PDBは研究者によって自由にアクセスでき、データのダウンロードや可視化ツールも提供されています。また、各構造データは、詳細な実験手法や解析結果を含むメタデータと共に管理され、科学研究の透明性と再現性を高めています。
スクレイピング(Webスクレイピング)は、ウェブサイトからデータを抽出するプロセスです。これは、特定の情報を自動的に収集し、整理するための方法として広く用いられています。スクレイピングは、通常、専用のソフトウェアやスクリプトを使用して行われ、ウェブページのHTMLやXMLコードを解析して、必要なデータを取り出します。
この技術は、大量のデータを手動で収集するのが非現実的な場合に特に有用です。例えば、市場調査、価格比較、天気予報のデータ収集、ソーシャルメディア分析、不動産リストの収集など、多岐にわたる用途で利用されています。
しかし、スクレイピングは法的および倫理的な問題を引き起こす可能性もあります。ウェブサイトの利用規約違反になることがあり、プライバシーや著作権の問題を引き起こすこともあります。そのため、スクレイピングを行う際には、対象のウェブサイトのポリシーや法的制約を十分に理解し、遵守することが重要です。他のサイトにスクレイピングを使用する際は十分に注意して下さい。
Webデータ構造の確認
まずスクレイピングするために、データ構造の確認(どこにどのデータがあるかを確認していきます。
PDBから生物種が「コロナウイルス」阻害データKiとIC50が存在するデータを検索します。
こちらにアクセスしてみて下さい。
以下は検索画面です。
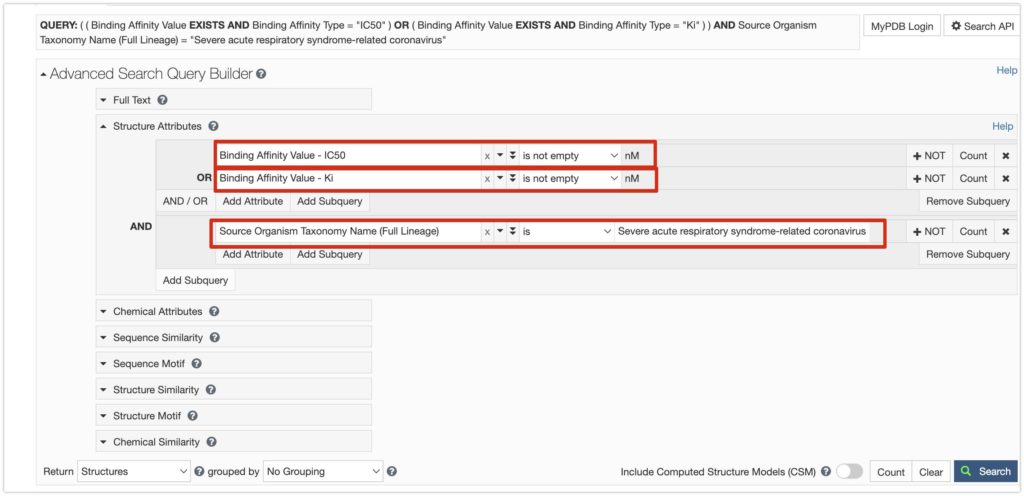
Structure Attributeに
Binding Affinity Value -IC50, is not empty
OR
Binding Affinity Value -Ki, is not empty
AND
Source Organism Taxonomy Name (Full Lineage), is, Severe acute respiratory syndrome-related coronavirus
を入力し、検索を押すと、
22件の結果が出力されます。
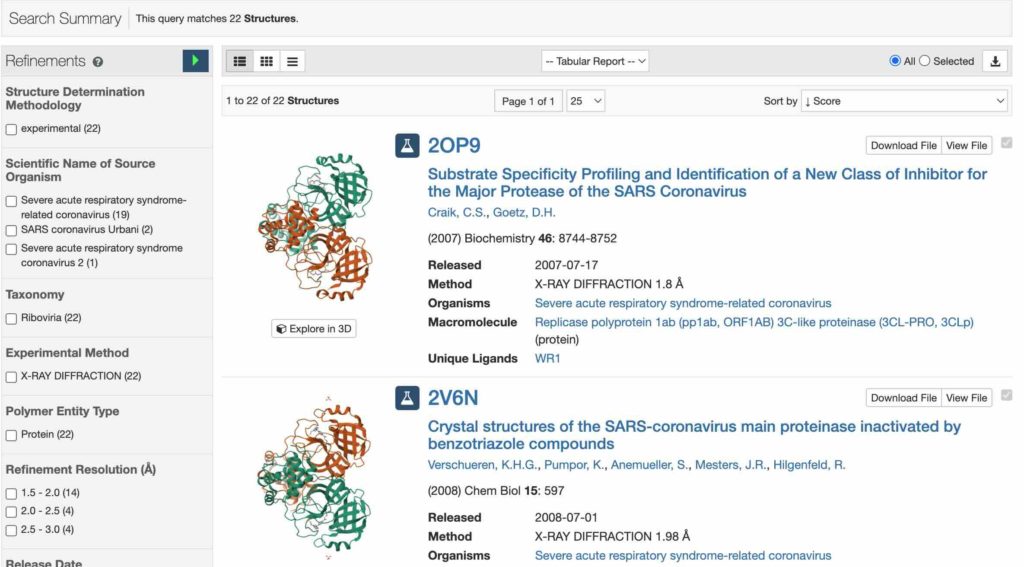
ここでスクレイピングの動作を確認するために、2OP9を例に取得するものを確認していきます。
2OP9をクリックします。
URLはhttps://www.rcsb.org/structure/2OP9 です。https://www.rcsb.org/structure/ + ligandIDでページが作られています。
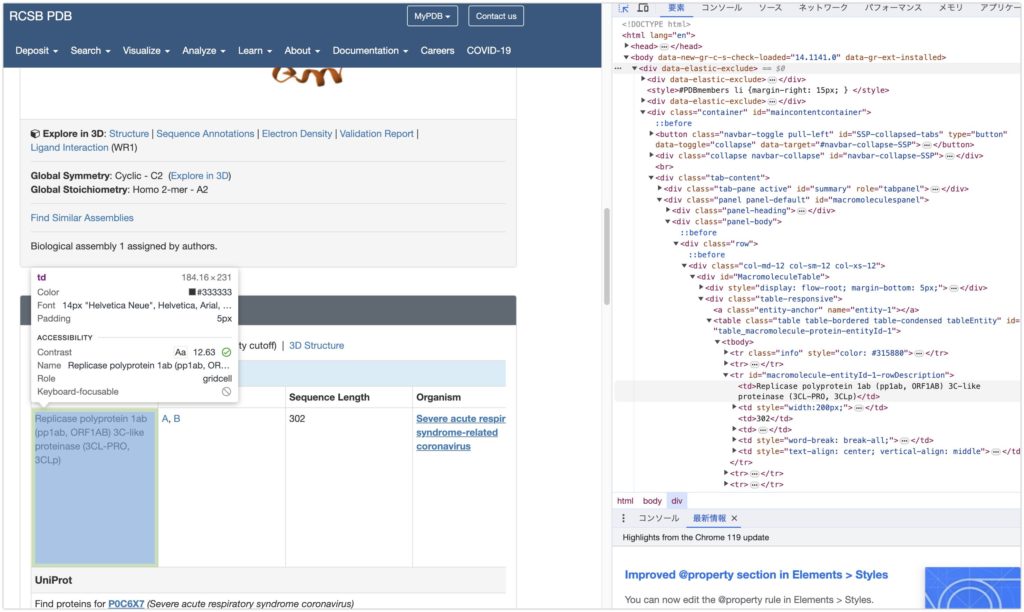
欲しい情報はMacromoleculesの情報です。標的のタンパク質名が書いてあります。
右クリック→検証を押すと、開発者ツールでHTML表示が出てきます。この領域を確認すると id=”macromolecule-entityId-1-rowDescription” のtrタブにあり、さらにその子要素tdとして表示されていることがわかります
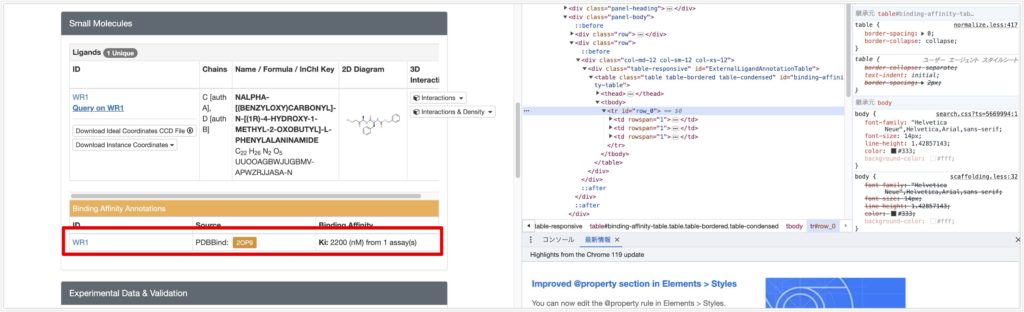
欲しい情報は中央部のBinding Affinity Annotationsの情報です。
右クリック→検証を押すと、開発者ツールでHTML表示が出てきます。この領域を確認すると id=”row_0” のtrタブにあり、さらにその子要素tdとして表示されていることがわかります
さらにこの2OP9のタンパク質のリガンドID WR1をクリックします。リガンドのURLはhttps://www.rcsb.org/ligand/WR1 であり、https://www.rcsb.org/ligand/ + ligand_IDでページが表示されています。
欲しい情報はChemical Component SummaryのsmileとinChIとInChiKeyです。
同様にHTML表示を行うと、例えば、smileの欲しい情報はid='chemicalIsomeric’ のtrタブの中のtdタブにあることがわかります。他も同様に各idで指定されたtrタブのtdタブにあるかと思います。
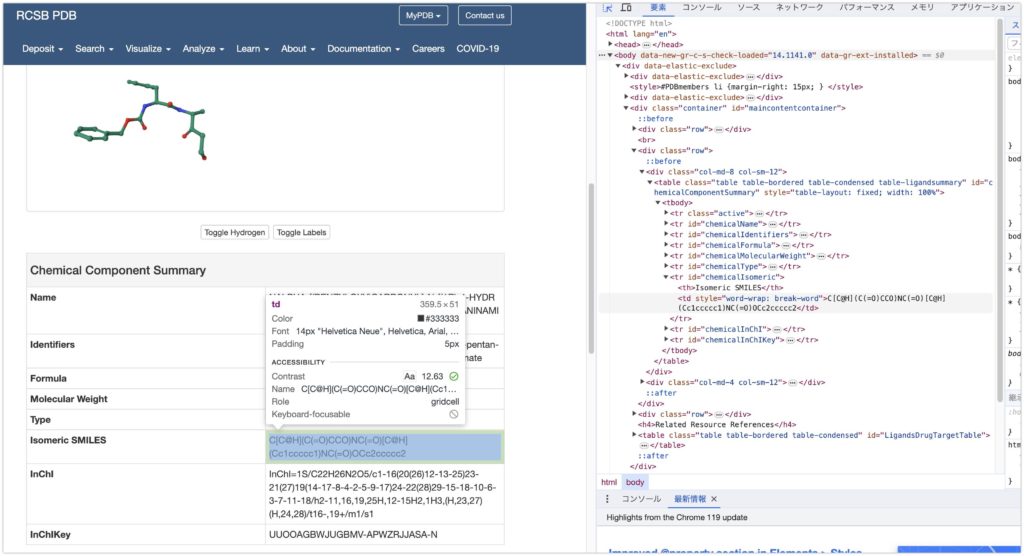
ということで、欲しい情報の確認とそのHTMLがそうなっているかについて確認しました。
それでは実際に以下のコードでスクレイピングを行っていきます。**@kimisyo(晶 公畑)**の記事の方法ではなぜかできなかったので、ここはオリジナルです。
リガンド情報のスクレイピング
以下をGoogle colabで最初から実行してみて下さい。
必要ライブラリとツールのインストール
まずスクレイピングに必要なライブラリとツールをインストールをします。
# 必要なライブラリとツールのインストール
!pip install selenium
!apt-get update # ChromeDriverをインストールする前に更新
!apt install chromium-chromedriver
!pip install pandas
from selenium import webdriver
from selenium.webdriver.common.by import By
chrome_options = webdriver.ChromeOptions()
chrome_options.add_argument('--headless')
chrome_options.add_argument('--no-sandbox')
chrome_options.add_argument('--disable-dev-shm-usage')
driver = webdriver.Chrome(options=chrome_options)
driver.implicitly_wait(10)
from bs4 import BeautifulSoup
import requests
soup = BeautifulSoup()
import re
import pandas as pd
このコードは、Pythonでウェブスクレイピングを行うためのセットアップと初期化の手順を含んでいます。それぞれの部分について説明します(よくわからなければ飛ばしても大丈夫です):
必要なライブラリとツールのインストール
- Seleniumのインストール:
!pip install selenium: Seleniumは、ウェブブラウザを自動操作するためのライブラリです。このコマンドはPython環境にSeleniumをインストールします。
- システム更新とChromeDriverのインストール:
!apt-get update: これはLinuxシステムのパッケージリストを更新します。!apt install chromium-chromedriver: ChromeDriverは、SeleniumでGoogle Chromeブラウザを操作するためのドライバーです。このコマンドはシステムにChromeDriverをインストールします。
- Pandasのインストール:
!pip install pandas: Pandasは、データ分析と操作のための強力なライブラリです。このコマンドはPandasをインストールします。
Seleniumの設定と初期化
from selenium import webdriver: Seleniumのwebdriverモジュールをインポートします。これはブラウザを自動操作するために使用されます。from selenium.webdriver.common.by import By: **By**クラスをインポートします。これは、要素を特定するためのさまざまな方法を提供します。chrome_options = webdriver.ChromeOptions(): Chromeのオプションを設定するためのオブジェクトを作成します。- Chromeオプションの設定:
-headless: ブラウザをヘッドレスモードで起動します。つまり、GUIなしで動作します。-no-sandbox: セキュリティサンドボックスを無効化します。Dockerなどの特定の環境ではこれが必要です。-disable-dev-shm-usage: **/dev/shm**の使用を無効化します。これは一時的なメモリ領域の問題を回避するためです。
driver = webdriver.Chrome(options=chrome_options): ChromeDriverのインスタンスを作成し、設定したオプションを適用します。driver.implicitly_wait(10): 要素が見つかるまでの最大待機時間を10秒に設定します。
その他のライブラリ
from bs4 import BeautifulSoup: BeautifulSoupライブラリをインポートします。これはHTMLやXMLファイルを解析するために使われます。import requests: Requestsライブラリをインポートします。これはHTTPリクエストを簡単に送るために使われます。soup = BeautifulSoup(): BeautifulSoupオブジェクトを初期化します。import re: Pythonの標準ライブラリである正規表現(re)モジュールをインポートします。import pandas as pd: Pandasライブラリをpdという名前でインポートします。
このコードは、ウェブページのデータを抽出し、解析するための基本的なセットアップを行っています。Seleniumを使用してウェブページを取得し、BeautifulSoupやPandasを利用してデータを解析および操作することができます。
検索結果のタンパク質IDの取得
続いて、以下のコードを実行してください。これを実行すると、検索結果のprotein ID(プログラム上ではProtein_code)が取得できます。search_urlには検索を実行したURLを入れています。
def get_protein_list(search_url):
# 検索結果のurlを取得する
driver.get(search_url)
proteins_list = driver.find_elements(By.CLASS_NAME, 'results-item-header')
protein_code_lists = []
# proteinID(Protein_code)の取得
for index, protein in enumerate(proteins_list):
lines = protein.text.split('\\n')
protein_code = lines[0]
protein_code_lists.append(protein_code)
return (protein_code_lists)
search_url ='<https://www.rcsb.org/search?request=%7B%22query%22%3A%7B%22type%22%3A%22group%22%2C%22logical_operator%22%3A%22and%22%2C%22nodes%22%3A%5B%7B%22type%22%3A%22group%22%2C%22logical_operator%22%3A%22and%22%2C%22nodes%22%3A%5B%7B%22type%22%3A%22group%22%2C%22nodes%22%3A%5B%7B%22type%22%3A%22group%22%2C%22logical_operator%22%3A%22and%22%2C%22nodes%22%3A%5B%7B%22type%22%3A%22terminal%22%2C%22service%22%3A%22text%22%2C%22parameters%22%3A%7B%22attribute%22%3A%22rcsb_binding_affinity.value%22%2C%22operator%22%3A%22exists%22%2C%22negation%22%3Afalse%7D%7D%2C%7B%22type%22%3A%22terminal%22%2C%22service%22%3A%22text%22%2C%22parameters%22%3A%7B%22attribute%22%3A%22rcsb_binding_affinity.type%22%2C%22operator%22%3A%22exact_match%22%2C%22value%22%3A%22Ki%22%2C%22negation%22%3Afalse%7D%7D%5D%2C%22label%22%3A%22nested-attribute%22%7D%2C%7B%22type%22%3A%22group%22%2C%22logical_operator%22%3A%22and%22%2C%22nodes%22%3A%5B%7B%22type%22%3A%22terminal%22%2C%22service%22%3A%22text%22%2C%22parameters%22%3A%7B%22attribute%22%3A%22rcsb_binding_affinity.value%22%2C%22operator%22%3A%22exists%22%2C%22negation%22%3Afalse%7D%7D%2C%7B%22type%22%3A%22terminal%22%2C%22service%22%3A%22text%22%2C%22parameters%22%3A%7B%22attribute%22%3A%22rcsb_binding_affinity.type%22%2C%22operator%22%3A%22exact_match%22%2C%22value%22%3A%22IC50%22%2C%22negation%22%3Afalse%7D%7D%5D%2C%22label%22%3A%22nested-attribute%22%7D%5D%2C%22logical_operator%22%3A%22or%22%7D%2C%7B%22type%22%3A%22group%22%2C%22nodes%22%3A%5B%7B%22type%22%3A%22terminal%22%2C%22service%22%3A%22text%22%2C%22parameters%22%3A%7B%22attribute%22%3A%22rcsb_entity_source_organism.taxonomy_lineage.name%22%2C%22operator%22%3A%22exact_match%22%2C%22negation%22%3Afalse%2C%22value%22%3A%22Severe%20acute%20respiratory%20syndrome-related%20coronavirus%22%7D%7D%5D%2C%22logical_operator%22%3A%22and%22%7D%5D%2C%22label%22%3A%22text%22%7D%5D%7D%2C%22return_type%22%3A%22entry%22%2C%22request_options%22%3A%7B%22paginate%22%3A%7B%22start%22%3A0%2C%22rows%22%3A25%7D%2C%22results_content_type%22%3A%5B%22experimental%22%5D%2C%22sort%22%3A%5B%7B%22sort_by%22%3A%22score%22%2C%22direction%22%3A%22desc%22%7D%5D%2C%22scoring_strategy%22%3A%22combined%22%7D%2C%22request_info%22%3A%7B%22query_id%22%3A%2291426ae5cdeb051497a51a659ca9886f%22%7D%7D>'
protein_lists = get_protein_list(search_url)
protein_lists
このコードは、特定の検索条件に基づいてRCSB Protein Data Bank(PDB)ウェブサイトからタンパク質のリストを取得するためのPython関数 get_protein_list を定義しています。それぞれの部分について説明します:
関数 get_protein_list
- 引数:
search_url– RCSB PDBの検索結果ページのURLです。 - 動作:
driver.get(search_url): SeleniumのWebDriverを使用して、指定されたURL(search_url)のページを開きます。proteins_list = driver.find_elements(By.CLASS_NAME, 'results-item-header'): 指定されたクラス名('results-item-header')を持つ要素(この場合はタンパク質のリスト項目)をページから全て見つけ出します。protein_code_lists = []: タンパク質のコードを格納するための空のリストを作成します。- **
for**ループを使用して、各タンパク質項目に対して以下の処理を行います:lines = protein.text.split('\\n'): タンパク質項目のテキストを改行で分割してリストに変換します。protein_code = lines[0]: リストの最初の要素(タンパク質コード)を取得します。protein_code_lists.append(protein_code): 取得したタンパク質コードをリストprotein_code_listsに追加します。
- 戻り値:
return (protein_code_lists): 取得したタンパク質コードのリストを返します。
使用例
- 検索URLの設定:
search_urlには、RCSB PDBで特定の検索条件(例:特定の結合親和性、特定の生物種由来のタンパク質など)を使用して生成された検索結果ページのURLが設定されます。 - 関数の呼び出し:
protein_lists = get_protein_list(search_url)で関数を呼び出し、結果をprotein_listsに格納します。
使用上の注意
このコードは、RCSB PDBのウェブサイト構造やクラス名が変更されないことを前提としています。ウェブサイトが更新されると、コードを修正する必要があります。
以下のように出力されて、検索結果のProtein IDがあると思います。
['2OP9',
'2V6N',
'2Z94',
'2Z9G',
'2ZU4',
'2ZU5',
'3SN8',
'2ALV',
'2GZ7',
'2GZ8',
'2QIQ',
'2VJ1',
'2XYR',
'3E9S',
'3MJ5',
'3V3M',
'4MDS',
'4OVZ',
'4OW0',
'4TWW',
'4WY3',
'6W63']
阻害活性の取得
続いて、以下のコードを実行してください。
def get_ligand(protein_id):
protein_url = "<https://www.rcsb.org/structure/>" + protein_id
response = requests.get(protein_url)
if response.status_code != 200:
response.status_code, []
soup = BeautifulSoup(response.text, 'html.parser')
# リガンド情報を取得する。
binding_affinity_annotations_tables = soup.find_all('tr', id='row_0')
for tr in binding_affinity_annotations_tables:
binding_affinity_annotations_texts = [td.get_text(strip=True) for td in tr.find_all('td')]
ligand_id = binding_affinity_annotations_texts[0]
inhibitory_activity = binding_affinity_annotations_texts[2]
# 阻害がKiで表されている場合
ki_match = re.search(r'Ki', inhibitory_activity)
if ki_match:
ki = ki_match.group()
inhibitory_activity_value = int(float(re.search(r'\\d+', inhibitory_activity).group()))
else:
# 阻害がIC50で表されている場合
ki = "IC50"
inhibitory_activity_values = re.findall(r'(\\d+\\.?\\d*e[+\\-]?\\d+|\\d+)', inhibitory_activity)
int_values = [int(float(value)) for value in inhibitory_activity_values]
if len(inhibitory_activity_values) >= 4:
Inhibitory_min = int_values[1]
Inhibitory_max = int_values[2]
Inhibitory_number = 2
inhibitory_activity_value = (Inhibitory_min+Inhibitory_max)/Inhibitory_number
else:
inhibitory_activity_value = int_values[1]
unit = re.search(r'nM', inhibitory_activity).group()
# 対象のタンパク質を取得する。
macromolecule_tables = soup.find_all('tr', id='macromolecule-entityId-1-rowDescription')
for tr in macromolecule_tables:
macromolecule_tables_texts = [td.get_text(strip=True) for td in tr.find_all('td')]
macromolecule = macromolecule_tables_texts[0]
return(ligand_id, ki, inhibitory_activity_value, unit, macromolecule)
data_list =[]
for protein in protein_lists:
result = get_ligand(protein)
data_list.append(result)
# 各列を格納するための辞書を初期化
ligand_data = {
'ligand_id': [],
'ki/IC50': [],
'inhibitory_activity_value': [],
'unit': [],
'macromolecule': []
}
# データリストから各要素を抽出し、対応する列に追加
for record in data_list:
ligand_data['ligand_id'].append(record[0])
ligand_data['ki/IC50'].append(record[1])
ligand_data['inhibitory_activity_value'].append(record[2])
ligand_data['unit'].append(record[3])
ligand_data['macromolecule'].append(record[4])
print(ligand_data)
このコードは、特定のタンパク質IDに対応するリガンド(小分子)のデータをRCSB Protein Data Bank(PDB)ウェブサイトから取得し、それらのデータを整理して表示するためのPythonスクリプトです。具体的には、次の手順で動作します:
関数 get_ligand
- 引数:
protein_id– タンパク質のID。 - 動作:
requests.get(protein_url): 指定されたタンパク質IDに基づいてPDBのURLにアクセスし、HTMLページを取得します。- ステータスコードのチェック: ページが正常に取得できたかを確認します。
BeautifulSoup(response.text, 'html.parser'): 取得したHTMLページを解析します。soup.find_all('tr', id='row_0'): HTML内で特定のID(row_0)を持つテーブル行(**<tr>**タグ)をすべて見つけます。- リガンドID、阻害活性、阻害活性の単位、および対象のタンパク質を解析して取得します。IC50に関しては最大値と最小値があるものに関してはその平均をとっています。
リガンドデータの取得と整理
for protein in protein_lists: **protein_listsに含まれる各タンパク質IDに対してget_ligand関数を呼び出し、得られたリガンドデータをdata_list**に追加します。
データの整理と表示
- **
ligand_data辞書を初期化し、data_list**から得られたリガンドデータを各列に格納します。 - 最後に、**
ligand_data**を印刷し、取得したデータを表示します。
結果は以下のように、各タンパク質に対応するリガンドの阻害活性と検索結果で取得したID毎に辞書型として、リガンドID、阻害活性、阻害活性の単位、および対象のタンパク質が取得できます。
Ki: 2200 (nM) from 1 assay(s)
Ki: 1380 (nM) from 1 assay(s)
Ki: 1400 (nM) from 1 assay(s)
Ki: 700 (nM) from 1 assay(s)
Ki: 38 (nM) from 1 assay(s)
Ki: 99 (nM) from 1 assay(s)
Ki: 2240 (nM) from 1 assay(s)
IC50: min: 7.00e+4, max: 8.00e+5 (nM) from 2 assay(s)
IC50: 300 (nM) from 1 assay(s)
IC50: 3000 (nM) from 1 assay(s)
IC50: 8.00e+4 (nM) from 1 assay(s)
IC50: 5000 (nM) from 1 assay(s)
IC50: 740 (nM) from 1 assay(s)
IC50: min: 230, max: 2640 (nM) from 3 assay(s)
IC50: min: 320, max: 1200 (nM) from 3 assay(s)
IC50: 4800 (nM) from 1 assay(s)
IC50: 6200 (nM) from 1 assay(s)
IC50: 490 (nM) from 1 assay(s)
IC50: 150 (nM) from 1 assay(s)
IC50: 6.30e+4 (nM) from 1 assay(s)
IC50: 2.40e+5 (nM) from 1 assay(s)
IC50: 2300 (nM) from 1 assay(s)
{'ligand_id': ['WR1', 'XP1', 'TLD', 'PMA', 'ZU3', 'ZU5', 'S89', 'CY6', 'D3F', 'F3F', 'CYV', 'XP1', 'SFG', 'TTT', 'GRM', '0EN', '23H', 'P85', 'S88', '3A7', '3X5', 'X77'], 'ki/IC50': ['Ki', 'Ki', 'Ki', 'Ki', 'Ki', 'Ki', 'Ki', 'IC50', 'IC50', 'IC50', 'IC50', 'IC50', 'IC50', 'IC50', 'IC50', 'IC50', 'IC50', 'IC50', 'IC50', 'IC50', 'IC50', 'IC50'], 'inhibitory_activity_value': [2200, 1380, 1400, 700, 38, 99, 2240, 435000.0, 300, 3000, 80000, 5000, 740, 1435.0, 760.0, 4800, 6200, 490, 150, 63000, 240000, 2300], 'unit': ['nM', 'nM', 'nM', 'nM', 'nM', 'nM', 'nM', 'nM', 'nM', 'nM', 'nM', 'nM', 'nM', 'nM', 'nM', 'nM', 'nM', 'nM', 'nM', 'nM', 'nM', 'nM'], 'macromolecule': ['Replicase polyprotein 1ab (pp1ab, ORF1AB) 3C-like proteinase (3CL-PRO, 3CLp)', 'REPLICASE POLYPROTEIN 1AB', 'Replicase polyprotein 1ab', '3C-like proteinase', '3C-like proteinase', '3C-like proteinase', '3C-like proteinase', 'Replicase polyprotein 1ab', 'Replicase polyprotein 1ab', 'Replicase polyprotein 1ab', 'Replicase polyprotein 1ab', 'SARS CORONAVIRUS MAIN PROTEINASE', "PUTATIVE 2'-O-METHYL TRANSFERASE", 'Non-structural protein 3', 'Replicase polyprotein 1a', '3C-like proteinase', '3C-like proteinase', 'Papain-like proteinase', 'papain-like protease', '3C-like proteinase', '3C-like proteinase', '3C-like proteinase']}
リガンドの特徴の取得
続いて以下のコードを実行してください。
def get_ligand(ligand_id):
tmp_url = "<https://www.rcsb.org/ligand/>" + ligand_id
response = requests.get(tmp_url)
if response.status_code != 200:
response.status_code, []
# smile、inChI, inChikeyテーブルの取得
soup = BeautifulSoup(response.text, 'html.parser')
smile_tables = soup.find_all('tr', id='chemicalIsomeric')
smile = smile_tables[0].find('td').string.strip()
InChI_tables = soup.find_all('tr', id='chemicalInChI')
inChI = InChI_tables[0].find('td').string.strip()
InChIKey_tables = soup.find_all('tr', id='chemicalInChIKey')
inChIKey = InChIKey_tables[0].find('td').string.strip()
return [smile, inChI, inChIKey]
ligand_properties_list = []
for ligand_id in ligand_data['ligand_id']:
result = get_ligand(ligand_id)
ligand_properties_list.append(result)
ligand_properties_data = {
'smile': [],
'inChI': [],
'inChIKey': []
}
for record in ligand_properties_list:
ligand_properties_data['smile'].append(record[0])
ligand_properties_data['inChI'].append(record[1])
ligand_properties_data['inChIKey'].append(record[2])
print(ligand_properties_data)
このコードは、特定のリガンドIDに対応する化学情報をRCSB Protein Data Bank(PDB)ウェブサイトから取得し、それらのデータを整理して表示するためのPythonスクリプトです。コードは以下のように構成されています:
関数 get_ligand
- 引数:
ligand_id– リガンドのID。 - 動作:
requests.get(tmp_url): 指定されたリガンドIDに基づいてPDBのリガンドページのURLにアクセスし、HTMLページを取得します。- ステータスコードのチェック: ページが正常に取得できたかを確認します。
BeautifulSoup(response.text, 'html.parser'): 取得したHTMLページを解析します。soup.find_all('tr', id='...'): HTML内で特定のIDを持つテーブル行(**<tr>**タグ)を見つけ、SMILES表記、InChI、およびInChIKeyの情報を取得します。
- 戻り値:
[smile, inChI, inChIKey]– 取得したSMILES表記、InChI、InChIKeyの情報のリスト。
リガンドプロパティの取得と整理
for ligand_id in ligand_data['ligand_id']: **ligand_data辞書に格納されているリガンドIDのリストに対してget_ligand関数を呼び出し、得られたリガンドプロパティデータをligand_properties_list**に追加します。
データの整理と表示
- **
ligand_properties_data辞書を初期化し、ligand_properties_list**から得られたリガンドプロパティデータを各列に格納します。 - 最後に、**
ligand_properties_data**を印刷し、取得したデータを表示します。
データの統合、エクセルファイルへの抽出
これまで出てきたリガンド情報を以下のコードでまとめ、エクセルファイルに抽出します。
ligand_merged_data = ligand_data | ligand_properties_data
ligand_merged_data
df = pd.DataFrame(ligand_merged_data)
df.to_excel('output.xlsx', index=False)
このコードの目的は、二つの辞書 ligand_data と ligand_properties_data を結合し、その結果をPandasのDataFrameに変換してExcelファイルとして出力することです。コードは以下の手順で構成されています:
辞書の結合
ligand_merged_data = ligand_data | ligand_properties_data: ここで、Pythonの辞書結合演算子|を使用して、ligand_dataとligand_properties_dataの二つの辞書を結合します。これにより、両辞書のキーと値のペアが結合された新しい辞書ligand_merged_dataが作成されます。
データフレームの作成
df = pd.DataFrame(ligand_merged_data): 結合された辞書ligand_merged_dataをPandasのDataFrameに変換します。DataFrameは表形式のデータを扱うための強力なツールです。
Excelファイルへの出力
df.to_excel('output.xlsx', index=False): DataFramedfをExcelファイルoutput.xlsxとして出力します。index=Falseパラメータは、DataFrameのインデックスをExcelファイルに含めないように指定しています。
Google Colabのファイルに新しくoutput.xisxが出力されていると思います。
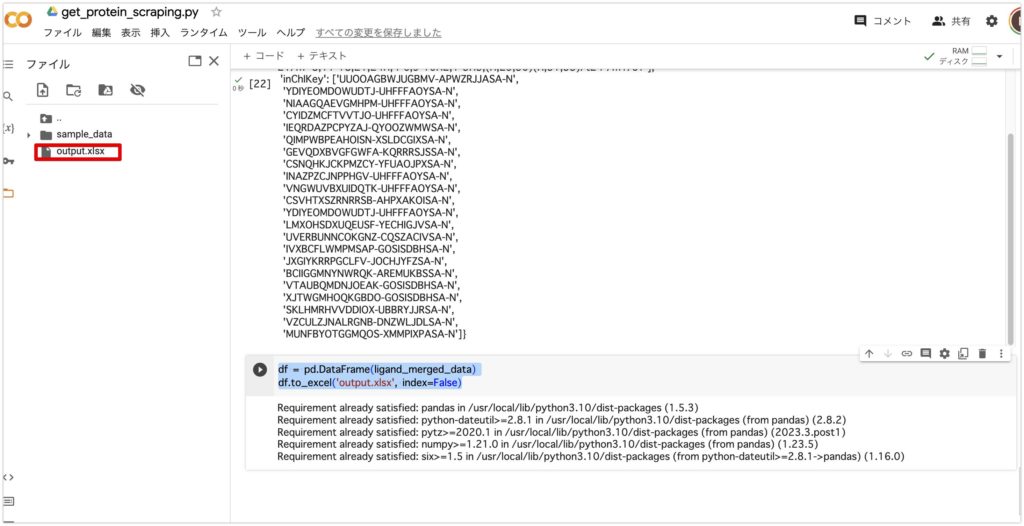
エクセルファイルを開くと以下のようになっていると思います。ligand_id、ki/IC50の値、標的タンパク質、smile、inChI、inChIKeyが取得できています。
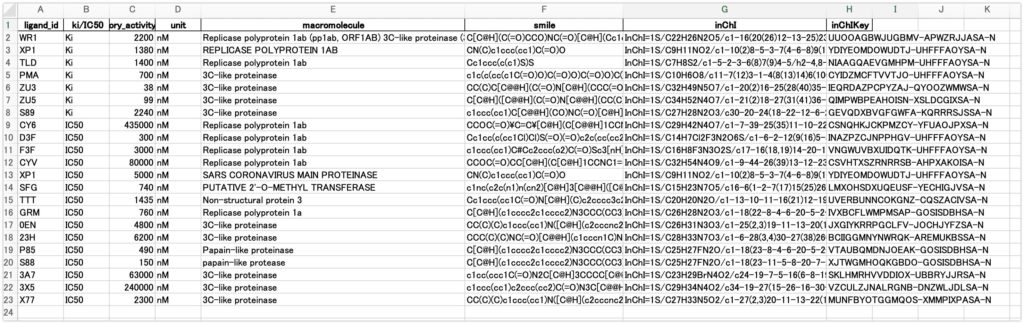
ここからmacromoleculeが3C-like proteaseとMain proteaseのみのデータに絞ります。22件しかないので、手作業で3C-like proteaseとMain protease以外は消して、新しく3C-like_and_main_proteases.xisxとして保存しておきます。
最後に
いかがでしたでしょうか?第1章と共にこの章ではデータベースを作るためにリガンドの情報をPDBからスクレイピングによって取得しました。スクレイピングは他のwebサイトでも使えるので、サイトの注意事項をよく読んで使ってみて下さい。第3章では得られたデータの整形を行なっていきます。
参考文献
スクレイピングをもっと基礎から学びたい方
【PythonでWebスクレイピング】Beautiful Soupの使い方解説! 〜 初心者向け 〜 プログラミング入門
【PythonでWebスクレイピング】Seleniumの使い方解説! Web操作自動化もできる!〜 初心者向け 〜 プログラミング入門
謝辞
この記事は以下の論文のフォローとなります。機械学習とAI創薬についてわかりやすく書かれています。このような論文を書いて下った筆者らに感謝を申し上げます。
NCBI – WWW Error Blocked Diagnostic
この記事を書くにあたり、**@kimisyo(晶 公畑)**さんの記事が大変参考になりました。ありがとうございます。





