本記事はAI創薬の一つである機械学習を用いたin silico screeningについて書かれた記事です。第5章まであり、すべての内容が理解できると、目的の標的にあった薬物候補化合物を発見することができます。こちらの記事は第1章で、公共データベース(ChEMBL)からのデータを収集となります。機械学習用のデータの取得ができるようになります。ぜひ、トライしてみてください!
macOS Ventura(13.2.1), Google colaboratory
今回は直ぐにpythonが実行できるようにGoogle colabを利用していきます。
自宅でできるin silico創薬の技術書を販売中
¥3,200 → ¥1500 今なら50%OFF!!
新薬探索を試したい方必読!
ITエンジニアである著者の視点から、wetな研究者からもdryの創薬研究をわかりやすく身近に感じられるように解説しています
自宅でできるin silico創薬の技術書を販売中
¥3,200 → ¥1,500 今なら50%OFF!!
分子ドッキングやMDシミュレーションなど、
自宅でできるin silico創薬の解析方法を解説したものになります!
AI創薬とは?
AI創薬は、人工知能(AI)技術を利用して新しい薬物を発見、開発するプロセスです。AIは大量のデータを高速に処理し、薬物の候補を予測したり、薬物相互作用を評価したりします。また、AIは薬物の効果や安全性をシミュレートすることも可能で、臨床試験の前の段階でリスクを評価することができます。これにより、薬物開発のコストと時間を大幅に削減することが期待されています。AI創薬は、薬物開発の新しいパラダイムとして注目を集め、製薬企業や研究機関で積極的に研究、導入が進められています。また、バイオインフォマティクス、ケモインフォマティクス、機械学習、ディープラーニングなどの技術が組み合わされ、薬物開発のプロセスを革新しています。さらに、AI創薬は個人化医療の推進にも寄与し、患者にとって最適な治療法を提供する可能性を秘めています。
今回はAI創薬の中でも、in silico screeeningに焦点を当てて解説してきます。
以下の論文、記事をフォローしていきます。
NCBI – WWW Error Blocked Diagnostic
論文と公共データベースを使って無料で始めるAI創薬 – Qiita
この論文では新型コロナウイルスの標的タンパク質である3C-like プロテアーゼ(メインプロテアーゼ:Mpro)に対して、公共データベースにある化合物でスクリーニングを行い、最終的に41個のの化合物を薬物候補化合物としています。
AI創薬によるIn silico screeningの流れ
以下のような流れで行っていきます。全5章となります。
- 公共データベース(ChEMBL)からの機械学習の学習データを収集(本記事)
- スクレイピングによる公共データベース(PDB)からの機械学習の学習データを収集
- 機械学習の学習データの整形
- 整形データを用いた予測モデルの作成
- 予測モデルによる候補化合物のin silico screening
公共データベース(ChEMBL)からの機械学習の学習データを収集
Postgre SQL(データベースの作成)
まずChEMBLから機械学習のモデル作成用のデータを入手します。
ChEMBLは、主に薬理学的に活性な生体分子やその標的となるタンパク質に関する情報を収集したデータベースです。このデータベースは、化学構造、生物活性データ、ターゲット情報、文献情報などを含んでおり、研究者や薬剤師にとって重要なリソースとなっています。特に、新しい薬剤の発見や、既存の薬剤の再利用に関心がある研究者にとっては貴重な情報源です。ChEMBLは、ヨーロッパバイオインフォマティクス研究所 (EMBL-EBI) によって運営されており、公開データとして無料でアクセスできます。データは定期的に更新され、薬剤化学、薬理学、生物学などの分野の最新の研究成果を反映しています。また、ユーザーフレンドリーなインターフェースを備え、検索やデータのダウンロードが容易に行える点も特徴です。
こちらにアクセスしてください。chembl_33_postgresql.tar.gzをダウンロードしてください。
そして、解凍をお願いします。
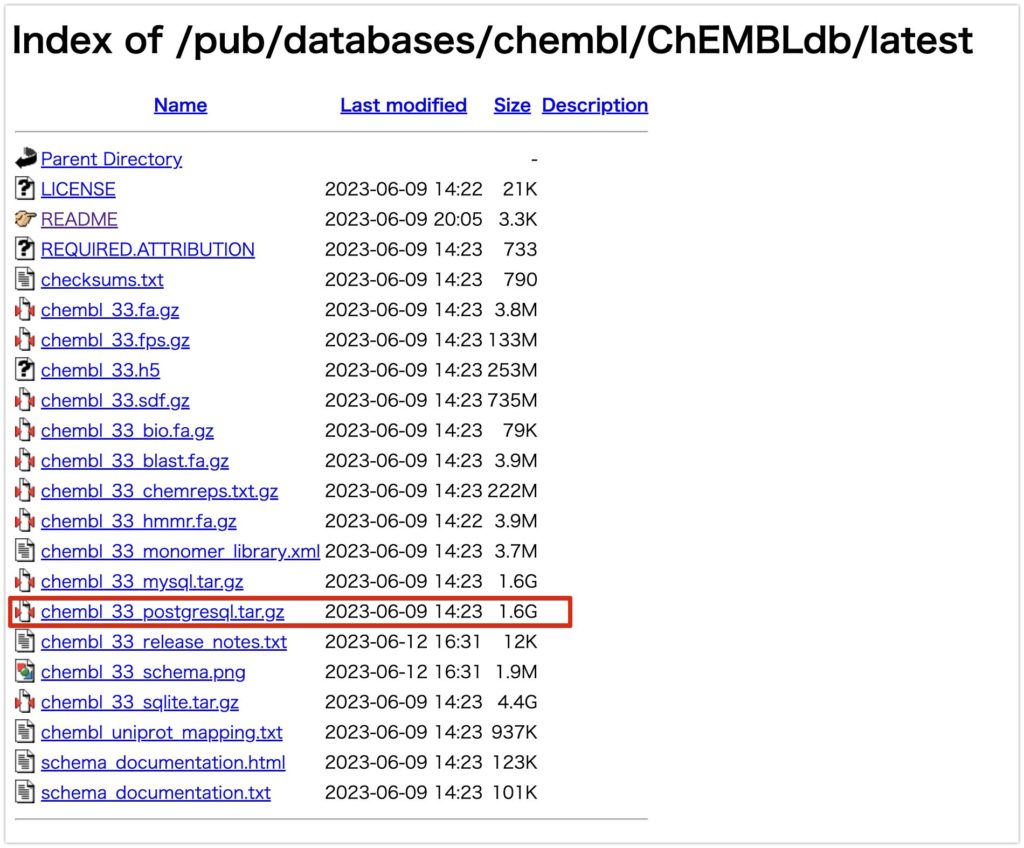
続いて、DBを作成していきます。
PostgreSQLはオープンソースの関係データベース管理システムで、SQLとProcedural Languageをサポート。企業向け機能如くトランザクションやインデックス、フルテキスト検索、JSONデータ格納が可能です。拡張可能なアーキテクチャでカスタム関数やデータ型を追加でき、安定性とデータ整合性に優れています。アクティブなコミュニティに支えられ、継続的にアップデートと改善が行われている。幅広い用途に対応し、個人のプロジェクトから大規模企業アプリケーションまで利用されています。公式ドキュメントでさらに学べます。
こちらの動画を参考にPostgre SQLをインストールします。
以下に簡単に概要を載せます。Mac用です。
まずHomebrewをインストールします。
Homebrewは、macOS向けのパッケージ管理システムです。パッケージ管理システムは、ソフトウェアのインストールやアップデートを簡単に行えるツールであり、開発者やユーザーにとって便利です。
まずターミナルで
/bin/bash -c "$(curl -fsSL <https://raw.githubusercontent.com/Homebrew/install/HEAD/install.sh>)"
と打ち、Homebrewをインストールします。
続いて以下のコードでpostgresqlをインストールします。
brew install postgresql
psql --version を実行し、psql (PostgreSQL) 14.9 (Homebrew) などと返ってくれば、インストールは完了でしています。
続いて、データを収納するデータディレクトリの作成と初期化を行います。以下のコードを実行して下さい。
sudo mkdir -p /usr/local/var/postgres
initdb /usr/local/var/postgres
sudo mkdir -p /usr/local/var/postgres:sudo: このプレフィックスは、スーパーユーザー(root)の権限でコマンドを実行するためのものです。多くのシステム操作、特にシステムの中心部に影響を及ぼすものは、通常のユーザー権限では実行できません。そのため、**sudo**を使用してこれらの操作を行います。mkdir: これは”make directory”の略で、新しいディレクトリを作成するためのコマンドです。p: このオプションは、指定されたパスに必要なすべての親ディレクトリを作成することを意味します。例えば、**/usr/local/varが存在しない場合、このオプションによって/usr/local/var**も自動的に作成されます。/usr/local/var/postgres: これは作成するディレクトリのパスです。このコマンドによって、PostgreSQLのデータを格納するためのディレクトリが作成されます。
initdb /usr/local/var/postgres:initdb: このコマンドは、PostgreSQLデータベースシステムの新しいデータディレクトリを初期化するためのものです。初期化には、ディレクトリの構造の作成、システムカタログの設定、テンプレートデータベースの作成などが含まれます。/usr/local/var/postgres: これは**initdbによって初期化されるデータディレクトリのパスを指定するものです。このディレクトリは、上記のmkdir**コマンドによって既に作成されているはずです。
要するに、これらのコマンドは、PostgreSQLのデータディレクトリを作成し、そのディレクトリをデータベースシステムとして初期化するためのものです。
続いて、以下のコードを打って、PostgreSQLサーバーを起動します。ここではポート番号を指定しています。
postgres -D /usr/local/var/postgres -p 5432
以下のような出力が出れば大丈夫です。
2023-10-21 21:50:01.999 JST [10995] LOG: starting PostgreSQL 14.9 (Homebrew) on aarch64-apple-darwin23.0.0, compiled by Apple clang version 15.0.0 (clang-1500.0.40.1), 64-bit
2023-10-21 21:50:02.002 JST [10995] LOG: listening on IPv6 address "::1", port 5432
2023-10-21 21:50:02.002 JST [10995] LOG: listening on IPv4 address "127.0.0.1", port 5432
2023-10-21 21:50:02.002 JST [10995] LOG: listening on Unix socket "/tmp/.s.PGSQL.5432"
2023-10-21 21:50:02.008 JST [10996] LOG: database system was shut down at 2023-10-21 08:18:37 JST
2023-10-21 21:50:02.012 JST [10995] LOG: database system is ready to accept connections
以下のログの詳細を説明します:
LOG: starting PostgreSQL 14.9 (Homebrew) on aarch64-apple-darwin23.0.0, compiled by Apple clang version 15.0.0 (clang-1500.0.40.1), 64-bit: PostgreSQL 14.9がaarch64のMacOS上で起動されていることを示しています。LOG: listening on IPv6 address "::1", port 5432: PostgreSQLがIPv6アドレスでポート5432でリッスンしていることを示します。LOG: listening on IPv4 address "127.0.0.1", port 5432: PostgreSQLがIPv4アドレスでポート5432でリッスンしていることを示します。LOG: listening on Unix socket "/tmp/.s.PGSQL.5432": PostgreSQLがUnixソケットを使用してリッスンしていることを示します。LOG: database system was shut down at 2023-10-21 08:18:37 JST: 以前のシャットダウンのタイムスタンプを示しています。LOG: database system is ready to accept connections: PostgreSQLが接続を受け入れる準備が整っていることを示します。
これでPostgreSQLが正常に動作していることが確認できました。データベースへの操作や他の作業を進めることができます。
続いて、データベースを作成します。command+Tを押して、新しいコマンドプロンプトを作って下さい。その後、PostgreSQL用のユーザーを以下のようで設定します。
createuser -P (お好きなユーザー名)
パスワードを聞かれるので、お使いのパソコンのパスワードを入力して下さい。
続いて、以下のコードをうち、データベースにアクセスして下さい。
psql -d postgres
以下のように出力されると思います。
psql (14.9 (Homebrew))
Type "help" for help.
postgres=#
postgres=# の続きに対して、以下のように記述して下さい。これでデータベースが作成されます。
postgres=# create database chembl_33;
続いて、\\q を実行し、dataベースから退出して下さい。
次に以下のコードを入力して下さい。
ダウンロードし、解凍したchembl_33_postgresqlファイルのディレクトリに行って下さい。
cd (chembl_33_postgresqlファイルのパス)
続いて、以下のコードを実行して下さい。実行が終わるには10分くらいかかります。
pg_restore --no-owner -U kshinba -d chembl_33 chembl_33_postgresql.dmp
- pg_restore: PostgreSQLデータベースのバックアップを復元するためのコマンド。このコマンドは、
pg_dumpコマンドによって作成されたバックアップをデータベースに復元する際に使用します。 - -no-owner: このオプションを使用すると、リストア時にオブジェクトの所有者を復元しません。実際の利用シナリオとしては、バックアップ元のデータベースのユーザーとリストア先のデータベースのユーザーが異なる場合や、権限の問題を回避したい場合などが考えられます。
- U kshinba:
Uオプションは、データベースに接続するユーザー名を指定するためのオプションです。このコマンドではkshinbaというユーザー名でデータベースに接続しています。このkshinbaというのはPCの名前なので、自分のPCの名前に変更して下さい。 - d chembl_33:
dオプションは、リストア対象のデータベース名を指定するオプションです。このコマンドではchembl_33というデータベースにリストアを行っています。 - chembl_33_postgresql.dmp: リストアするバックアップファイルの名前。このコマンドでは
chembl_33_postgresql.dmpというファイルからデータベースchembl_33にデータを復元しています。
要するに、このコマンドは chembl_33_postgresql.dmp というバックアップファイルから、chembl_33 という名前のデータベースにデータを復元するためのもので、その際にオブジェクトの所有者の情報は復元せず、kshinba というユーザー名でデータベースに接続してリストアを行っています。
以上でデータベースの作成は終了です。
pgadmin4を用いた学習データのダウンロード
続いて、以下を実行し、pgadmin4をインストールして下さい。
brew install pgadmin4
pgAdmin 4は、PostgreSQLデータベースのための最も人気のあるオープンソースの管理ツールの1つです。Webベースのインターフェースを持ち、多様な操作システム上で動作することができます。ユーザは、このツールを使って、データベースの管理、SQLクエリの実行、データの視覚化、スキーマの変更など、多岐にわたるタスクを行うことができます。
pgAdmin 4の特徴として、ドラッグアンドドロップのサポート、SQLエディタに統合されたコード補完、複数のデータベースとの同時接続、そしてデータベースのパフォーマンスや健康状態を視覚的にモニタリングできるダッシュボードなどがあります。また、セキュリティ機能も強化されており、ロールベースのアクセス制御やデータの暗号化など、データベースの安全性を確保するための多くのオプションが提供されています。
インストールが終了すると、象のアイコンのアプリが出てきていると思います。

起動すると以下のような画面になっていると思います。
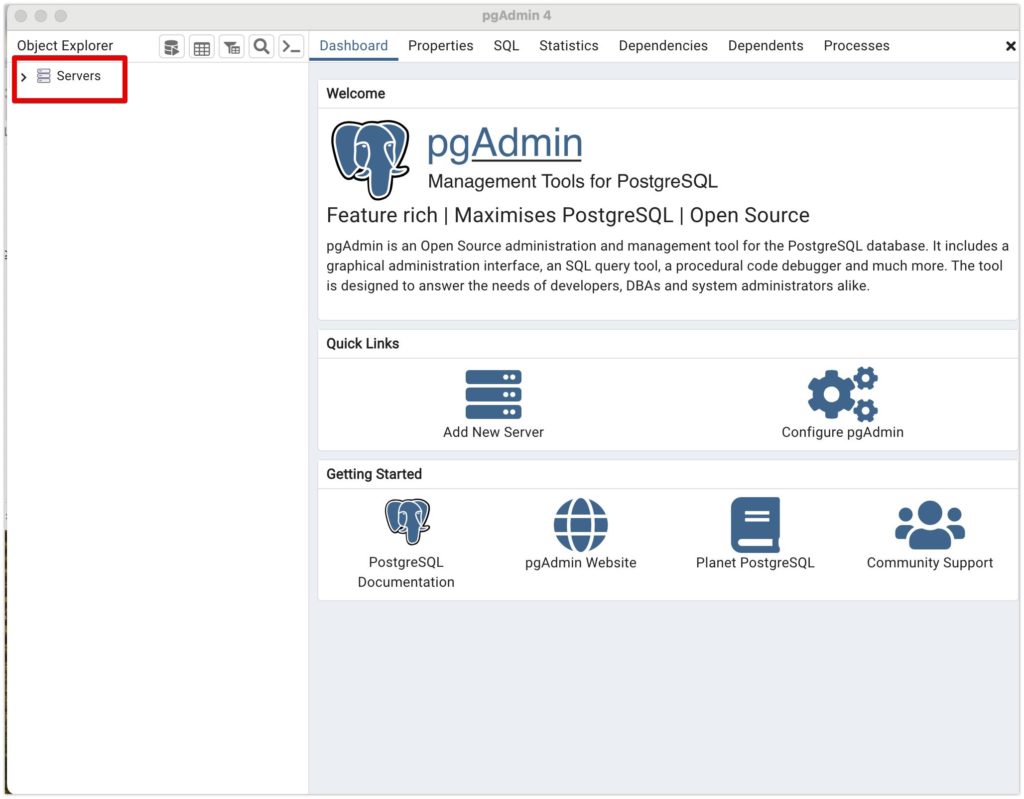
Serversを右クリックし、Resister→Serverをクリックして下さい。
GeneralタブのNameにお好きな名前を入れて下さい。
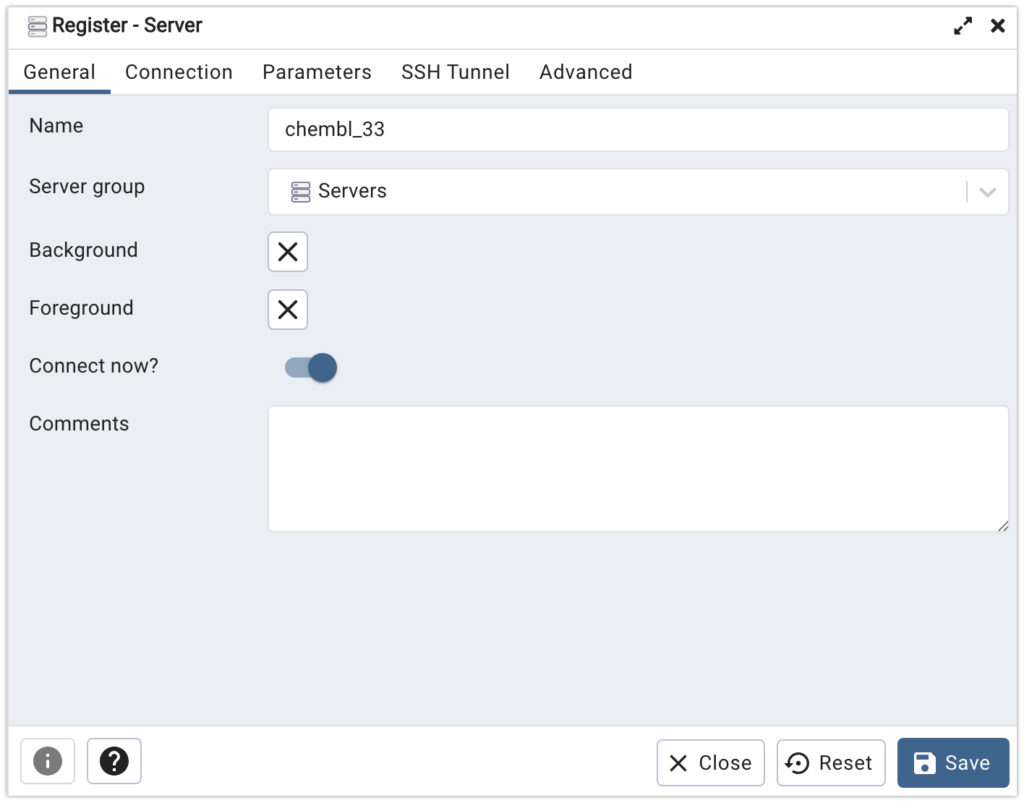
続いて、Connectionタブを以下のように設定して下さい。
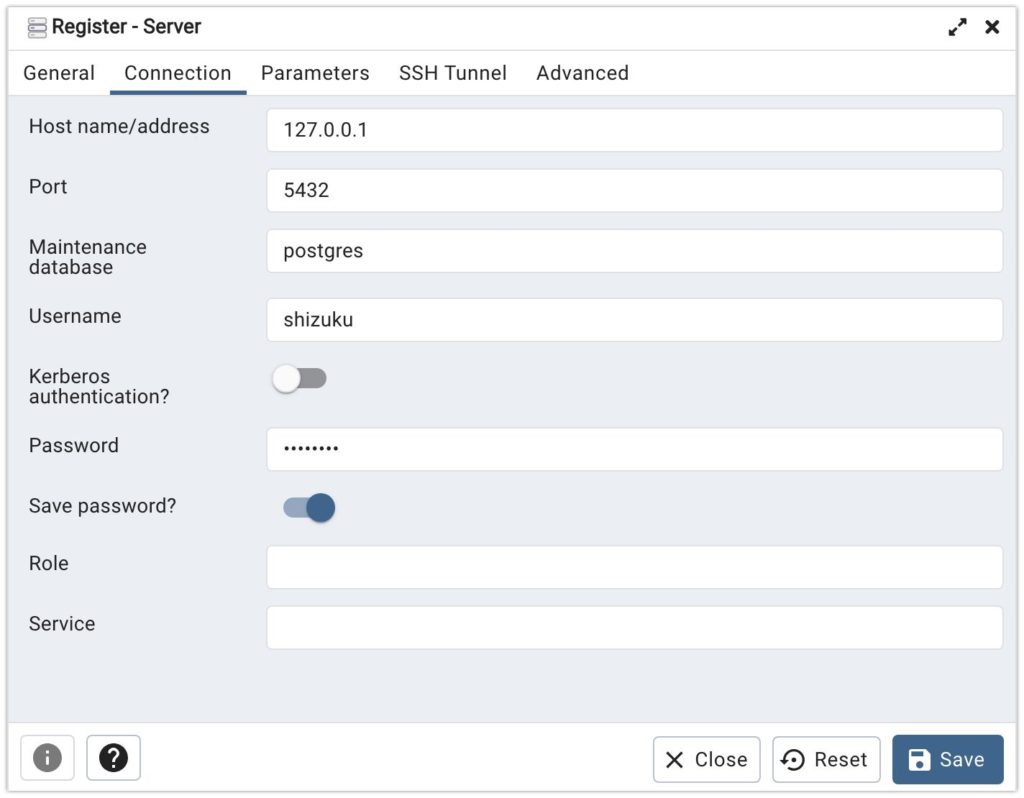
Host name/addressは127.0.0.1を設定します。これはlocal hostと言って、自分のパソコンを示し得ています。Port番号は先ほど設定した5432を設定します。Username、Passwordも先ほど設定したものを設定して下さい。記入したらsaveを押して下さい。
以下のような画面が出てくると思います。
左のタブのchembl_33/Schemas/public/Tablesの中にダウンロードした各化合物データのテーブルがあると思います。
続いて、Tablesを右クリック→Query ToolをクリックするとQueryが開きます。ここでSQL文を書けるようになります。
Tables中にあるtarget_dictionaryではタンパク質と非タンパク質のターゲット情報が含まれているので、ここから今回のコロナの標的に対する情報を見つけ出します。
Queryに以下のコードを打って、上の▶️を押して下さい。
select * from target_dictionary where upper(pref_name) like '%SARS%'
- select:
- は「すべてのカラム」を意味します。このクエリのこの部分は「
target_dictionaryテーブルのすべてのカラムのデータを選択する」という意味になります。
- は「すべてのカラム」を意味します。このクエリのこの部分は「
- from target_dictionary:
- これは、
target_dictionaryという名前のテーブルからデータを選択することを示しています。
- これは、
- where:
- このキーワードは条件を指定するために使用されます。以下の条件式がTrueと評価される行のみが結果として返されます。
- upper(pref_name) like ‘%SARS%’:
upper(pref_name):pref_nameというカラムの文字列を大文字に変換します。**UPPER**関数は文字列を全て大文字に変換する関数です。like '%SARS%': LIKE演算子は、文字列が指定されたパターンに一致するかどうかを確認するために使用されます。ここでのパターン'%SARS%'は、SARSを含む任意の文字列に一致します(%は任意の文字列を示すワイルドカードです)。
全体として、このクエリは「target_dictionary テーブルから pref_name カラムに「SARS」を含む(大文字小文字を区別しない)すべての行のデータを選択する」という意味になります。
以下のような結果になるかと思います。
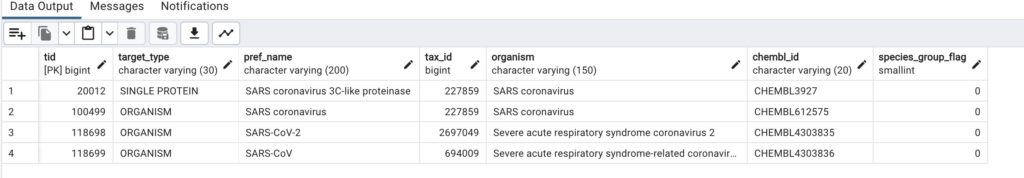
この結果からCHEMBL3927には3C-like Proteaseを標的にした阻害データがあることがわかります。
このページのRetrieve compound activity details for a targetのSQLを参考に、
以下のSQLを実行すると、活性のある化合物の一覧が得られます。
SELECT m.chembl_id AS compound_chembl_id,
s.canonical_smiles,
r.compound_key,
d.pubmed_id,
d.doi,
a.description,
act.standard_type,
act.standard_relation,
act.standard_value,
act.standard_units,
act.activity_comment
FROM compound_structures s,
molecule_dictionary m,
compound_records r,
docs d,
activities act,
assays a,
target_dictionary t
WHERE s.molregno = m.molregno
AND m.molregno = r.molregno
AND r.record_id = act.record_id
AND r.doc_id = d.doc_id
AND act.assay_id = a.assay_id
AND a.tid = t.tid
AND t.chembl_id = 'CHEMBL3927'
AND (act.standard_type ='IC50' or act.standard_type ='Ki')
order by compound_chembl_id;
このSQLクエリは、複数のデータベーステーブルから特定の情報を取得する目的で作成されています。クエリは以下の点について説明されています:
- 選択されたフィールド:
m.chembl_idをcompound_chembl_idとして選択。s.canonical_smiles,r.compound_key,d.pubmed_id,d.doi,a.description。- **
act**テーブルからstandard_type,standard_relation,standard_value,standard_units,activity_commentを選択。
- 使用されるテーブル:
compound_structures(s)molecule_dictionary(m)compound_records(r)docs(d)activities(act)assays(a)target_dictionary(t)
- 結合条件:
- 各テーブルは特定のフィールドに基づいて結合されます。たとえば,
s.molregno = m.molregnoとm.molregno = r.molregnoなど。
- 各テーブルは特定のフィールドに基づいて結合されます。たとえば,
- フィルタ条件:
t.chembl_idが'CHEMBL3927'であるレコードをフィルタリング。act.standard_typeが'IC50'または'Ki'であるレコードをフィルタリング。
- ソート条件:
- 結果は
compound_chembl_idに基づいてソートされます。
- 結果は
具体的には、このクエリは以下のような処理を行っています:
- まず、複数のテーブルを指定されたフィールドに基づいて結合します。この結合は、特定の化合物、文書、アクティビティ、およびアッセイに関連する情報を取得するために行われます。
- 次に、指定されたフィルタ条件に基づいてレコードをフィルタリングします。これは、特定のターゲット (
CHEMBL3927) とアクティビティタイプ (IC50またはKi) に関連する情報だけを取得するためです。 - 最後に、結果を
compound_chembl_idに基づいてソートして、結果セットを整理します。
このクエリにより、関連する化合物、その構造、文書、アクティビティ、アッセイ、およびターゲットに関する詳細な情報を一覧表示することができます。
以下のように取れれば大丈夫です。エクスポートマークからエクスポートしてください。
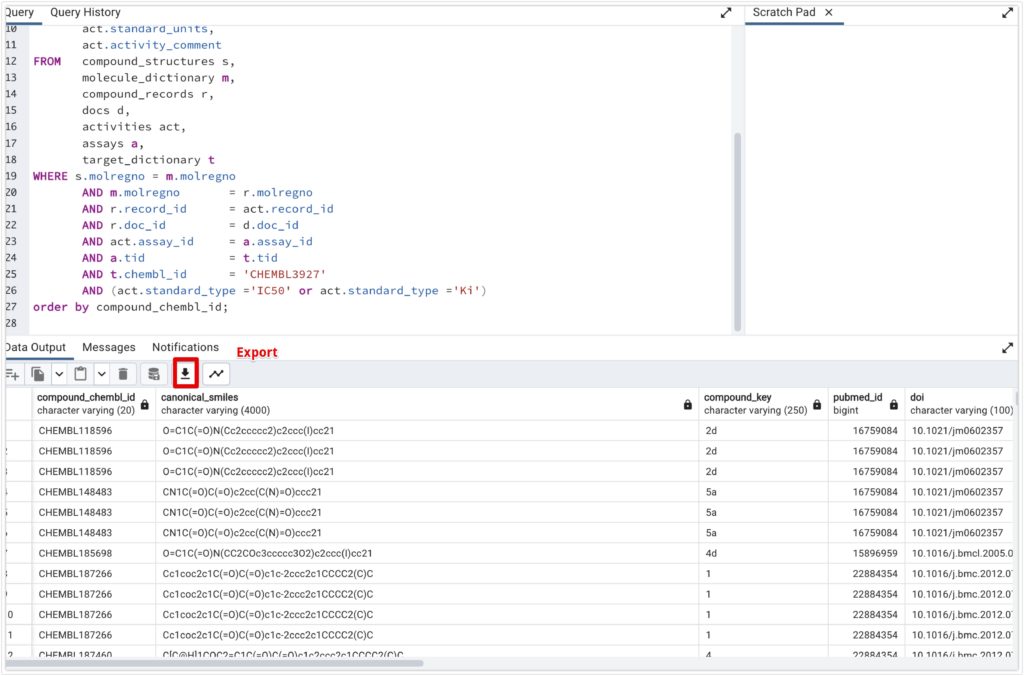
活性非活性データの追加
続いて、Google colabを開き、ダウンロードしたcsvファイルをアップロードします。
私の場合、ダウンロードしたcsvファイルはcompounds of detail.csv としています。
以下のコードを実行してください。 threshold = 10000を閾値として、それ以下のものを活性あり(outcome = 1)、それ以上のものを活性なしとしています。
import csv
from collections import defaultdict
# ファイルパスを直接指定
input_file_path = '/AI _Drug _discovery/compounds of details.csv'
output_file_path = '/AI _Drug _discovery/processed_compounds_of_details.csv'
def process_csv(input_path, output_path):
rows_by_id = defaultdict(list)
# csvを読み込み、CHEMBLID毎にrowをまとめる
with open(input_path, "r") as f:
reader = csv.DictReader(f)
for row in reader:
rows_by_id[row["compound_chembl_id"]].append(row)
# activeとみなす値
threshold = 10000
# CHEMBLID毎に値を処理しながら出力する
with open(output_path, "w") as f:
writer = csv.writer(f, lineterminator="\\n")
writer.writerow(["chembl_id", "canonical_smiles", "value", "outcome"])
# CHEMBLID毎に値を処理
for id in rows_by_id:
# 合計を求める
total = 0.0
for row in rows_by_id[id]:
value = row["standard_value"]
total += float(value)
mean = total/len(rows_by_id[id])
print(f'{id},{mean}')
outcome = 0
if mean < threshold:
outcome = 1
writer.writerow([id, rows_by_id[id][0]["canonical_smiles"], mean, outcome])
# 関数を呼び出して処理を実行
process_csv(input_file_path, output_file_path)
このコードは、Pythonの関数として定義されており、指定された入力ファイルからデータを読み取り、それを処理し、結果を指定された出力ファイルに書き込むことを目的としています。以下は、コードの主要な部分についての説明です。
- ライブラリのインポート:
csv: CSVファイルの読み書きを行うためのライブラリです。defaultdict: 通常の辞書と同様ですが、キーが存在しない場合にデフォルト値を提供する辞書です。
- ファイルパスの指定:
- **
input_file_pathとoutput_file_path**変数を使って、入力と出力のCSVファイルのパスを指定します。
- **
process_csv関数:- この関数は、入力ファイルのパスと出力ファイルのパスを引数として受け取り、データの処理と出力を行います。
- データの読み取り:
with open(input_path, "r") as f:ブロック内で、**csv.DictReader**を使って入力CSVファイルを読み取ります。rows_by_iddefaultdictを使って、各化合物のデータを**compound_chembl_id**キーに基づいてグループ化します。
- 閾値の設定:
- **
threshold**変数に10000を設定し、これは後で活性化の判定に使います。
- **
- データの処理と出力:
with open(output_path, "w") as f:ブロック内で、**csv.writer**を使って出力CSVファイルを作成します。- ヘッダー行を書き込みます:
["chembl_id", "canonical_smiles", "value", "outcome"] - **
rows_by_id辞書をループして、各compound_chembl_id**ごとに処理を行います。- **
standard_value**の合計を計算し、平均値を求めます。 - 平均値が閾値未満の場合、**
outcome**を1に設定し、それ以外の場合は0に設定します。 - 出力ファイルに行を書き込みます。各行には**
chembl_id**,canonical_smiles,mean, **outcome**が含まれます。
- **
- 関数の呼び出し:
- **
process_csv(input_file_path, output_file_path)を使って、process_csv**関数を呼び出し、処理を実行します。
- **
このコードは、指定された入力ファイルから化合物データを読み取り、各化合物に対して**standard_value**の平均値を計算し、その平均値が特定の閾値未満であるかどうかに基づいて活性の判定を行い、そしてその結果を指定された出力ファイルに書き込むことを目的としています。
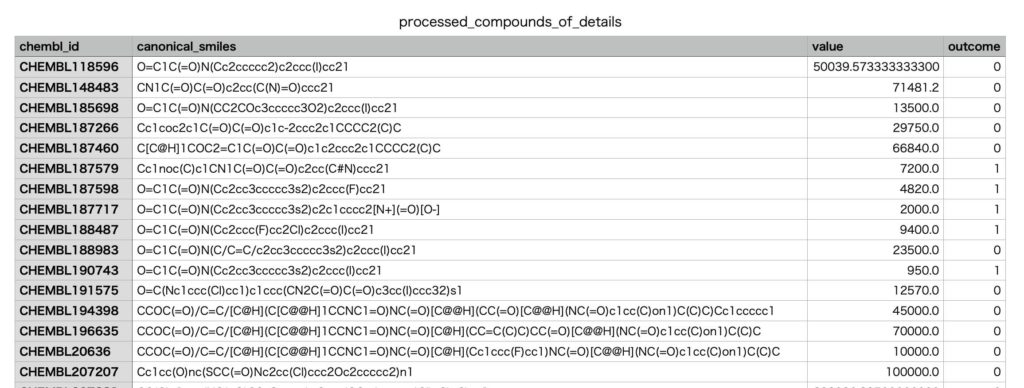
この処理を行うと、新たにprocessed_compounds_of_details.csv が生成していると思います。
最後に
いかがでしたでしょうか?作成したデータは第3章で第2章で得られるデータとマージして、機械学習のモデルの作成に使われます。いきなりDBの作成と少しハードルが高かったかもしれません。DBの作成は沢山の記事やYOUTUBEがあるので、それを参考にしても良いと思います。第2章ではさらにデータを増やすためにPDBのスクレイピングをやっていきます。
謝辞
この記事は以下の論文のフォローとなります。機械学習とAI創薬についてわかりやすく書かれています。このような論文を書いて下った筆者らに感謝を申し上げます。
NCBI – WWW Error Blocked Diagnostic
この記事を書くにあたり、**@kimisyo(晶 公畑)**さんの記事が大変参考になりました。ありがとうございます。





