RNA−seq解析で明らかとなった発現変動遺伝子について、どのような性質の遺伝子が集まっているのか可視化させる方法としてエンリッチメント解析が知られています。
今回は、エンリッチメント解析を行うことができるブラウザツールMetascapeの使い方と結果の見方について書きたいと思います。
ikraを使っている人からすると、出てきたoutput.tsvファイルを使った後続処理として使うことが可能です。発現変動遺伝子についてメタ的な視点で結果を考察できる強力な手法なので、ぜひマスターしていきましょう。
macOS Monterey(12.4), クアッドコアIntel Core i7, メモリ32GB

公共データを用いたRNA-seq解析に関する初心者向け技術書を販売中
¥2,500 → ¥1,500 今なら40%OFF!!
画面キャプチャをふんだんに掲載したわかりやすい解説!
自宅PCからドライ研究が始められます
エンリッチメント解析とは
エンリッチメント解析とは、変動が見られた遺伝子群がどのような性質を持った遺伝子が多いのかに注目する解析です。遺伝子には生物プロセスや分子機能、代謝経路に基づいてアノテーションが付与され分類されています。変動が見られた遺伝子群の中で、同様の生物プロセスや分子機能を持つ遺伝子群が多く見られるのなら、無作為に選ばれたわけではなく、統計的に有意に変動している可能性があります。変動が見られた遺伝子群を考察するための一つの手法です。

GO termエンリッチメント解析とパスウェイエンリッチメント解析
エンリッチメント解析には大きく分けて二種類の解析があります。生物プロセスや分子機能に基づいて割り振られたGO termを用いた GO termエンリッチメント解析と生物学的な代謝経路に基づいたパスウェイエンリッチメント解析があります。
GO term エンリッチメント解析
- 「GO:+7桁」で分類がされており、それぞれに“Lipid Metabolism”, ”Cell Cycle”, “Apoptosis” など、生物学的な機能やイベントを表すタームが振られている
パスウェイエンリッチメント解析
- パスウェイによって分類がされており、それぞれに”Lysosome”, ”Drug metabolism”, ”Metabolic pathways”などのタームが振られています。
- hsaとR-HSAと2つアノテーションがあるのは大まかに以下の理由があります。それぞれ参考にしている出典が違うことを理解しておきましょう。
- hsa…KEGG由来のデータ
- R-HSA…ReactomeDB由来のデータ
Metascape
Metascapeは複数のサイトリソースにまたがってエンリッチメント解析を行えるツールです。ブラウザで動くため、個人のPC環境に用意する必要はありません。
KEGG Pathway, GO Biological Processes, Reactome Gene Sets, Canonical Pathways, CORUM, WikiPathways, PANTHER Pathwayを情報リソースとしており、解析したいデータを投げると、それらサイトを横断した出力を返します。
Metascapeの使い方
Metascapeにはサンプルファイルが用意されていますので、今回はそちらで使い方を学びましょう。様々なフォーマットがありますが、今回はcsvを使います。
Upload File Formatのcsvをクリックしましょう。
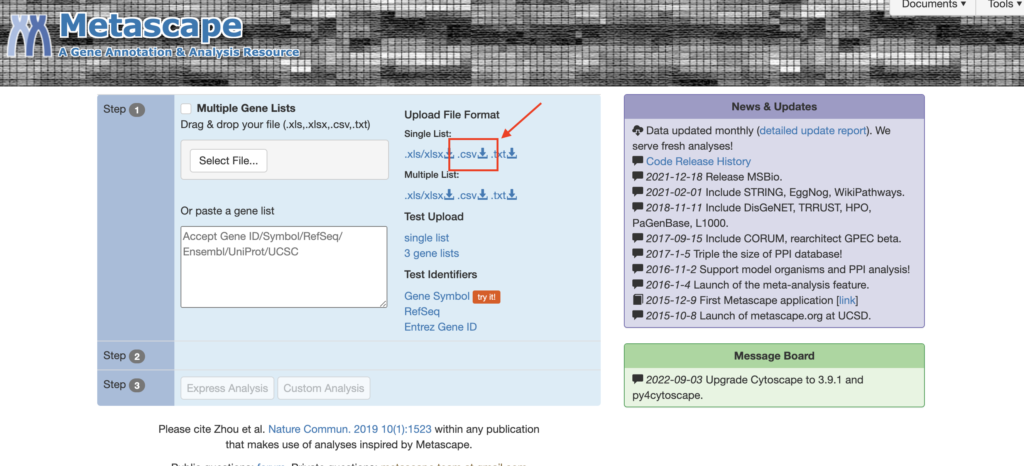
ダウンロードが始まります。好きなところに保存しておいてください。
ちなみにダウンロードしたファイルの中身は以下の通りです。Gene、OptionalDataColumnsという列があります。OptionalDataColumnsは遺伝子の発現量だと考えてください。自分でデータを用意するときも、遺伝子名と発現量の二列のデータを作るといいと思います。
| Gene | OptionalDataColumns |
|---|---|
| UBL5 | 2.6 |
| NDUFB8 | 3.5 |
| CHMP2B | 4.9 |
| PRPF8 | 2.9 |
| NOSTRIN | 4.2 |
| MFAP1 | 3.7 |
| CWC22 | 2.1 |
| PLCH2 | 3.1 |
| PRPF31 | 3.9 |
| ATP6AP1 | 3 |
| DSC3 | 3.6 |
| CLN5 | 4 |
| CHDC2 | 2 |
| (以下略) |
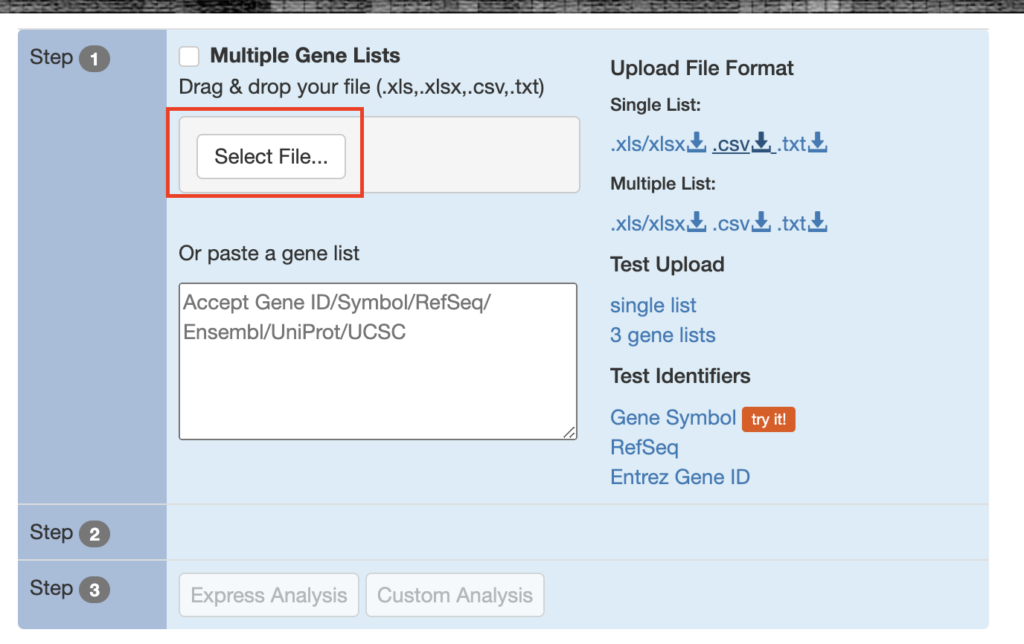
Select Fileをクリックし、ダウンロードしたファイルをアップロードします。
Step2で対象の遺伝子セットを持つ生物種を選びます。今回はサンプルファイルなので、Step2は変えませんが、自身のサンプルを用いてやる場合は変更するようにしてください。
Express Analysisをクリックするとファイルのアップロードが始まります。
アップロードが完了したら、Analysis Report Pageをクリックして結果を見てみましょう。
Metascapeの結果の見方
解析のSummery(Bar Graph Summary)が一番上に棒グラフとして書かれています。グラフ横軸はp値を対数で表したものになります。PDFをクリックすると図表がダウンロードできるので適宜利用してください。
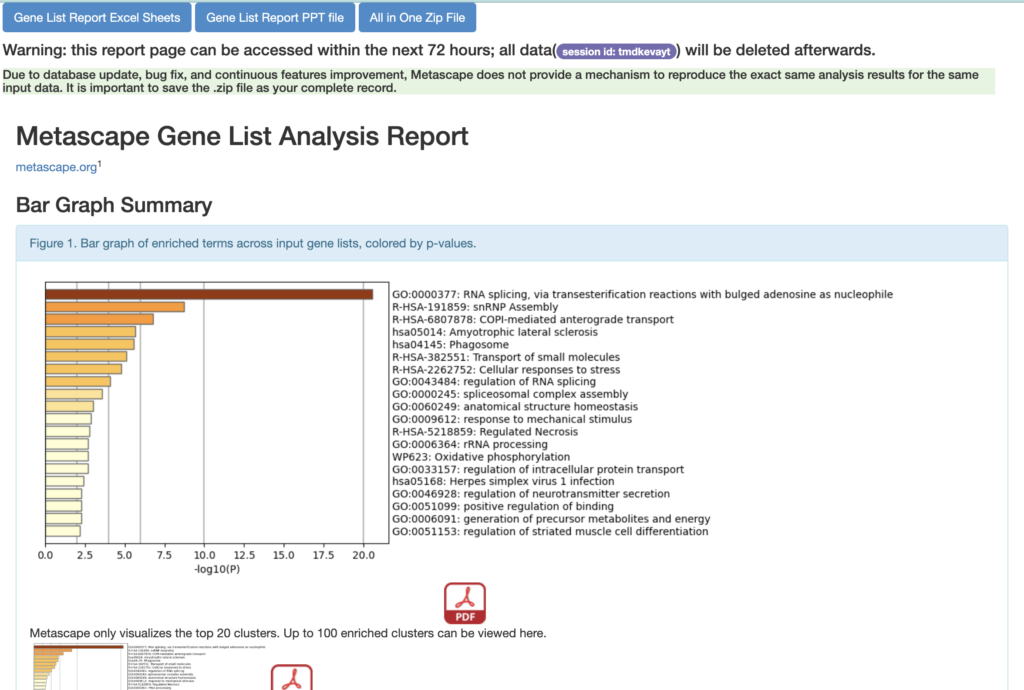
棒グラフ一番上の図は、pathwayとGOtermが合わさった結果が返されています。今回の結果だとGO:0000377:RNA splicing, via transesterification reactions ~が一番です。
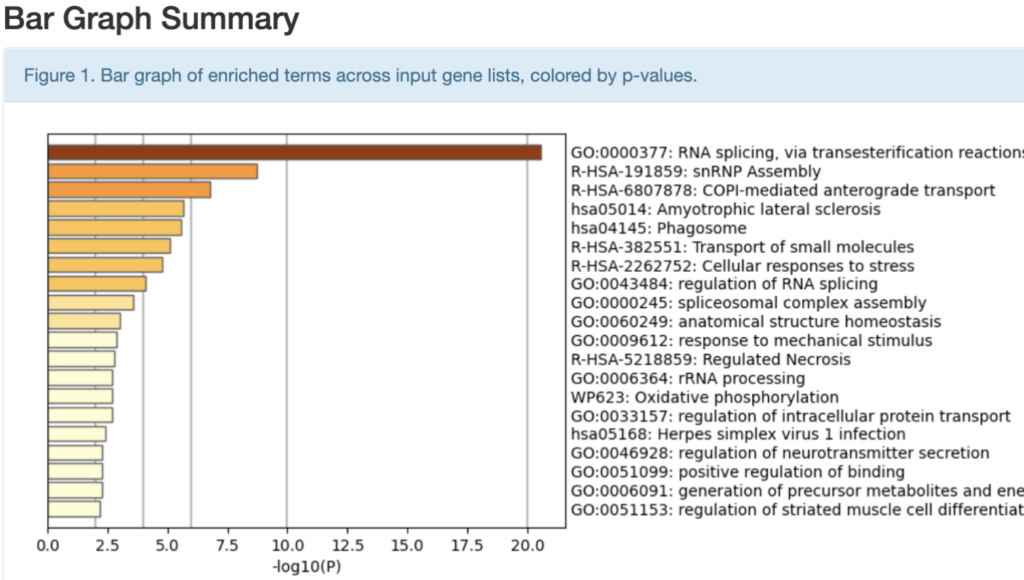
GOエンリッチメント解析の結果だけを確認したければ、下図赤枠の部分で確認できます。
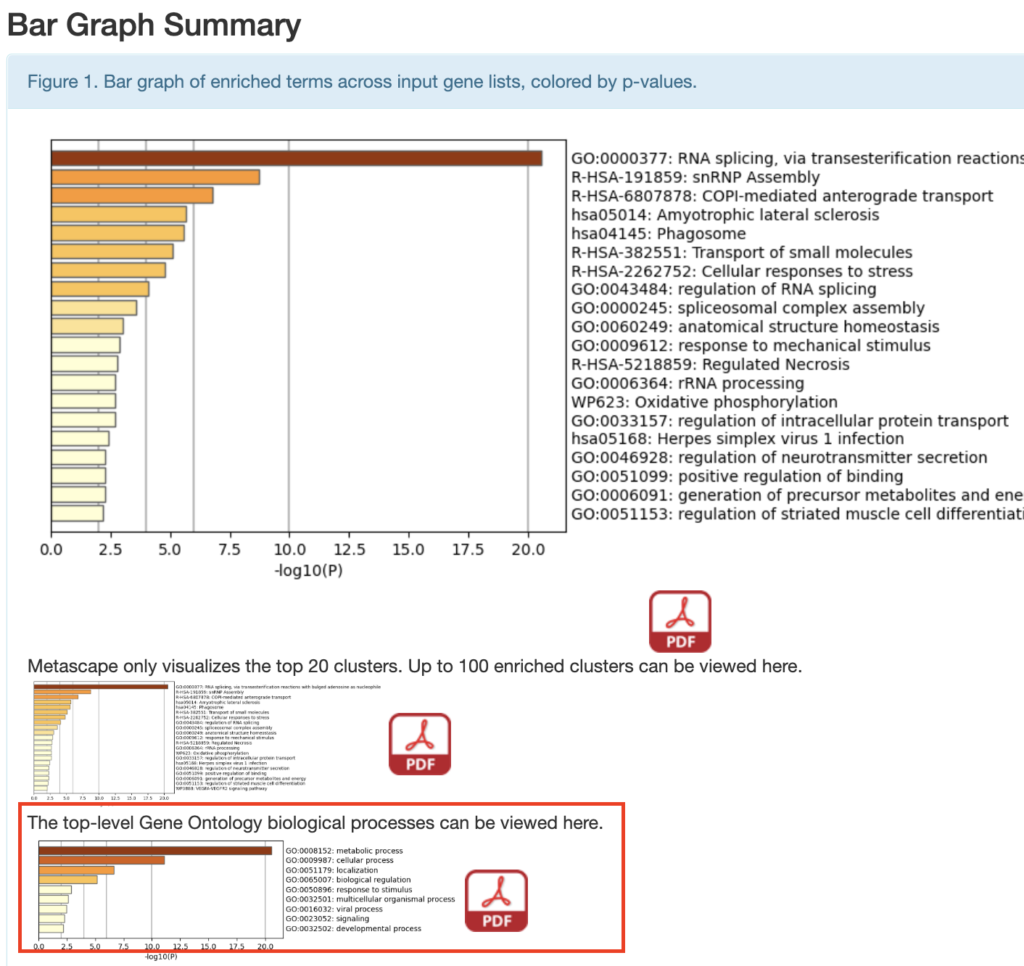
Gene Listsでは解析の対象になった遺伝子数が表されています。元のCSVファイルを見れば分かりますが、解析した遺伝子数は121なので一致しています。

Metascapeでは他にも以下の結果が見れます。
- Pathway and Process Enrichment Analysis
- Protein-protein Interaction Enrichment Analysis
- Quality Control and Association Analysis
Pathway and Process Enrichment Analysis
Pathway and Process Enrichment Analysisでは発現変動が見られた遺伝子についてPathwayと生物プロセスtermに基づいたエンリッチメント解析の結果が見れます。上のBar Graph Summaryのテーブルデータバージョンです。Bar Graph Summaryと同じ結果ですが、こちらは情報の出典としてのCategoryが見られるのと、実際に解析した遺伝子の中でこのプロセスに当てはまる遺伝子数をCountしたデータが見れるので、参考にすると良いです。
例えばSummaryで一番だった、GO:0000377:RNA splicing, via transesterification reactions ~は21遺伝子あったことが確認できます。

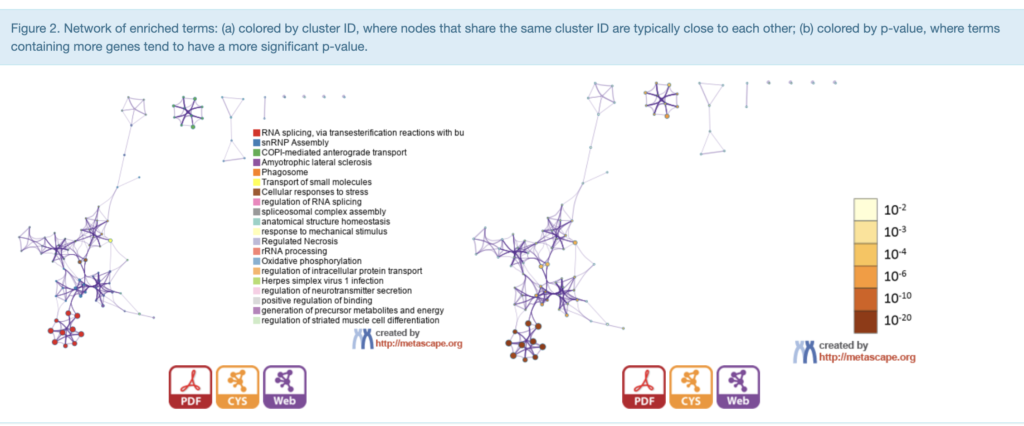
下にスクロールすると、ネットワーク解析の結果が見れます。左の図はtermによってクラスター化されており、それを右図ではp値で塗り分けております。
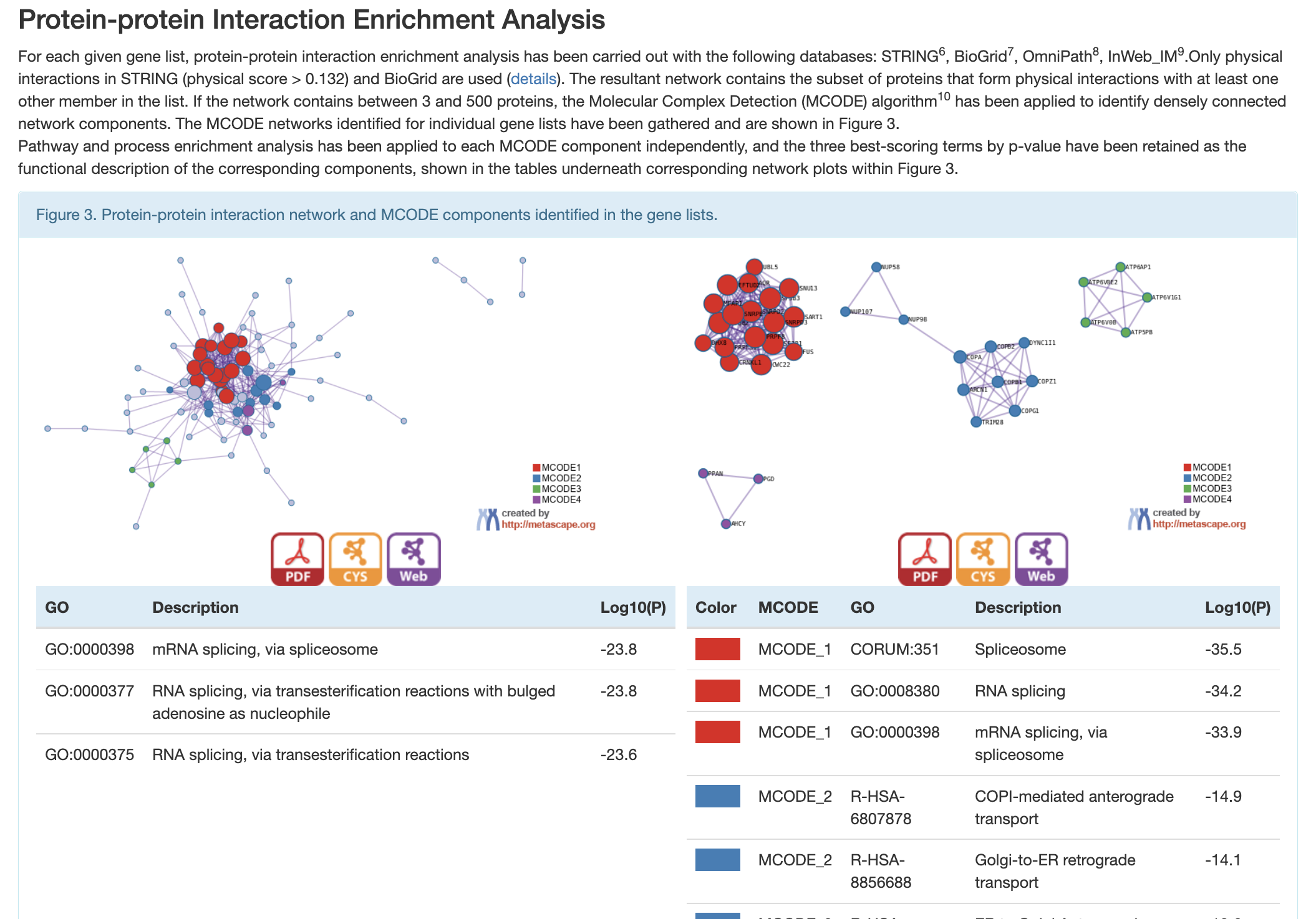
Protein-protein Interaction Enrichment Analysis
Protein-protein Interaction Enrichment Analysisではタンパク質-タンパク質相互作用のエンリッチメント解析がなされており、物理的相互作用をネットワーク図で可視化させています。STRING6, BioGrid7, OmniPath8, InWeb_IMといったタンパク質-タンパク質相互作用を可視化させるツールを情報源にしております。
パスウェイおよびプロセスのエンリッチメント分析を各MCODE構成要素に適用して、p値で最もスコアの高い3つの用語を対応する構成要素の機能を説明するために残しています。
Quality Control and Association Analysis
Quality Control and Association Analysisではオントロジーカテゴリーによってエンリッチメント解析された結果が見れます。現在対応しているカテゴリは以下になります。
- COVID
- Cell_Type_Signatures
- DisGeNET
- TRRUST
- Transcription_Factor_Targets
Metascapeの結果を出力する
Metascapeでは得られた結果をppt形式でダウンロードできます。結果の一番上にスクロールし、Gene List Report PPT fileをクリックするとダウンロードが始まります。
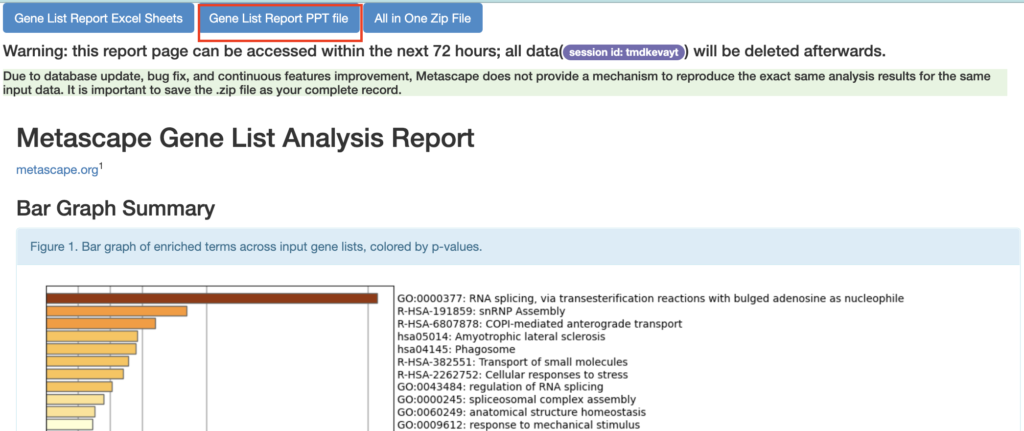
開いて見ると図表が掲載されたpptファイルがダウンロードされています。
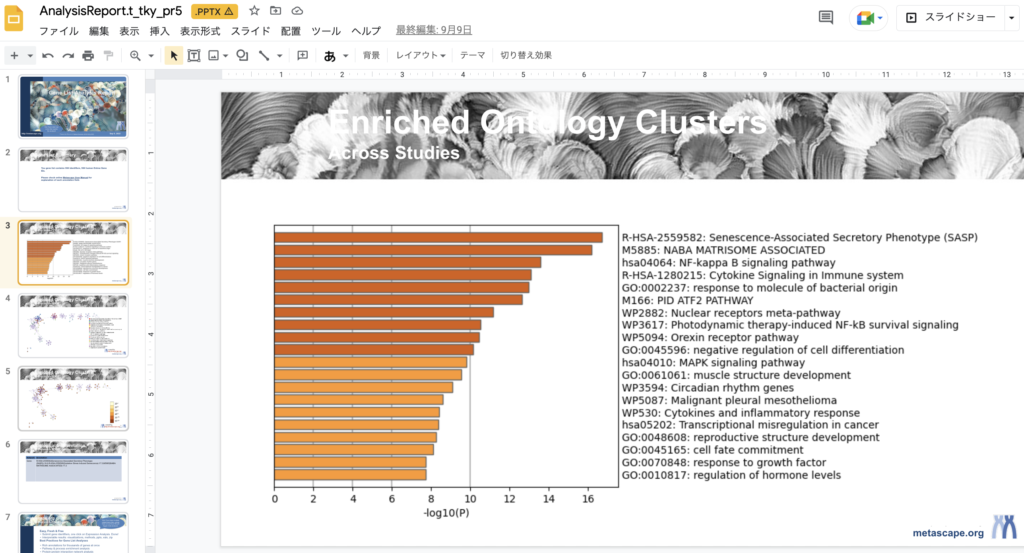
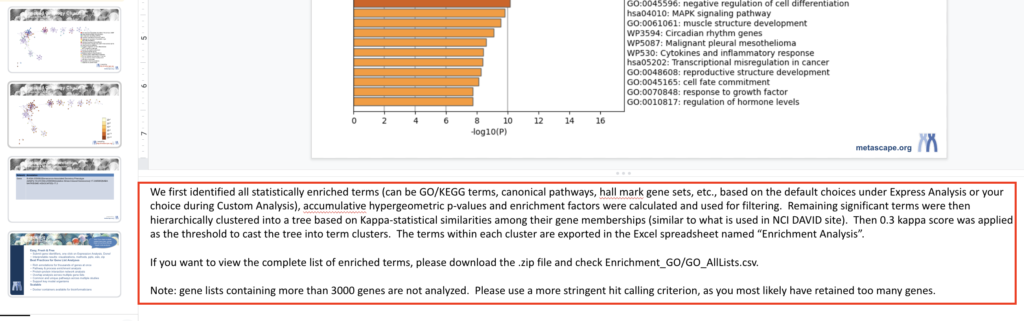
またパワポの説明欄には、実際に行った解析方法が記載されているので、読んで理解を深めることができます。論文のマテメソなどに利用することも可能です。
終わりに
いかがだったでしょうか。メタ解析やトランスクリプトーム解析を行えばエンリッチメント解析は必ずと言っていいほど使われます。結果全体を俯瞰的に見て考察ができる手法となっていますので、ぜひマスターしていきましょう。

公共データを用いたRNA-seq解析に関する初心者向け技術書を販売中
¥2,500 → ¥1,500 今なら40%OFF!!
画面キャプチャをふんだんに掲載したわかりやすい解説!
自宅PCからドライ研究が始められます



