RNA-seqデータからタンパク質間相互作用(PPI)解析をやってみたいと思いませんか?
本記事ではPPI解析によく使われるデータベースであるSTRINGを解説します。またSTRINGの情報を使ってCytoscape上でPPIネットワークを描画する方法を記載します。
これによって、RNA−seq解析のデータにタンパク質レベルでの洞察を与えることが可能になります。是非挑戦してみましょう。
今回記述するCytoscapeのバージョンは3.10.1 になります。バージョンによってはUIや機能仕様に変化がある可能性がありますので、異なるバージョンを使用する場合はご注意ください。
*Cytoscapeのインストールや詳細な使い方やこちらの記事を参考にしてください。
macOS Monterey (12.4), Quad-Core Intel Core i7, Memory 32GB Cytoscape 3.10.1

公共データを用いたRNA-seq解析に関する初心者向け技術書を販売中
¥2,500 → ¥1,500 今なら40%OFF!!
画面キャプチャをふんだんに掲載したわかりやすい解説!
自宅PCからドライ研究が始められます
タンパク質間相互作用ネットワーク解析とは?
タンパク質間相互作用ネットワーク解析(Protein-Protein Interaction Network Analysis)は、タンパク質同士がどのように相互作用し、生物学的プロセスやシグナル伝達経路に関与するかを研究する方法です。この解析は、細胞内のタンパク質が形成する複雑なネットワークを理解するために使用されます。
- ネットワークの構築: 同定された相互作用を基に、タンパク質間の関係を示すネットワークを構築します。このネットワークは、ノード(タンパク質)とエッジ(相互作用)で構成されます。
- データの解析と解釈: 構築されたネットワークを分析して、重要なタンパク質、相互作用のハブ、機能的なクラスターなどを同定します。これには、ネットワークトポロジーの解析や機能的注釈などが含まれます。
PPIは、細胞の振る舞いや疾患のメカニズムを理解するための洞察を提供します。これにより、新しい治療標的の同定や、薬剤の副作用などの予測に役立てられます。このように、タンパク質間相互作用ネットワーク解析は、分子生物学、生化学、システム生物学など多岐にわたる分野で重要なツールとなっています。
ref: Construction and Analysis of Protein-Protein Interaction Network of Heroin Use Disorder
Stringとは?

「STRING」は、タンパク質間相互作用ネットワーク解析に使用されるデータベースの一つで、その名称は「Search Tool for the Retrieval of Interacting Genes/Proteins」の略です。このデータベースは、広範なタンパク質間相互作用情報を提供し、生物学的な研究やシステム生物学の分析に役立てられています。STRINGデータベースの主な特徴は以下の通りです。
- 幅広い種のカバレッジ: STRINGは多くの生物種にわたるタンパク質間相互作用情報を含んでいます。これにより、様々な生物学的文脈での解析が可能になります。
- 異なる情報源からのデータ: STRINGは、実験的に検証された相互作用だけでなく、遺伝的相関、テキストマイニング、データベースの予測情報など、多様な情報源からのデータを統合しています。
- 相互作用の信頼性スコア: 各タンパク質間相互作用には、信頼性のスコアが割り当てられており、これによりデータの品質を評価することができます。
- 直感的なユーザーインターフェースとビジュアライゼーションツール: STRINGは、相互作用ネットワークを視覚的に表示し、簡単に操作できるインターフェースを提供します。これにより、非専門家でも利用しやすいです。
- 機能的な情報との統合: STRINGでは、タンパク質間相互作用に加えて、機能的なアノテーションやパスウェイ情報も提供され、より詳細な生物学的解釈が可能になります。
研究者はSTRINGを使用して、タンパク質間相互作用ネットワークを構築し、それを分析して生物学的な洞察を得ることができます。これは、疾患のメカニズムの理解、新しい治療標的の同定、あるいはシステムレベルでの生物学的プロセスの解明に役立ちます。
今回はString dbの情報を使って、Cytoscape上でタンパク質相互作用ネットワーク解析をやってみようと思います。
Cytoscapeにプラグインを読み込ませる
解析を始める前に使用するプラグインを読み込ませる必要があります。
stringApp: stringAppはCytoscape用のプラグインで、生物学的ネットワークの分析と視覚化を行うために使われます。STRING(Search Tool for the Retrieval of Interacting Genes/Proteins)データベースからの情報を利用して、タンパク質間の相互作用ネットワークを生成します。stringAppは、タンパク質間の相互作用の強さや信頼性を示すエッジ(線)のスコアや、機能的なアノテーションをネットワークに統合する機能を提供します。これにより、タンパク質の機能的クラスターやバイオロジカルパスウェイの解析が容易になります。
ツールバーのApp Storeをクリックしてください。こちらはプラグインを管理するところになります。Cytoscapeの上ではプラグインはAppと表記されるのでご注意ください。Appを検索できるバーが表示されるので、「stringApp」と打ち込んでください。
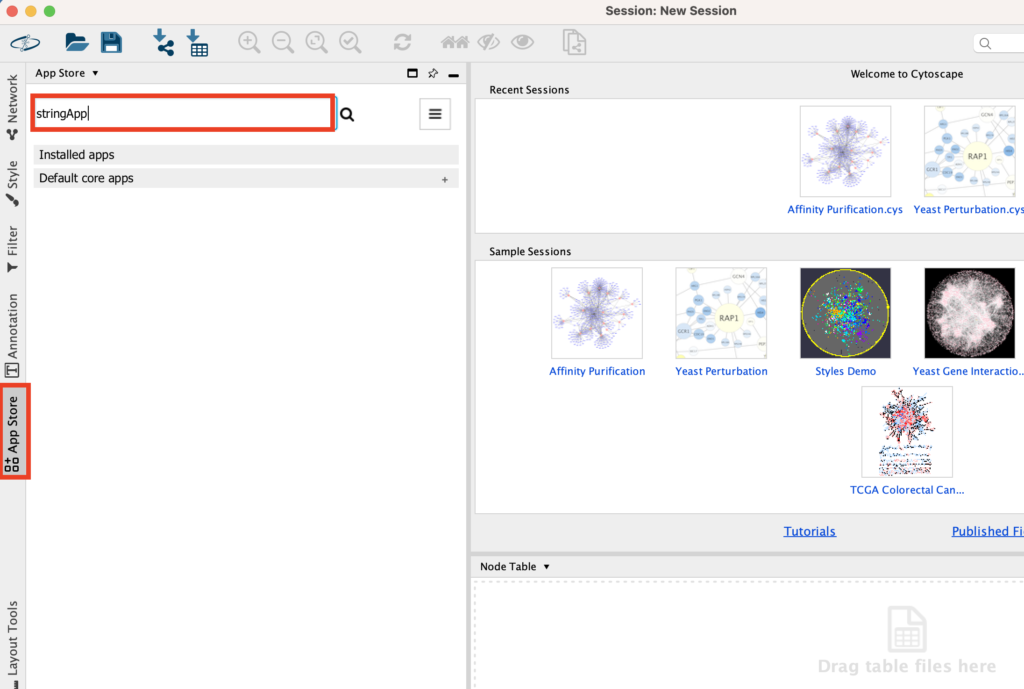
すると自動でAppStoreに遷移します。ここで、stringAppを選びましょう。
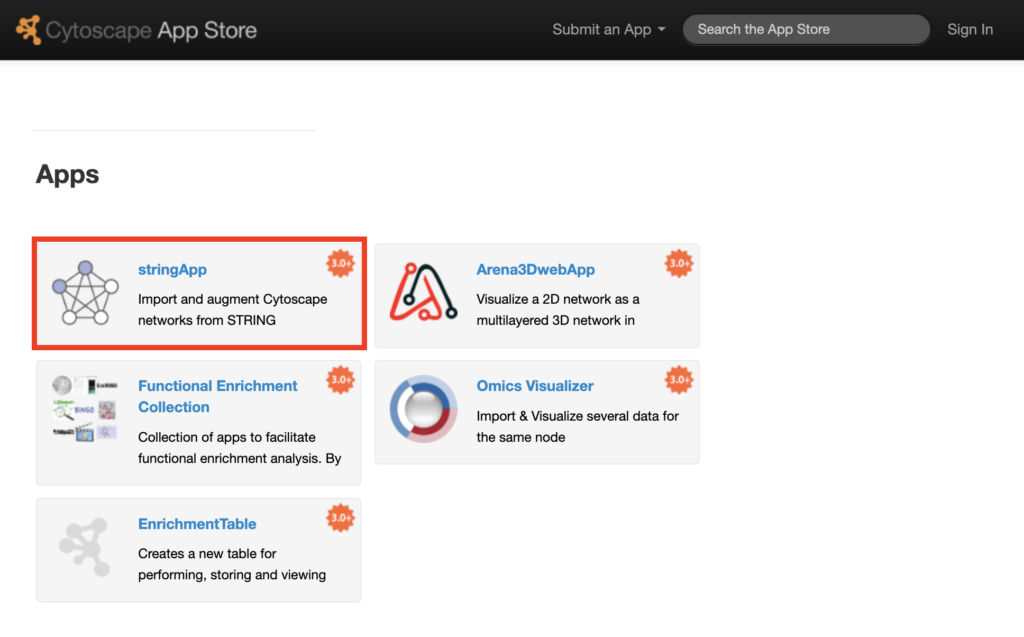
右のInstallを押してインストールをしていきましょう。インストールが完了するとボタンが「Installed」になります。
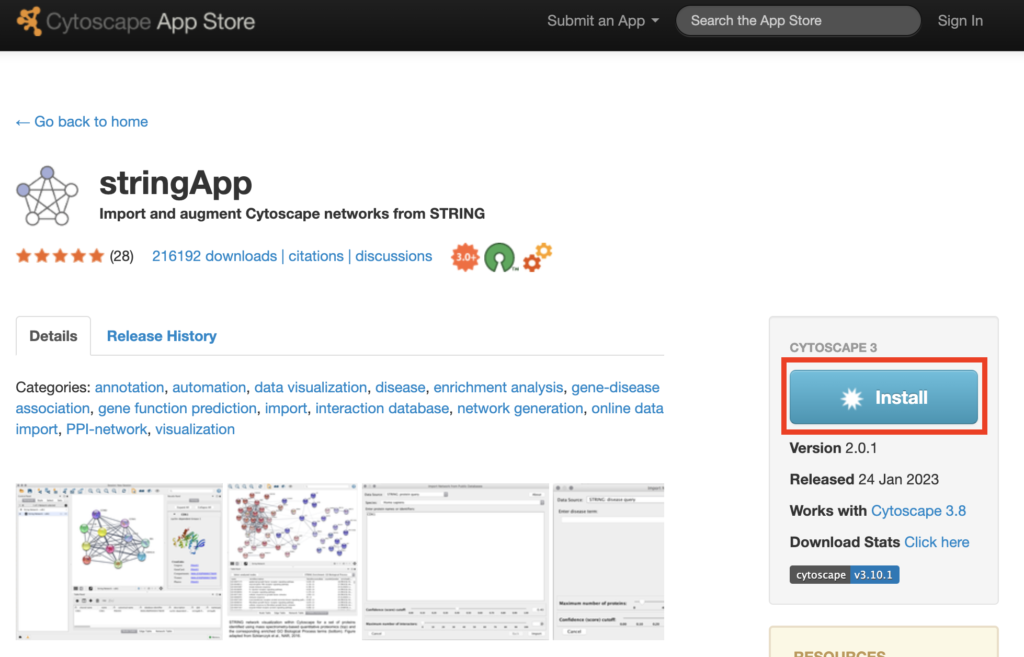
Cytoscapeの画面でstringAppがインストールされたことを確認してください。
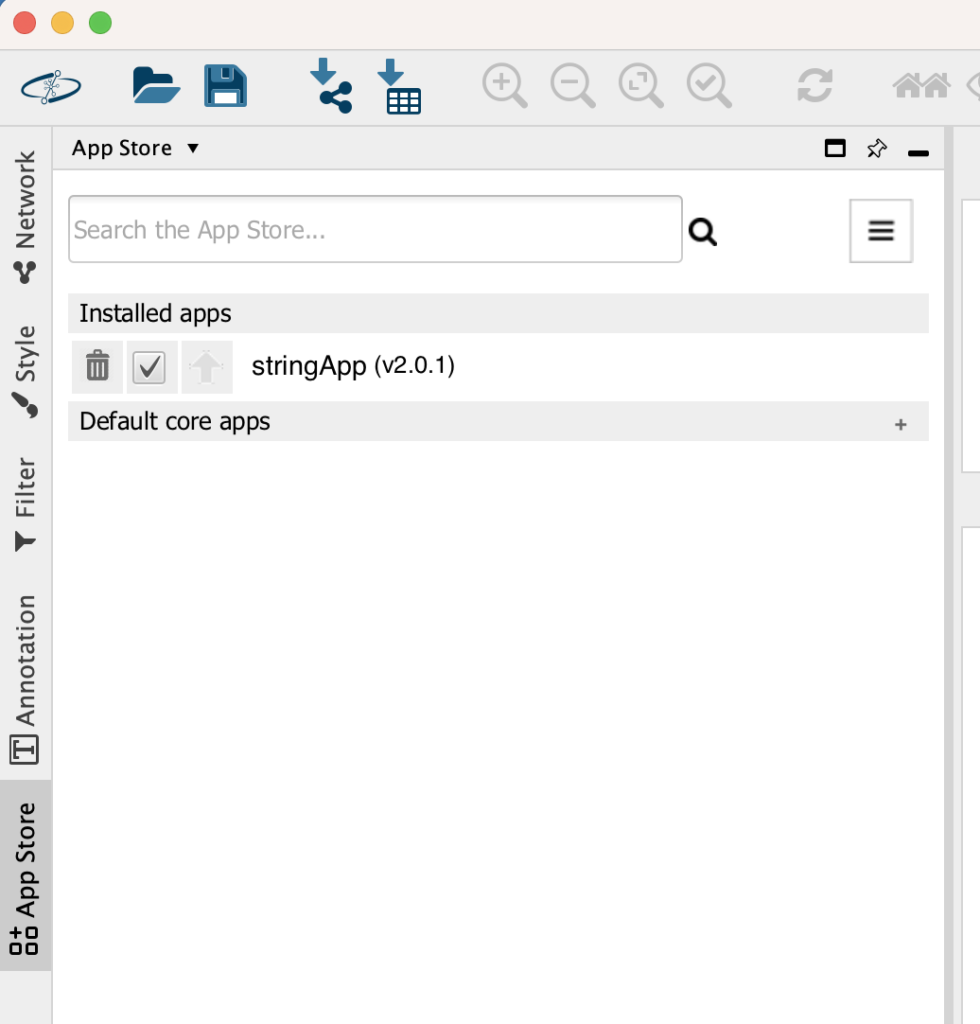
これでプラグインのインストールは完了です。
CytoscapeでstringAppを使ってPPIを描画する
それでは実際にRNA-seqのDEGデータを読み込ませて見ましょう。今回は以下の100個の遺伝子リストをDEGとして扱ってみます。実際に解析する際は自分のデータに置き換えてください。
UBL5,NDUFB8,CHMP2B,PRPF8,NOSTRIN,MFAP1,CWC22,PLCH2,PRPF31,ATP6AP1,DSC3,CLN5,CHDC2,PIP,ZNF473,DHX8,RAB5A,NUP98,NUPL1,HRK,SLC41A3,SNRPD3,SNRPD2,PCGF6,GSK3A,SLC2A2,ARCN1,ANGPTL3,COPB1,COPB2,STARD5,CYB5R4,DMD,FUS,COPS6,NOS3,SFXN2,CNTN5,IQSEC1,CRADD,STAC3,COPZ1,SULT1C4,EFTUD2,SLC35A1,B4GALT2,THRSP,NHP2L1,ZNF224,NXF1,PDLIM3,SAMM50,MTFR1,SART1,PCDHGA1,GINS2,MPG,GJA3,UBE2A,ESAM,CLK3,EIF4A2,NUTF2,INMT,SCYL3,XIAP,TRIM28,MPZL1,LY6G6C,RBM14,TPRX1,ATP6V0B,NUP107,ABCD1,TGS1,RPS4X,CRNKL1,YTHDC1,PPAN,PRRT1,KRTCAP2,GDPD5,ZNF132,WDR18,ATP5F1,AMOTL1,RPS16,CCDC74B,CCDC74A,SF3B1,SF3B3,SF3B2,MYOCD,MLKL,PRSS8,CACTIN,EIF2S1,ZNF16,PGD,SRP54,AQR,DYNC1I1,DCLRE1B,CALCOCO2,EVC2,LRP1B,ZNF552,COPG1,EPRS,COPA,ATP6V1G1,PIEZO1,FCGR2A,CPLX1,SNRPB,BACE1,ZNF154,RAB29,ATP6V0E2,AHCY,SLC1A3
それではデータのインポートをしていきましょう。
File > Import > Network from Public Databases… を選びます。
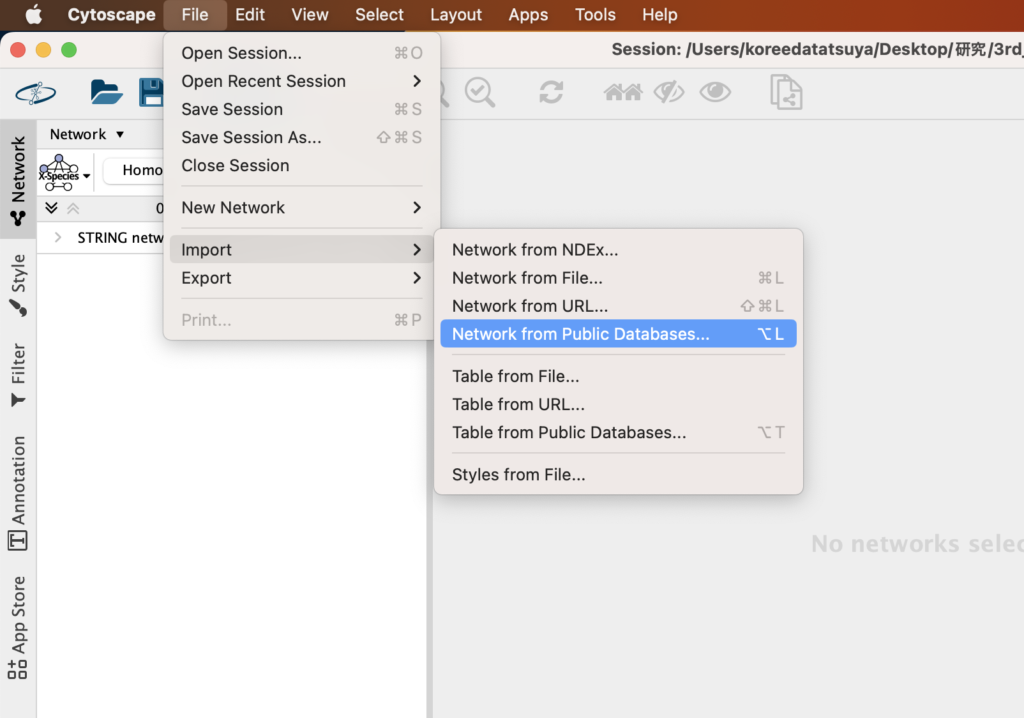
すると、STRINGデータベースからプロテイン相互作用ネットワークをインポートするためのUIが出てきますので必要な情報を入力していきます。Data Sourceを「STRING: protein query」、Speciesは「Homo sapience」、Enter protein names or identifiersで調べたい遺伝子名を入れます。ちなみに今回は、遺伝子名はSYMBOLでインポートしていますが、CytoscapeはENSEMBL ID(例:”ENSG00000012048″)、RefSeqアクセッション番号(例:”NM_007294″)、UniProt ID (例:”P38398″ )など多様な形式をサポートしています。
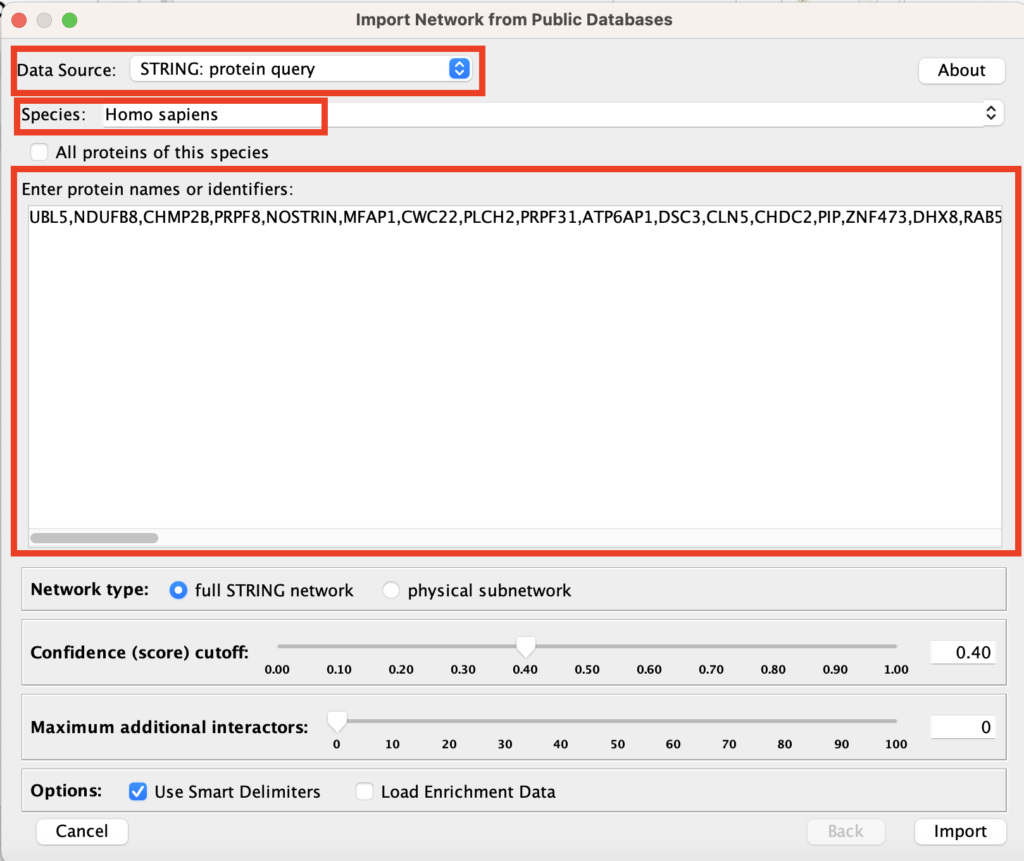
各オプションは以下のようになっています。
- Network type: 「full STRING network」は全ての利用可能な相互作用情報を含むネットワークを生成し、「physical subnetwork」は物理的なタンパク質相互作用に限定したサブネットワークを生成します。
- Confidence (score) cutoff: このスライダーは、ネットワークに含める相互作用の信頼性スコアのしきい値を設定します。高い値を設定するほど、より信頼性の高い相互作用のみがネットワークに含まれます。
- Maximum additional interactors: 追加される相互作用者(インタラクター)の最大数を指定します。これにより、元のクエリに含まれていないが、相互作用があると予測されるタンパク質をネットワークに追加することができます。
- Options:
- Use Smart Delimiters: このオプションを選択すると、入力されたタンパク質名や識別子を適切に区切るためのスマートデリミターが使用されます。
- Load Enrichment Data: このオプションを選択すると、タンパク質のセットに対してエンリッチメント解析用のデータが読み込まれ、分析が行われます。
今回は一旦デフォルトのままで設定します。終わったら「Import」ボタンをおしてImportを完了させてください。
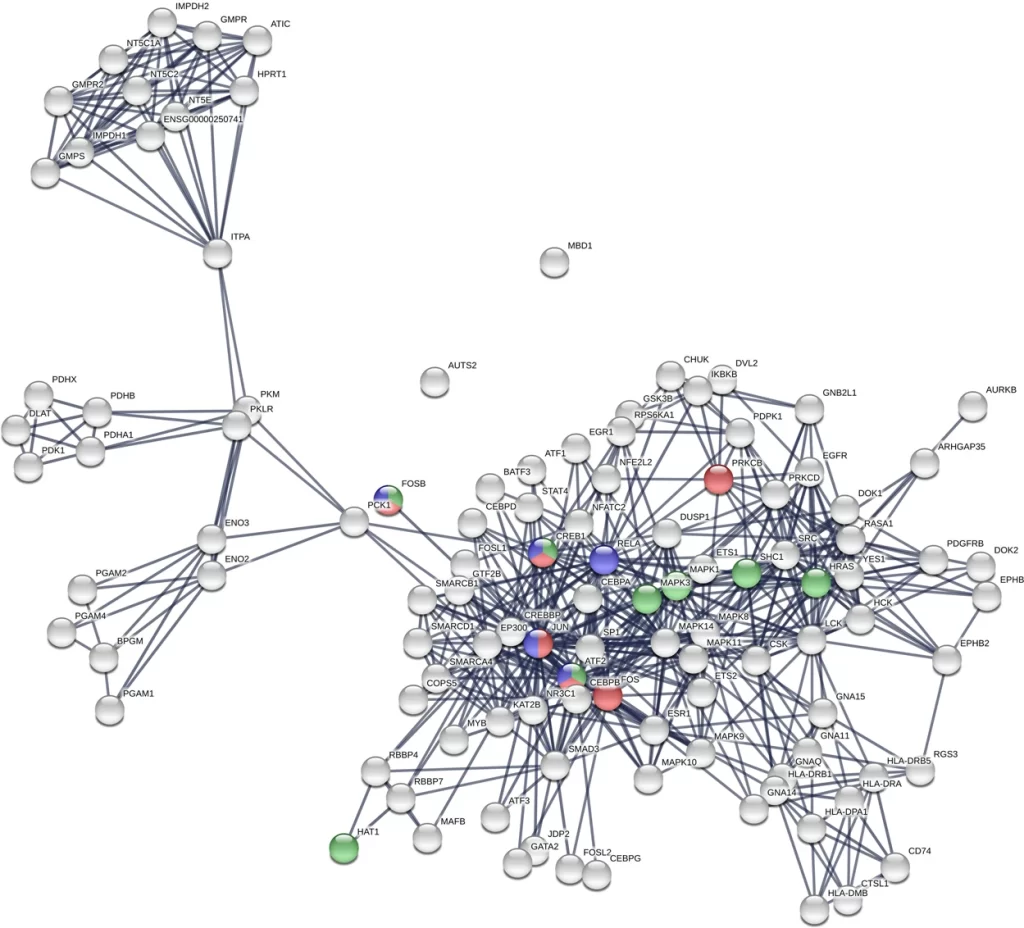
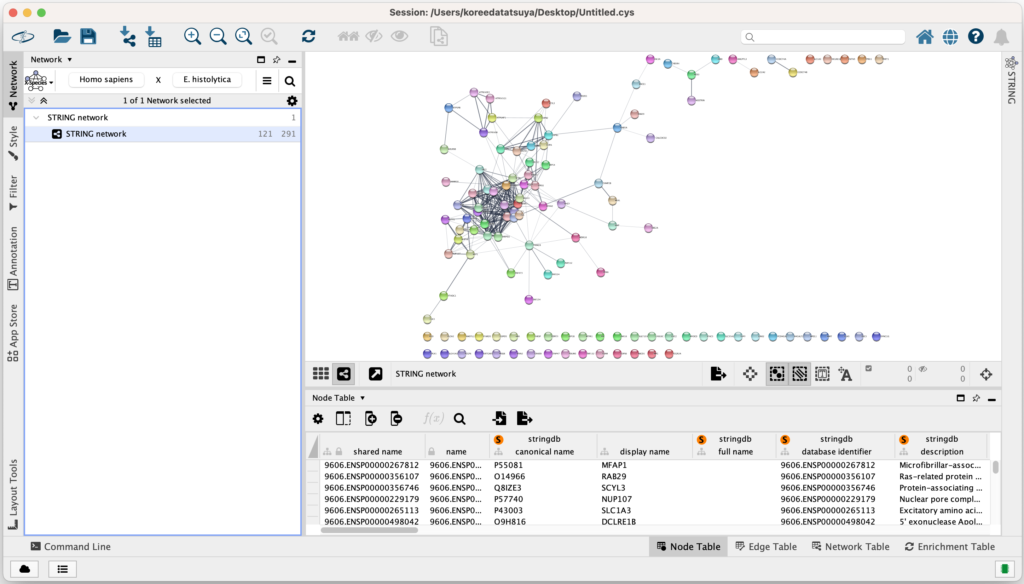
以下のようなPPIネットワーク図が出てきたら成功です!
最後に
いかがだったでしょうか。PPI解析ができると、RNA−seq解析のデータにタンパク質レベルでの洞察を与えることが可能です。ぜひとも習得してみましょう。

公共データを用いたSingle Cell RNA-seq解析に関する初心者向け技術書を販売中
¥3,600 → ¥1,800 今なら50%OFF!!
プログラミング初心者でも始められるわかりやすい解説!
RとSeuratで始めるSingle Cell RNA-seq解析!



