RNA-seqデータから疾患や発現のキーとなる遺伝子を探してみたいと思いませんか?
Cytoscapeを使って遺伝子ネットワーク解析をすれば、あなたも疾患や発現のキーとなる遺伝子を発見することができます。
この記事ではCytoscapeの基本的な使い方を紹介するので、Cytoscapeをインストールして遺伝子ネットワーク解析に入門してみましょう。
今回記述するCytoscapeのバージョンは3.10.1になります。バージョンによってはUIや機能仕様に変化がある可能性がありますので、異なるバージョンを使用する場合はご注意ください。
macOS Monterey (12.4), Quad-Core Intel Core i7, Memory 32GB Cytoscape 3.10.1

公共データを用いたRNA-seq解析に関する初心者向け技術書を販売中
¥2,500 → ¥1,500 今なら40%OFF!!
画面キャプチャをふんだんに掲載したわかりやすい解説!
自宅PCからドライ研究が始められます
遺伝子ネットワーク解析(Gene Network Analysis)とは
遺伝子ネットワーク解析は、遺伝子間の相互作用や関連性を理解するための手法です。このアプローチは、遺伝子発現データ、タンパク質相互作用データ、遺伝的関連性データなどを利用して、生物学的プロセスや疾患のメカニズムを解明するために用いられます。
特にRNA-seqデータをネットワーク解析に適用することで、遺伝子間の相互作用や関連性をより深く探ることが可能になり、どの遺伝子がクリティカルに表現型に効いているのか、ネットワークのHub遺伝子を特定することが可能になります。
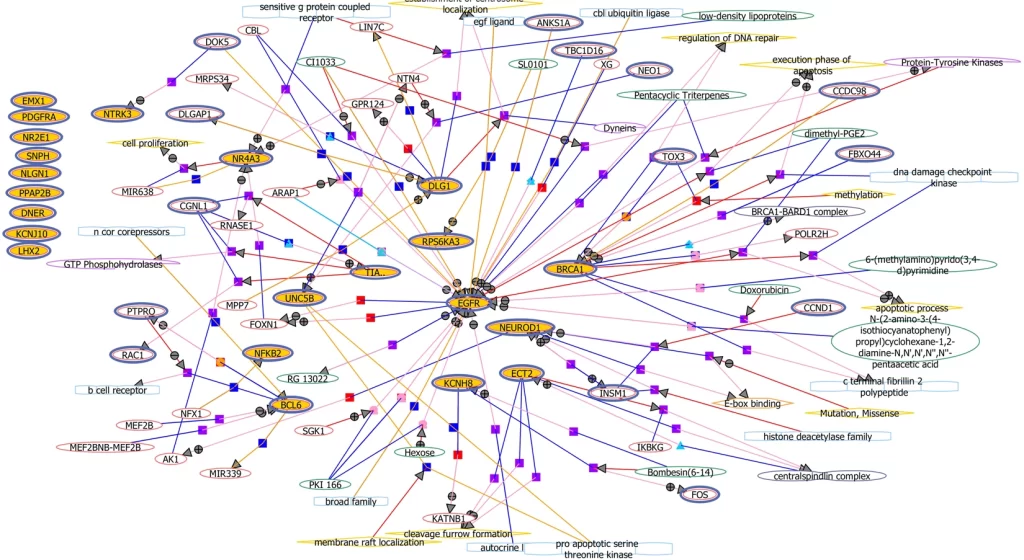
Cytoscapeとは?
Cytoscapeは、生物学的ネットワークを可視化、解析、および理解するための強力なオープンソースのソフトウェアプラットフォームです。遺伝子のみならずタンパク質や代謝産物など様々な相互作用関係をネットワークとして可視化することができます。

https://cytoscape.org/cytoscape-tutorials/presentations/network-analysis-ebi-2021.html より引用
Cytoscapeは活発に開発されておりこちらから確認することができます。
Cytoscapeの動作速度はロード、表示、操作したいネットワークのサイズに依存します。以下に、小規模ネットワークの可視化と大規模ネットワークの解析/可視化に対する要件を簡単に補足説明します
| 要件 | 小規模ネットワーク可視化 | 大規模ネットワーク解析/可視化 |
|---|---|---|
| プロセッサ | 1GHz | できるだけ高速で、複数コア |
| メモリ | 512MB | 2GB以上 |
| グラフィックスカード | 統合ビデオ | ハイエンドグラフィックスカード |
| モニター | XGA (1024×768) | ワイドまたはデュアルモニター |
Cytoscapeの魅力の一つは、その豊富なプラグイン(アプリとも呼ばれる)です。これらのプラグインは、ユーザーが特定の分析やビジュアライゼーションのニーズに合わせてCytoscapeの機能を拡張することを可能にします。
Cytoscapeのプラグインリストは、Cytoscape App Store(http://apps.cytoscape.org/)で確認でき、Cytoscape開発チームによって知られているすべての公開プラグインがリストアップされています。
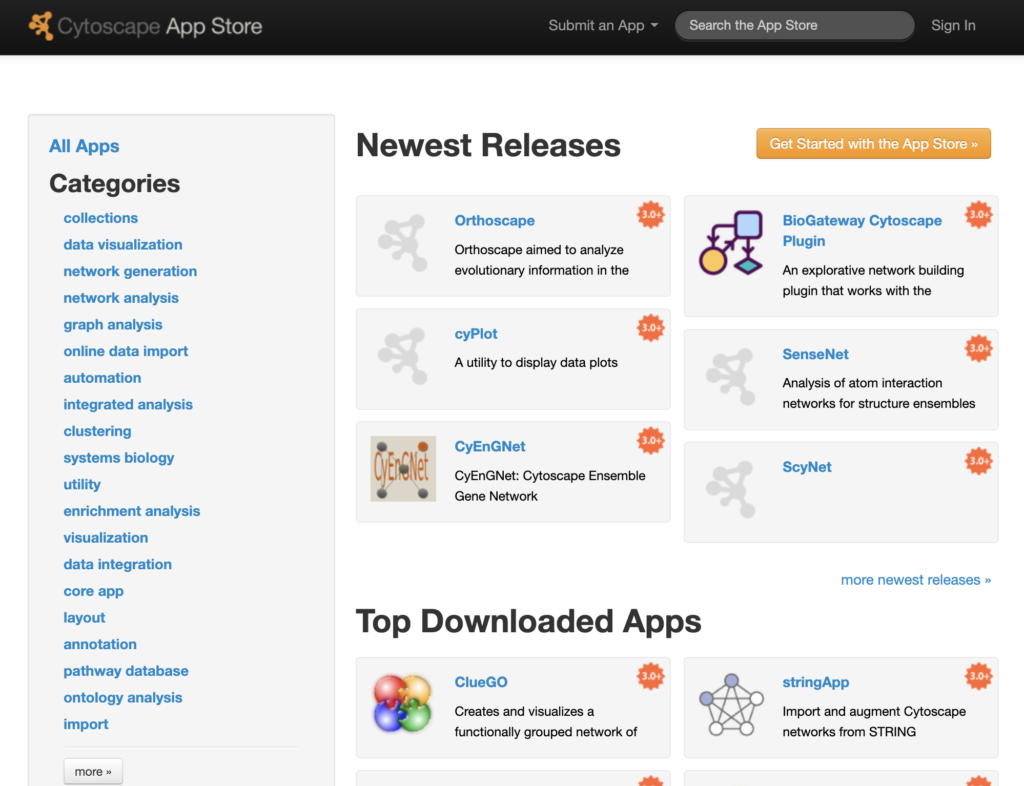
今回は遺伝子発現データの可視化、ネットワーク解析、および生物学的経路の探索を助ける代表的なプラグインを解説したいと思います。
Cytoscapeをインストールする
まずはCytoscapeをインストールしていきましょう。以下のサイトにアクセスしてダウンロードしてください。

ダウンロードが完了したらインストーラーに従ってインストールを進めてください。
アプリ一覧にCytoscapeが表示されるようになったと思いますので、クリックして起動してください。

これでCytoscapeを使う準備は完了です。
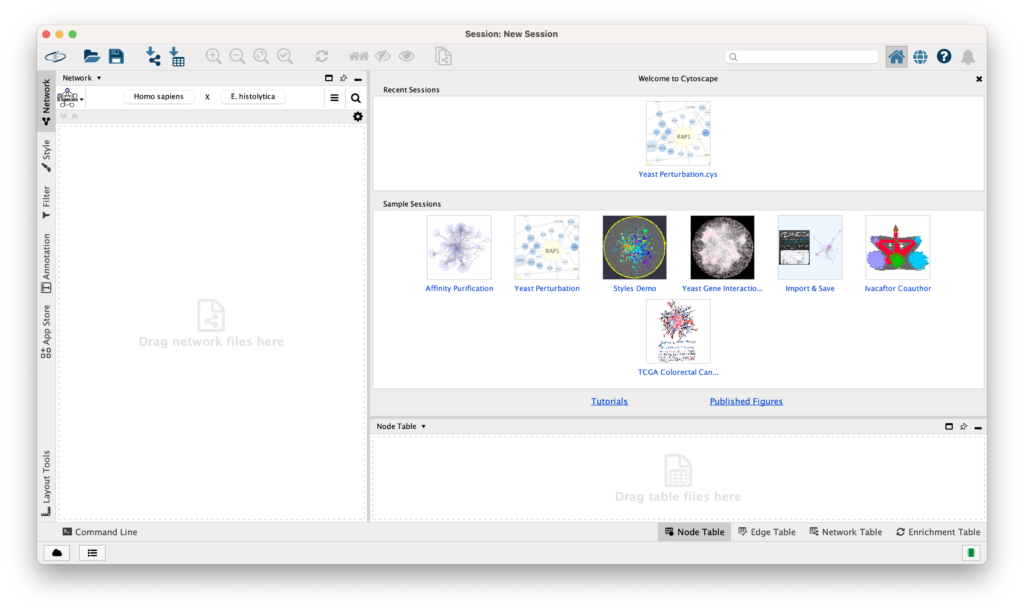
Cytoscapeの画面について
Cytoscapeの画面は以下のように構成されています。

各種ボタン・バー・パネル・ウィンドウを解説していきます。
- ツールバー:
- よく使う機能のアイコンが並んでいます。マウスカーソルをアイコンに合わせると、ツールチップが表示され説明を読むことができます。右クリックでカスタマイズも可能です。
- ネットワークパネル:
- コントロールパネルの一部で、ネットワーク検索バーがあります。これは外部リソースへの直接アクセスを可能にします。
- メインのネットワークビューウィンドウ:
- ネットワークが表示される部分です。ウィンドウ下部にはネットワークビューのためのツールセットがあります。
- テーブルパネル:
- 右下のパネルで、ノードやエッジ、ネットワーク全体に関連するデータの列を表示し、データ値を変更することができます。
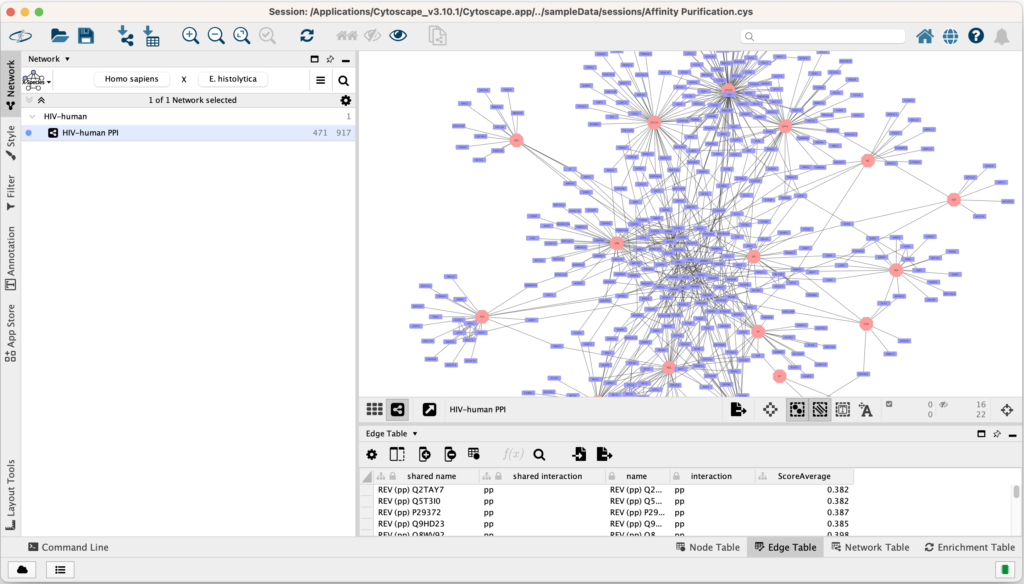
試しにネットワークビューウィンドウにある「Sample Sessons」のネットワークをどれか開いてみましょう。以下のようにウィンドウがネットワーク図に切り替わることが確認できると思います。
Cytoscapeにプラグインを読み込ませる
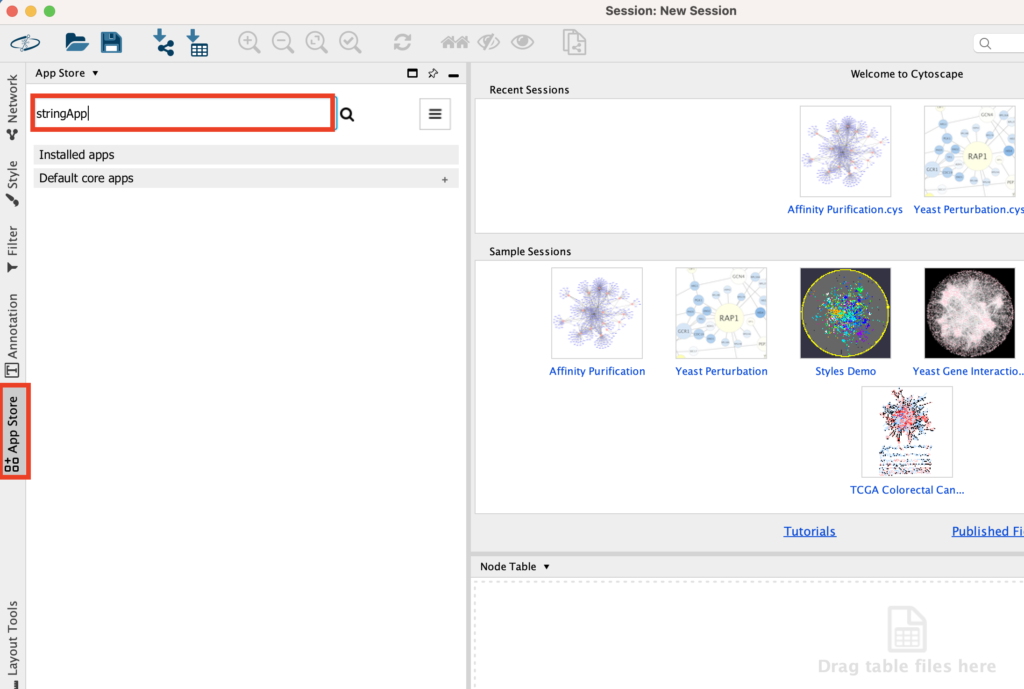
解析を始める前に使用するプラグインを読み込ませる必要があります。プラグインを入れる練習をしましょう。ツールバーのApp Storeをクリックしてください。こちらはプラグインを管理するところになります。Cytoscapeの上ではプラグインはAppと表記されるのでご注意ください。Appを検索できるバーが表示されるので、「stringApp」と打ち込んでください。
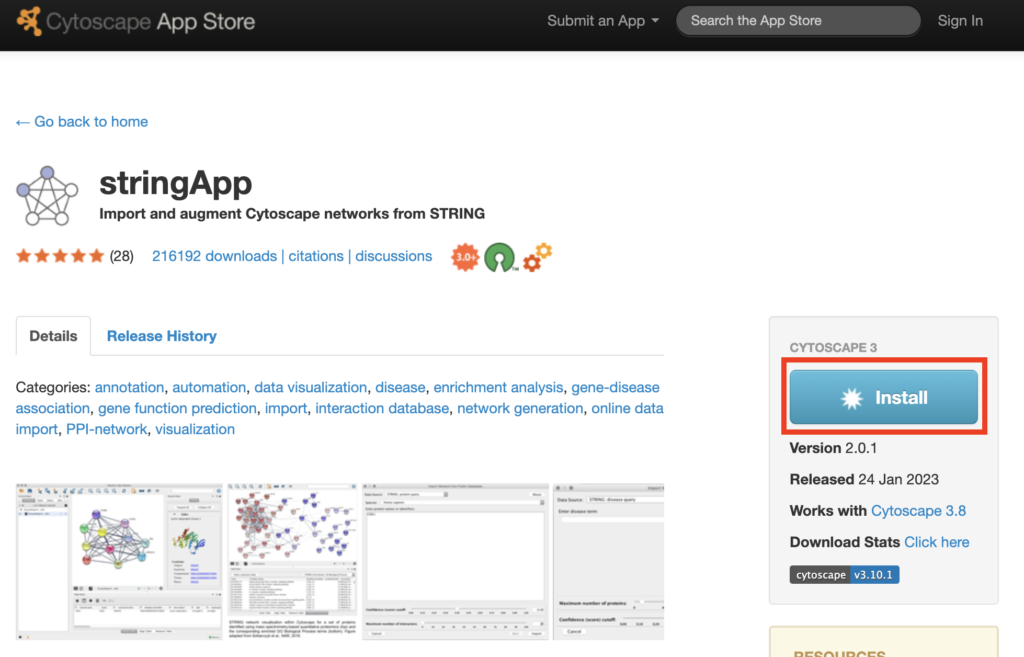
すると自動でAppStoreに遷移します。ここで、stringAppを選びましょう。

右のInstallを押してインストールをしていきましょう。インストールが完了するとボタンが「Installed」になります。
Cytoscapeの画面でstringAppがインストールされたことを確認してください。これでインストールは完了です。
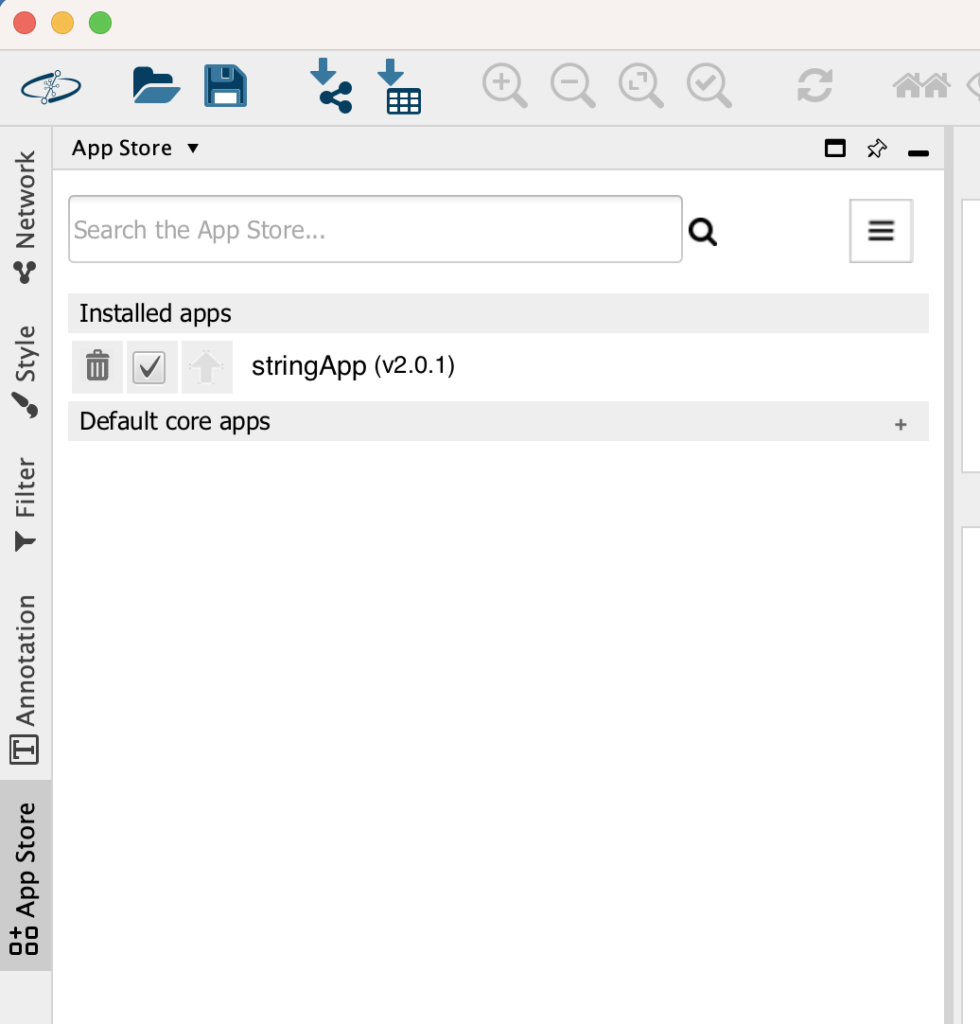
同様の手順でMCODEもダウンロードしてみてください。
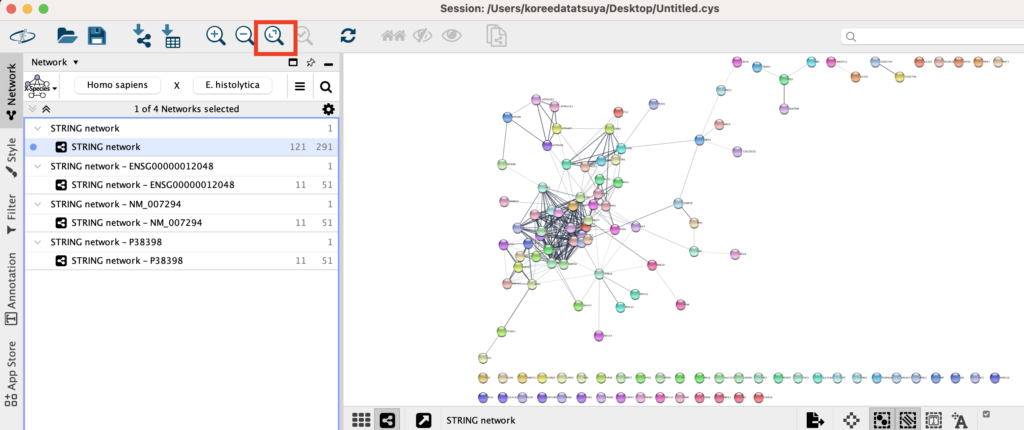
Cytoscapeでのネットワークの解析・操作方法
ネットワークの解析・操作方法については色々とあります。
左上の赤枠の虫眼鏡マークをクリックすると、ネットワーク全体を表示させます。ズームイン、ズームアウトで触った後はここをクリックして戻すことができます。
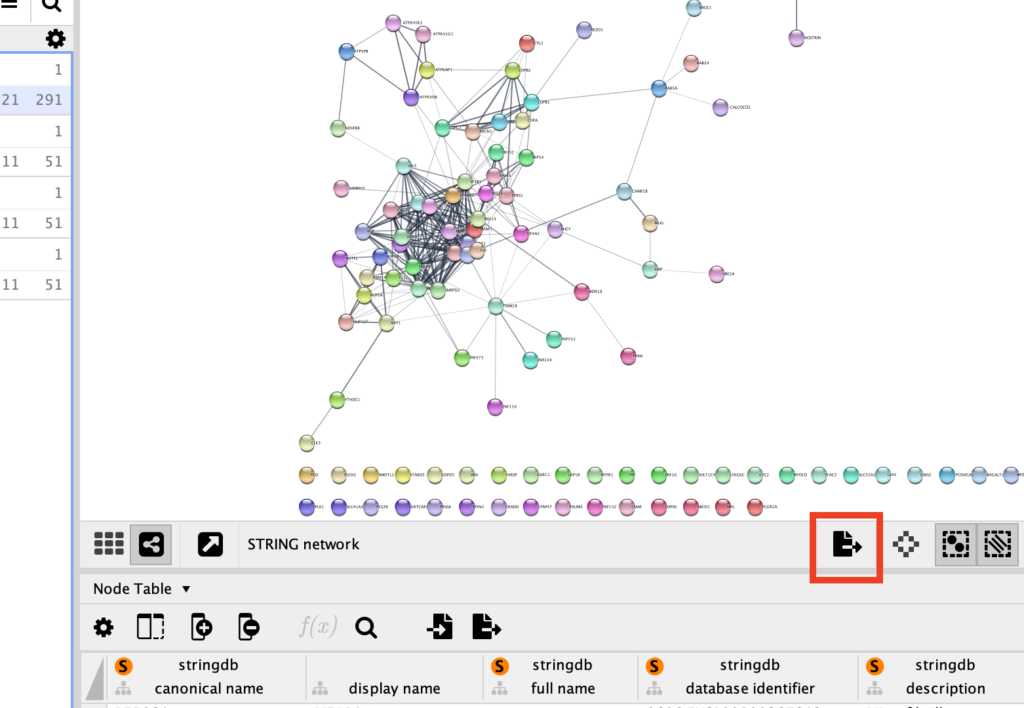
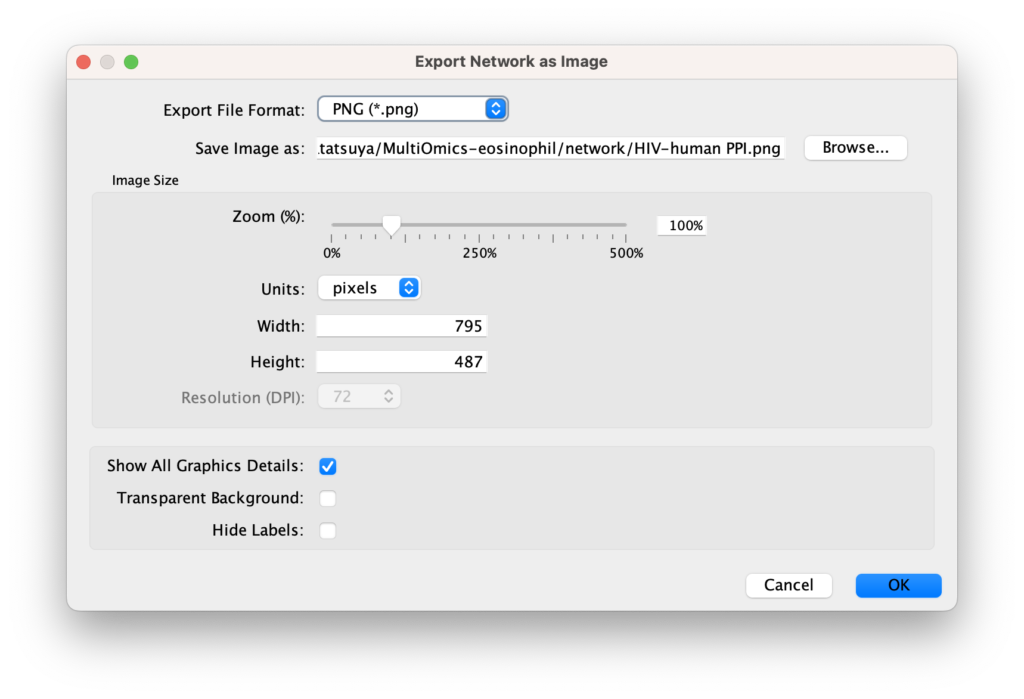
ネットワーク図を保存するときは、ファイルマークをクリックします。「Export as Image」を選ぶと画像として保存に進みます。
Cytoscapeでネットワークのnodeにfilterをかける
ネットワークを見てみるとエッジが無いnodeが下の方に表示されているかと思います。これらは実際にfigureとして保存するときに見づらくなりますので、非表示にしたいと思います。このときに便利なのがツールバーにあるFilter機能です。
Filterから、「Degree Filter」を選んでください。
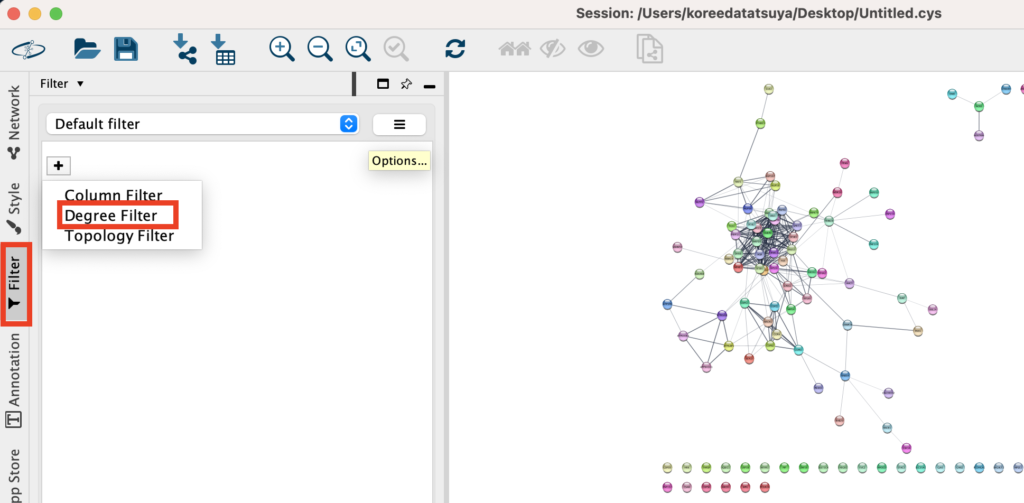
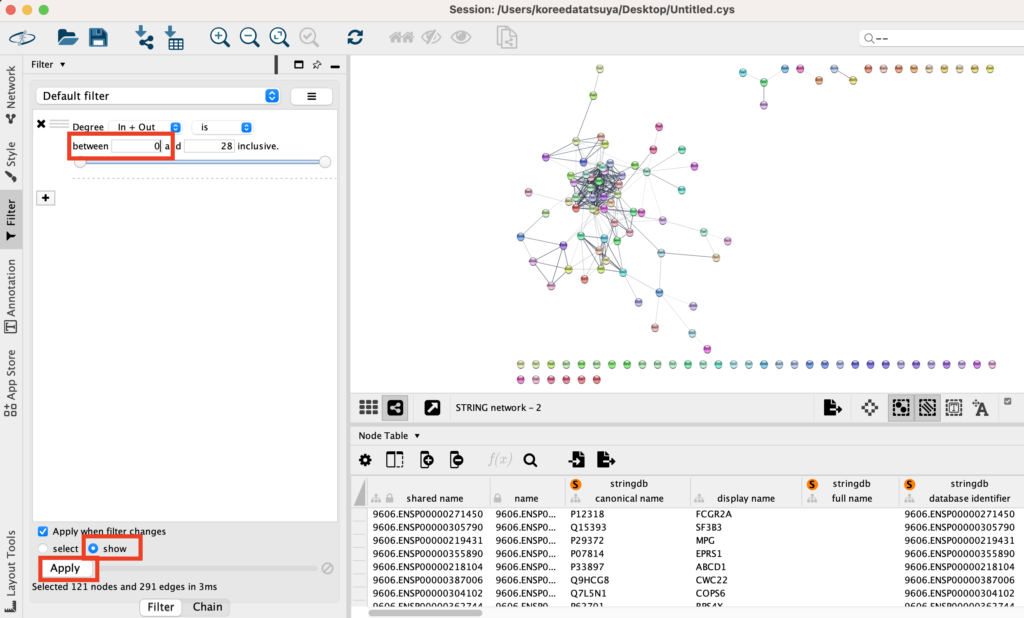
betweenを0から1に変更し、下で「show」を選んだあとにApplyをします。
Cytoscapeで「between」という用語は、しばしば「betweenness centrality(ビトゥイーンネス中心性)」と関連しています。これはネットワーク分析の概念の一つで、ノードがネットワーク内の他のノード間の通信にどの程度影響を及ぼすかを定量化する指標です。具体的には、特定のノードが他のノードペア間の最短経路上にどれだけ頻繁に現れるかを測定します。
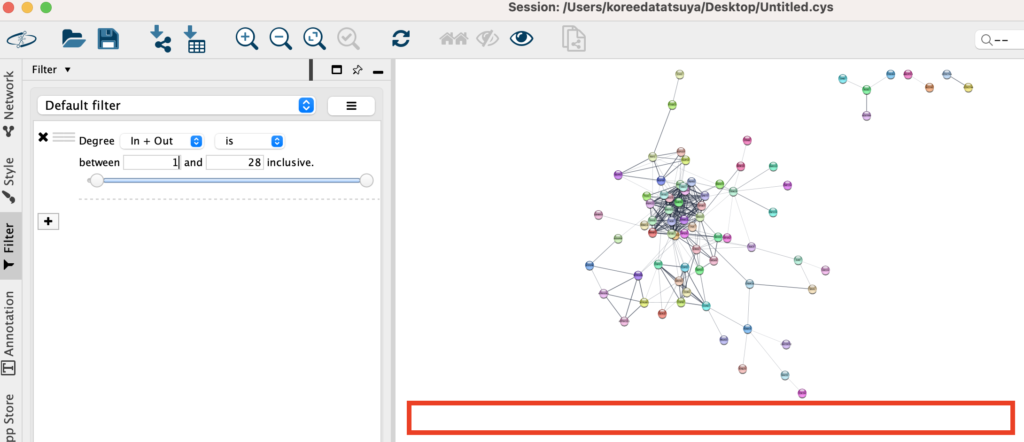
するとedgeが0のnodeが非表示になったのが確認できます。
CytoscapeでネットワークのStyle調整
「Layout」をクリックすると様々なレイアウトを確認することができます。
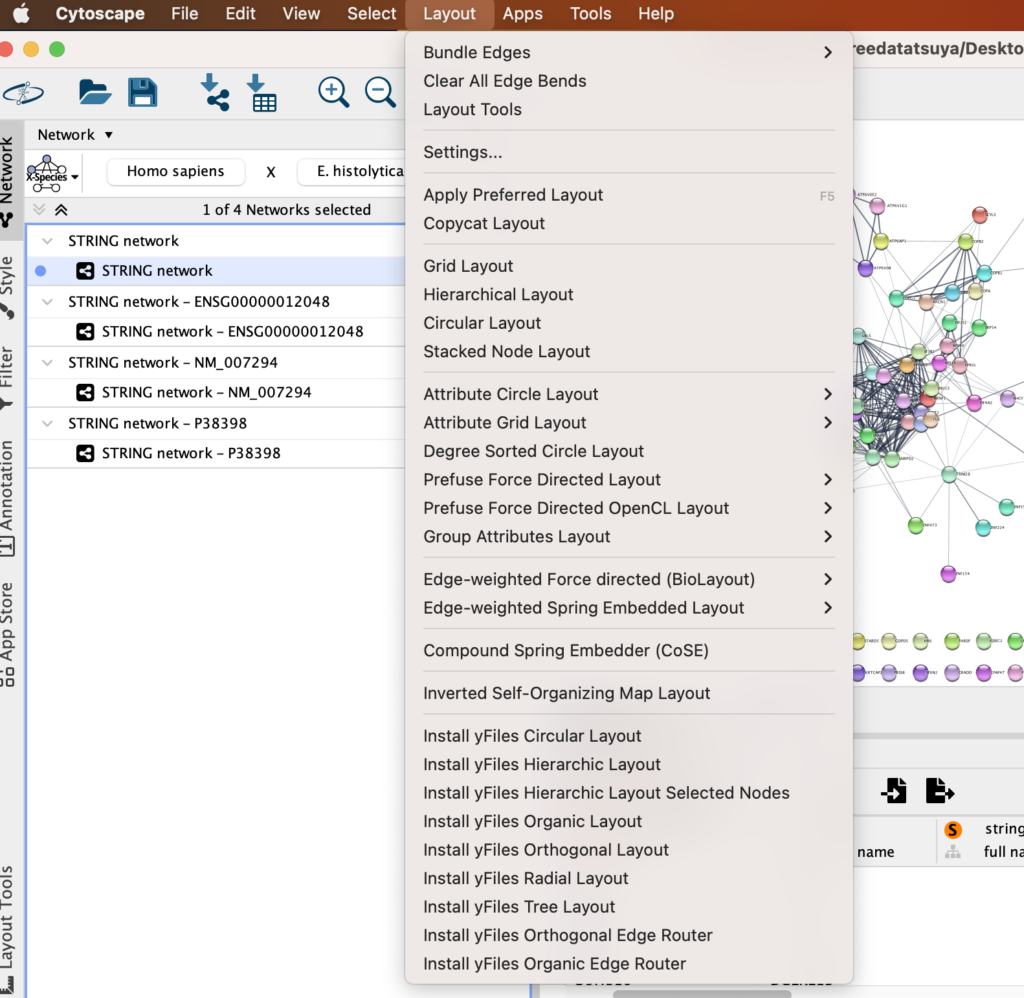
Degree Sorted Circle Layout:
もっともエッジが多いノードが円の最底辺に配置され、反時計周りにエッジの数が少なくなっていくレイアウト。

Prefuse Force Directed Layout > Score:
可能な限り、エッジやノードが重ならないように描画するレイアウト

この他にも色々ありますので、実際に自分で描画してみて試してみてください。
最後に
いかがだったでしょうか。こちらの記事は一旦ここまでにしたいと思います。次回はHub遺伝子の同定やMCODEの使い方などを解説していきますので続編も是非ご閲覧ください!

公共データを用いたSingle Cell RNA-seq解析に関する初心者向け技術書を販売中
¥3,600 → ¥1,800 今なら50%OFF!!
プログラミング初心者でも始められるわかりやすい解説!
RとSeuratで始めるSingle Cell RNA-seq解析!



