前回の記事で、公共データを用いて最速でRNA−seq解析を行う方法について書きました。その際に、ikraというツールを使い、output.tsvファイルを出すことができたと思います。しかしながら、output.tsvファイルは各遺伝子のリード数が定量されているのみで、「それで…こっから何がわかるの…?」状態になってしまってるかと思います。
ここでは、RNA-seq解析によって得られた定量データを解析するためのツールとしてiDEPを紹介したいと思います。
定量データを解析するためのツールを学ぶことによって、ただの数値データであったoutput.tsvファイルが、論文に載っているようなグラフィカルな図へと変身します。そのまま論文に掲載することも可能です。是非チャレンジしてみましょう。
本記事ではikraによってすでにoutput.tsv(TPMデータ)が得られた前提で解説します。まだoutput.tsvが得られてないよという方は、先に【バイオインフォマティクス】公共データを用いて最速でRNA−seq解析を行う方法 【ikra】に取り組んでください。
macOS Monterey(12.4), クアッドコアIntel Core i7, メモリ32GB

公共データを用いたRNA-seq解析に関する初心者向け技術書を販売中
¥2,500 → ¥1,500 今なら40%OFF!!
画面キャプチャをふんだんに掲載したわかりやすい解説!
自宅PCからドライ研究が始められます
iDEP
iDEPは、ウェブブラウザ上でRNA-seqデータ解析を行うことができるウェブツールです。1つのwebサイトで以下の解析が一括でできる優れものです!
- Heatmap
- k-means
- PCA
- DEG解析
- Volcano Plot
- MA Plot
- Scatter Plot
- GO term解析
- Pathway解析
- KEGG
- Genome view
- Bicluster
- Network解析
- RNA-seq解析
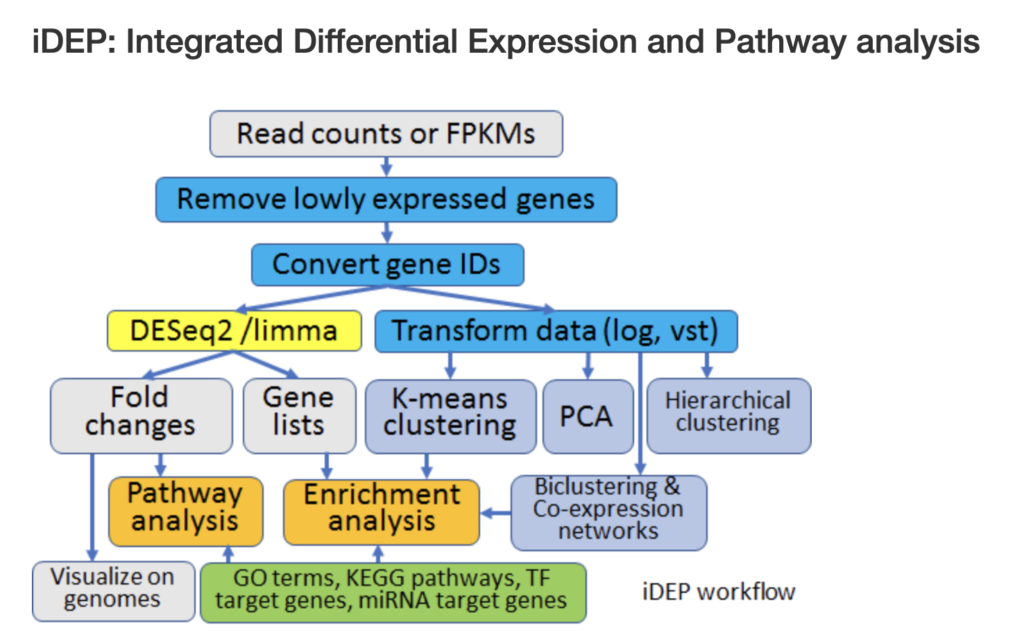
実はikraによって作成されたoutput.tsvはiDEPで解析するために作られています。そのため、output.tsvさえ用意できてしまえば、iDEPを使うのは難しくありません。実際に使ってみましょう。
iDEPへデータをアップする方法
Upload expression data(CSV or text)にoutput.tsvをuploadします。
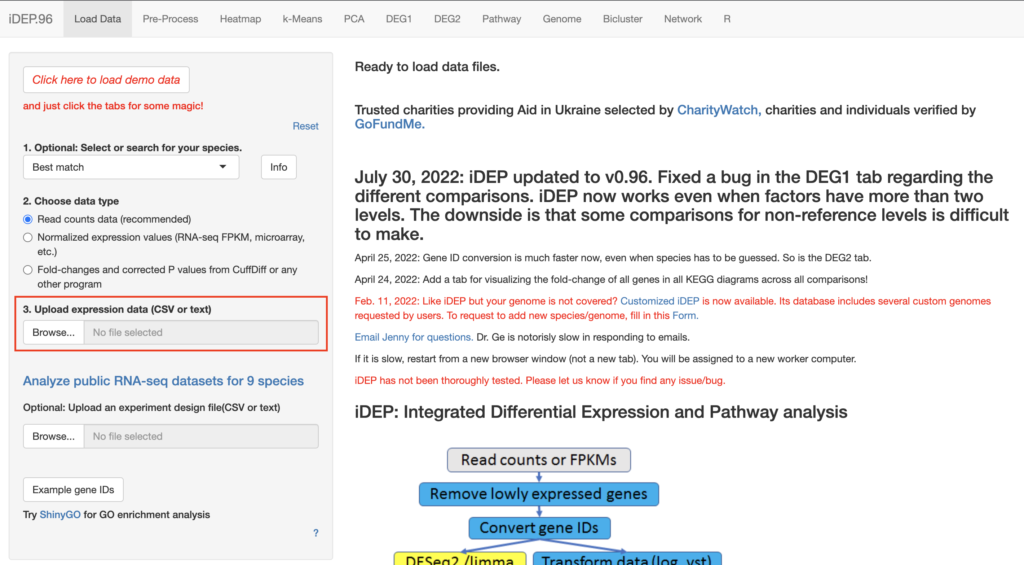
1. Optional: Select or seach for your species.で自分の解析した生物種、2. Choose data typeで「Read counts data (recommended)」を選びます
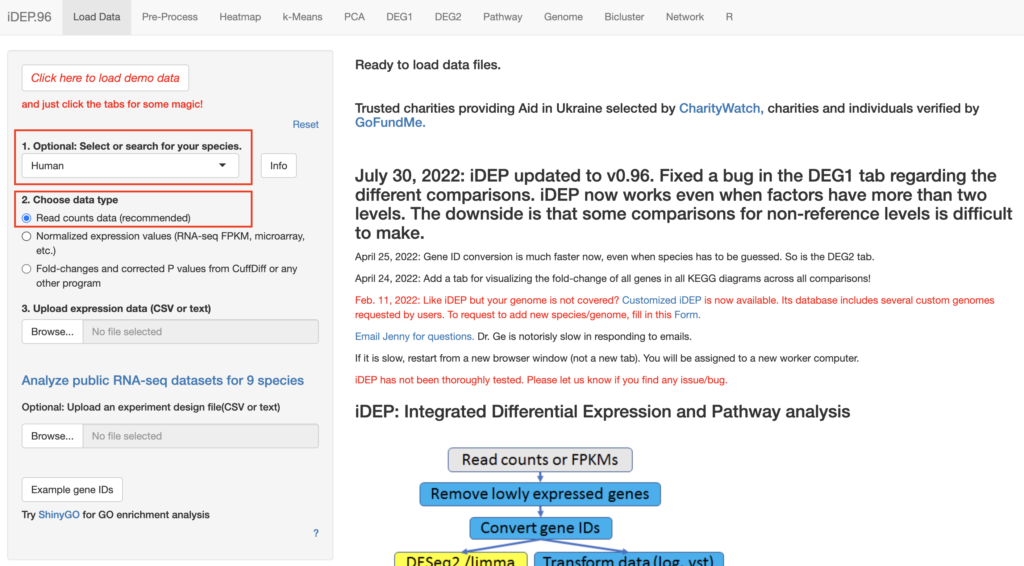
少し待つと、ファイルがuploadされます。右側の画面が切り替わったら成功です!実際に上タブから好きな解析結果を見てみましょう。
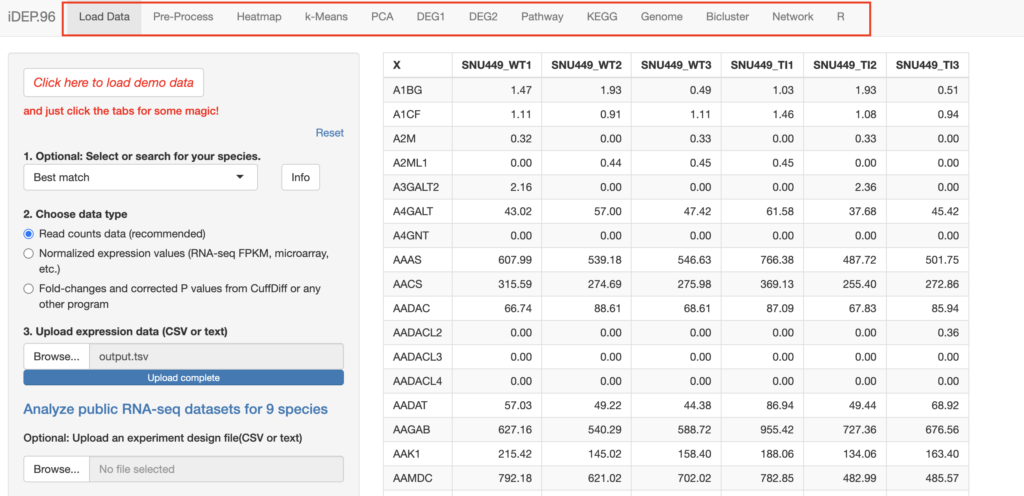
これでデータのアップロードは完了です。今回はHeatmapとPCA解析、DEG解析について更に紹介したいと思います。
Heatmap
Heatmapは縦軸に遺伝子名、横軸にサンプルをとって、遺伝子ごとの発現量を色で示したグラフになります。遺伝子は発現量に応じてクラスタリングされるようになっています。
左のスライドバーから発現変動が見られる遺伝子数を変更できます。
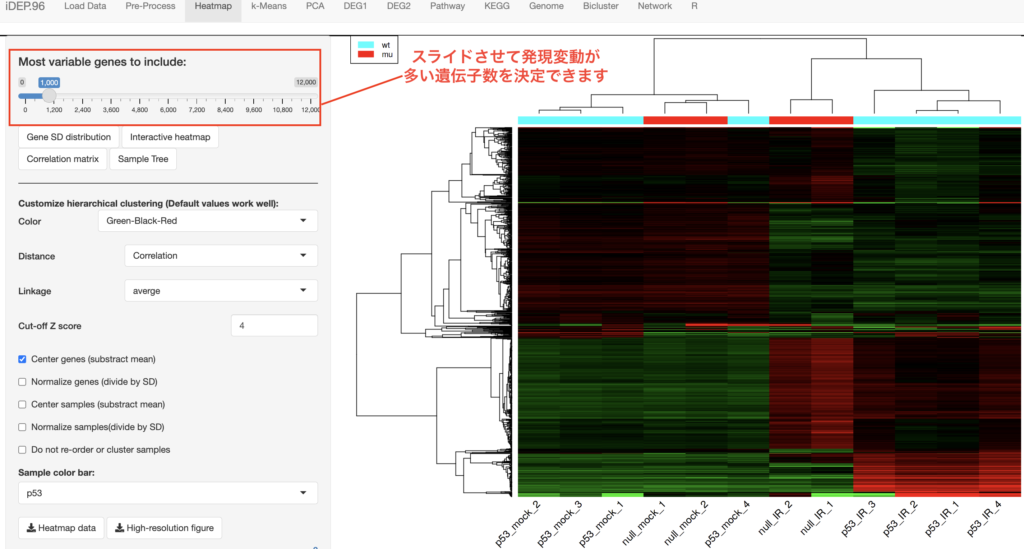
また、色の変更やクラスタリングの計算方法を変更できます。

PCA
PCA分析は主成分分析とも呼ばれ、各サンプルの類似度をプロット図で確認することができます。
バイオインフォマティクスの分野以外にもよく使用される類似度確認方法の一つです。
左サイドバーをPrincipal Component Analysisに合わせます。
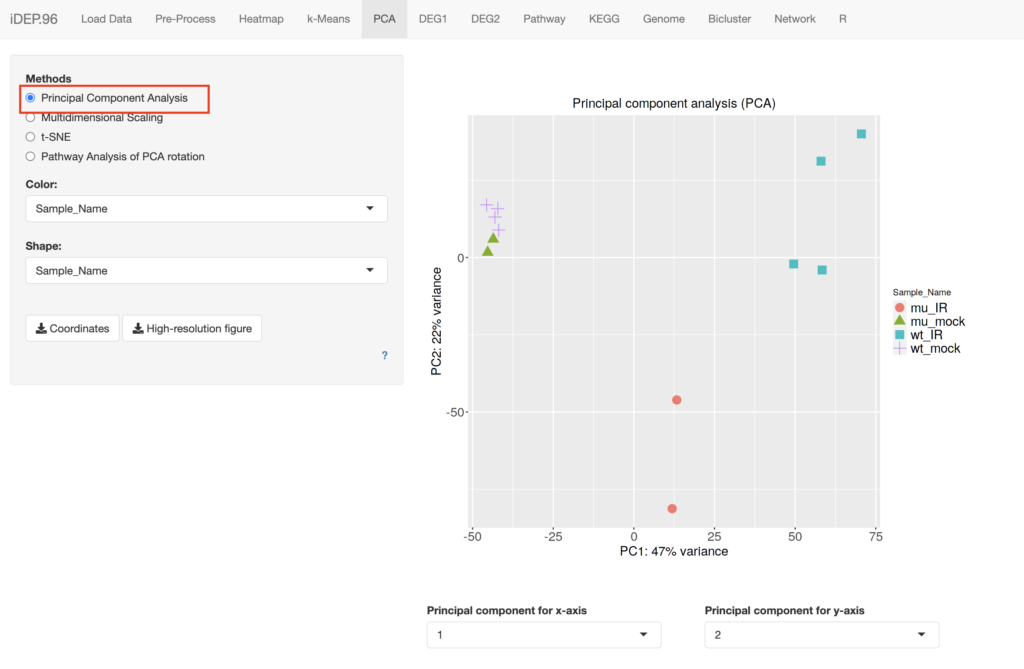
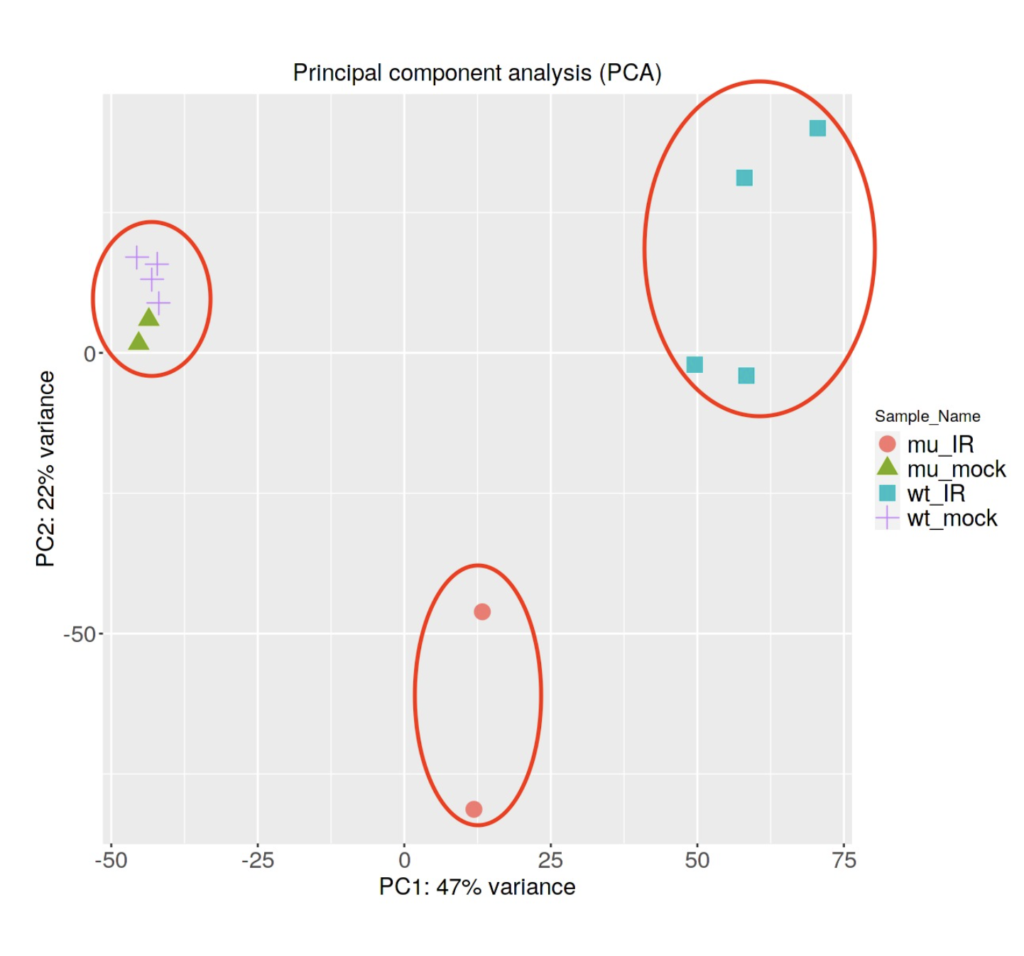
以下の図だと、実験のサンプルごとに近い位置にプロットがされているのが確認できます。またwt_mockとmu_mockはその他クラスターに比べて近い位置にあり、性質が近いサンプルである可能性が考察できます。
DEG1
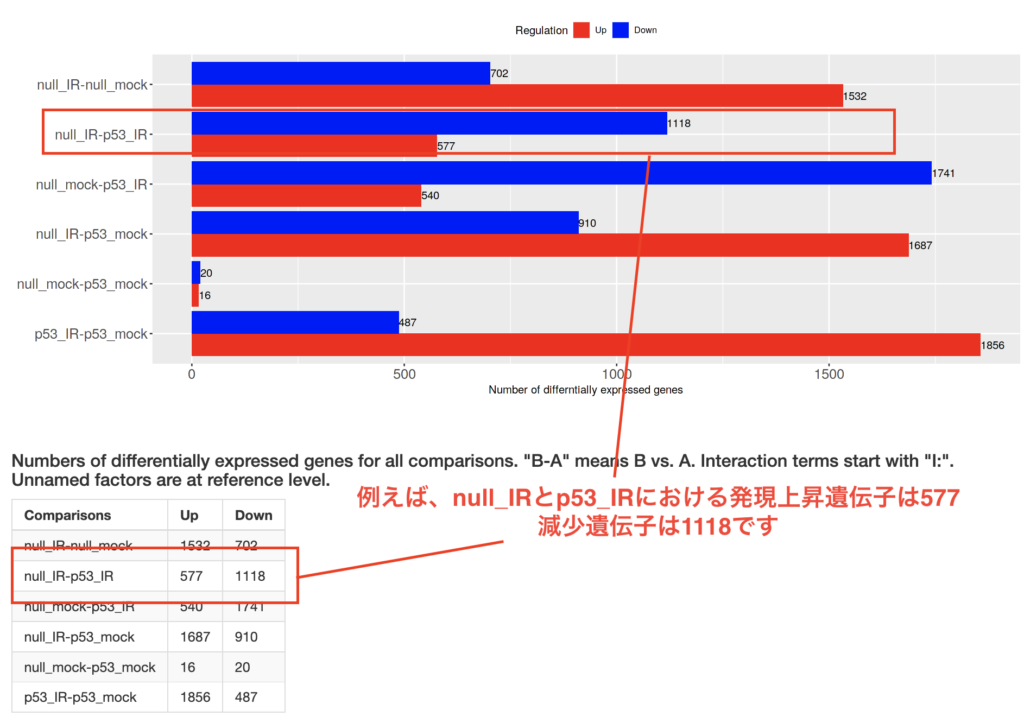
DEG1では各サンプル間を比較して発現変動が見られた遺伝子(DEG)を可視化させています。処理にした前と後のサンプルでup regulationしたか、down regulationしたか、または変動なしかかを確認するシンプルな解析です。トランスオミクス解析と聞くと皆さん、一番最初にDEG解析が思い浮かぶのではないでしょうか。
左サイドバーのMethod:は「DESeq2」に合わせてください。
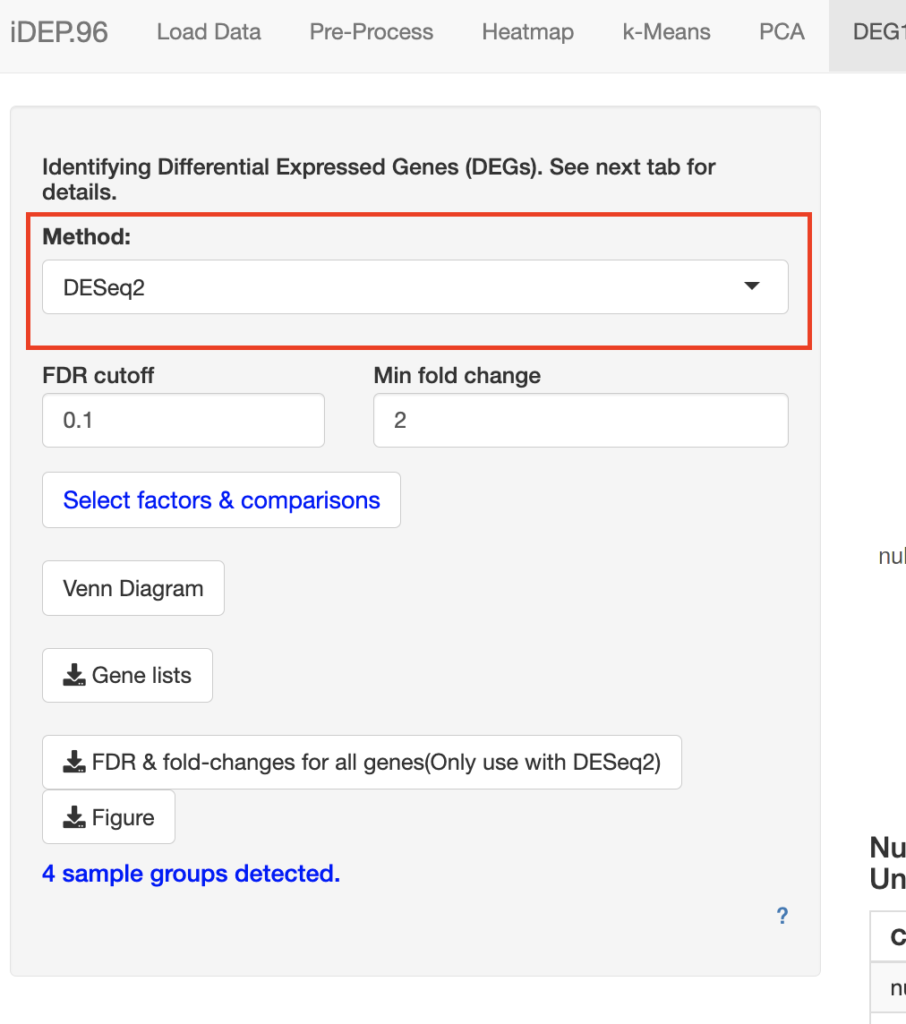
以下の図の例で行くと、例えば、null_IRとp53_IRにおける発現上昇遺伝子数は577で、減少は1118で有ることがわかります。
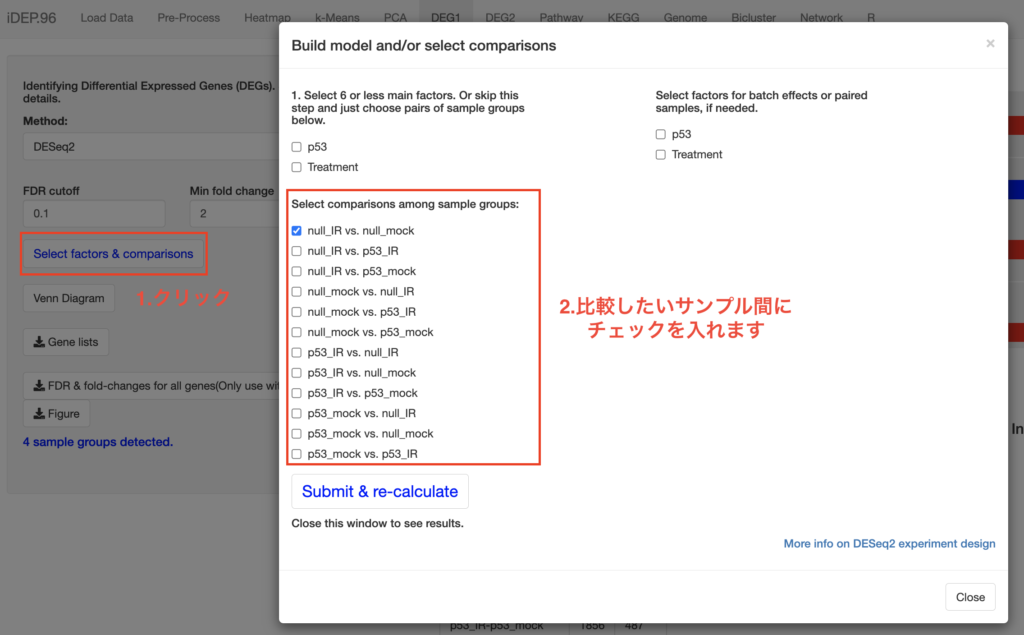
Select factors & comparisonsをクリックすると、比較したいサンプル間の選択が可能です。
Venn Diagramを使ってDEGの可視化をベン図を用いて行うことができます。
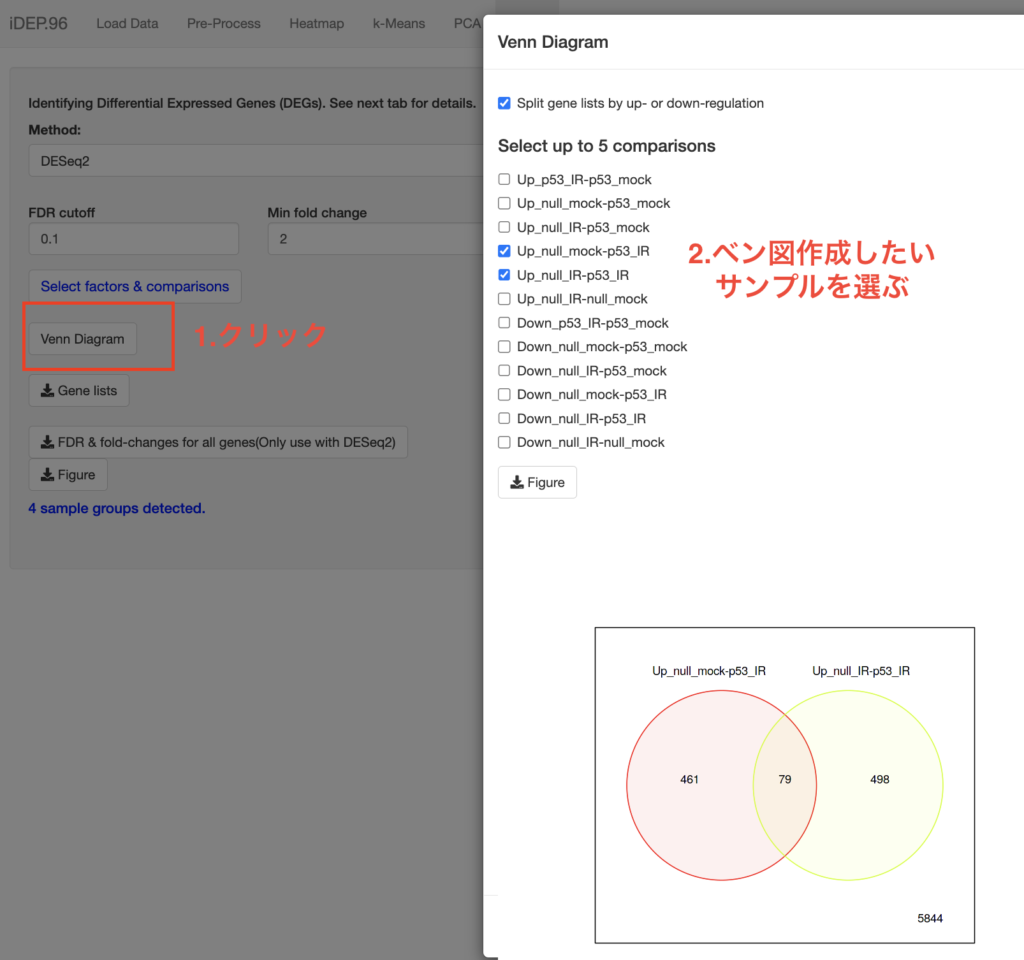
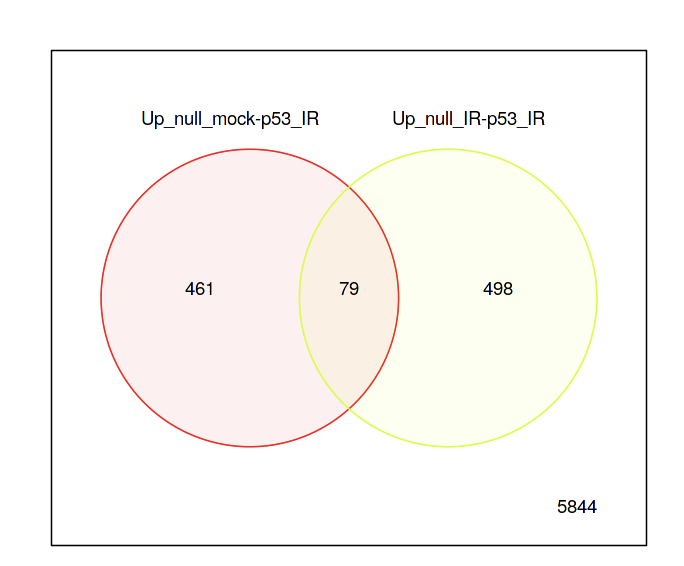
以下の例だと、Up_null_mock-p53_IRとUp_nullIR-p53_IRのサンプル間で発現上昇が共に見られた遺伝子数は79で有ることがわかります。この79の遺伝子に注目してGo term解析などをしてみれば、似たような役割を持つ遺伝子が上昇していることが解析可能です。
最後に
いかがだったでしょうか。今回はiDEPは使いこなせると大変便利であることがご理解いただけたかと思います。ikra→iDEPの流れをマスターすれば、Rなどのプログラミングを使えなくても論文に乗せるfigureがあっという間に完成します。
今回紹介した以外にも、まだまだRNA−seq解析結果を可視化させる手法はたくさんあります。RNA−seq解析結果を可視化の第二弾を作成予定ですので、楽しみにお待ちただけたら幸いです。

公共データを用いたRNA-seq解析に関する初心者向け技術書を販売中
¥2,500 → ¥1,500 今なら40%OFF!!
画面キャプチャをふんだんに掲載したわかりやすい解説!
自宅PCからドライ研究が始められます



