本記事は低分子-タンパク質ドッキングソフトウェアであるSwissDockについて書かれた記事です。こちらの内容が理解できると、望みの化合物とタンパク質の複合体の予測ができるようになります。ぜひ皆さんもトライしてみて下さい!
ぜひトライしてみてください!
Windows 11 Home, UCSF Chimera 1.17.3
自宅でできるin silico創薬の技術書を販売中
¥3,200 → ¥1500 今なら50%OFF!!
新薬探索を試したい方必読!
ITエンジニアである著者の視点から、wetな研究者からもdryの創薬研究をわかりやすく身近に感じられるように解説しています
自宅でできるin silico創薬の技術書を販売中
¥3,200 → ¥1,500 今なら50%OFF!!
分子ドッキングやMDシミュレーションなど、
自宅でできるin silico創薬の解析方法を解説したものになります!
本記事を進むにあたって、UCSF Chimeraのダウンロードをお願いします。
分子ドッキングとは?
分子ドッキングは薬物開発において重要な手法です。これは、タンパク質などの大きな分子と小さな分子(潜在的な薬物)との相互作用をコンピュータ上でシミュレートする過程です。分子ドッキングは、薬物がターゲットとなるタンパク質にどのように結合するかを予測し、その結合の強さや特性を評価します。これにより、新しい薬物候補の効果や副作用を理解し、薬物開発の初期段階でのスクリーニングを効率化することができます。分子ドッキングは、候補となる化合物の広範なライブラリから有望な薬物を特定するのに役立ちます。
Swiss-Dockとは
Swiss-Dockは、分子ドッキングを実行するための無料でアクセス可能なウェブベースのツールです。このツールは、特にタンパク質-リガンド相互作用のモデリングに焦点を当てています。ユーザーは、ターゲットとなるタンパク質とリガンド(潜在的な薬物分子)の3D構造をアップロードし、Swiss-Dockはこれらの分子間の最適な結合モードを予測します。結果は視覚的な表現として提供され、リガンドがタンパク質に結合する場所や形状、結合の強さを評価するのに役立ちます。Swiss-Dockはその使いやすさとアクセシビリティで、研究者や学生に広く利用されています。
Swiss-Dockによる分子ドッキング
タンパク質の準備
まずタンパク質の準備を行っていきます。
こちらのブログにある新型コロナの標的タンパク質MPro(PDB:2OPQ)を使っていきます。
PDB Formatをダウンロードしてみてください。
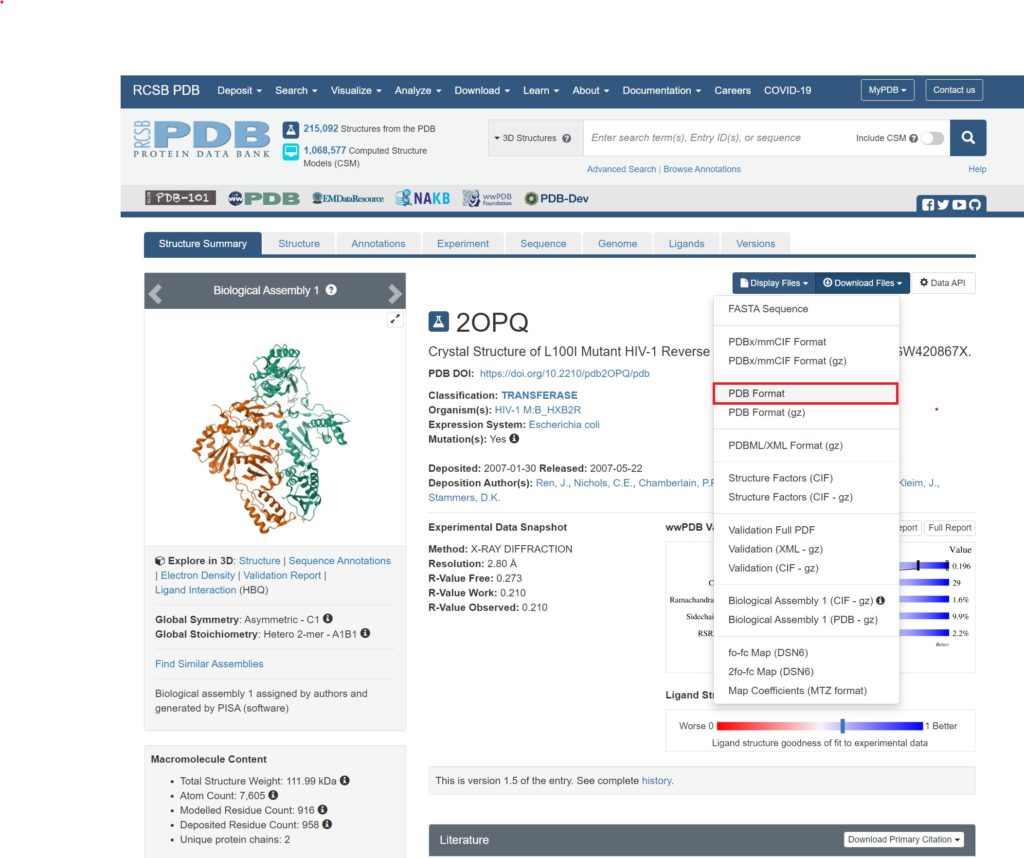
ダウンロードしたファイルをUCSF Chimeraで開いてください。以下のようになると思います。
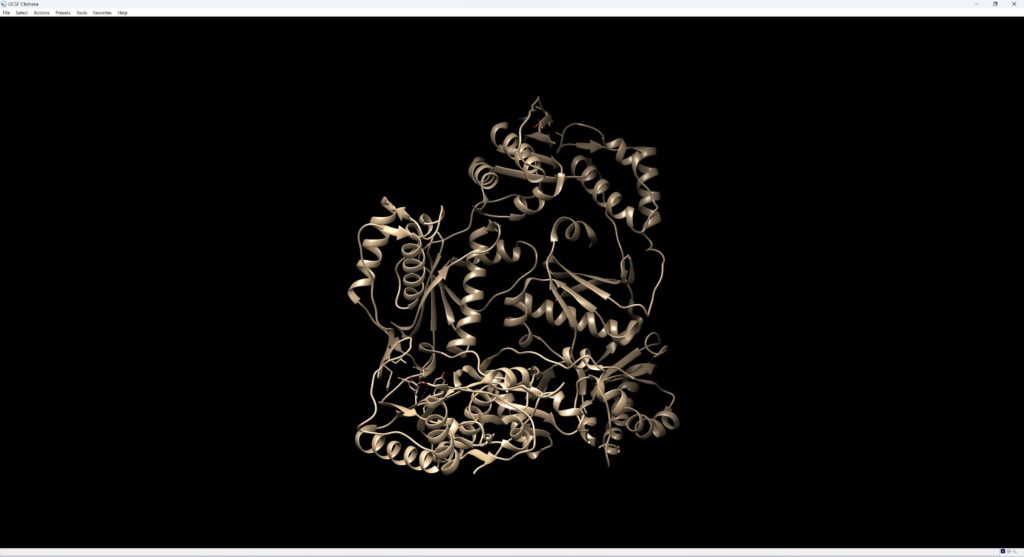
このタンパク質にはタンパク質のアミノ酸でないものも含まれているので、それらを消していきます。
左上のSelect→Residue→HBQを選択し、
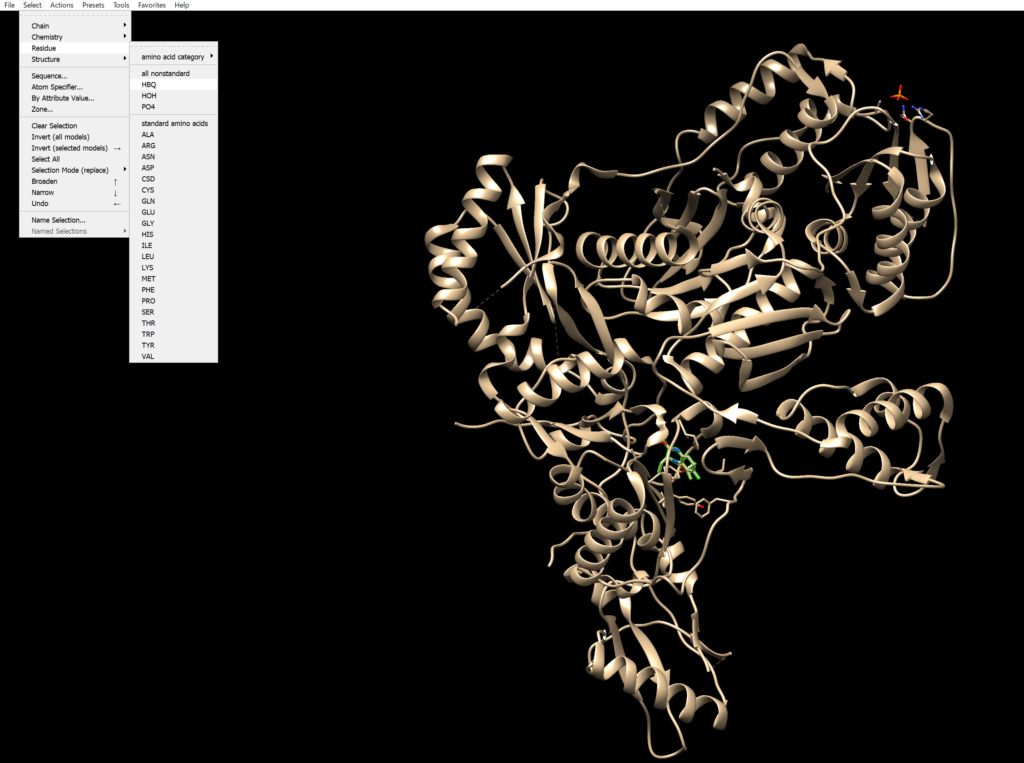
Action→DeleteでHBQを消してください。同様にHOHとPO4も消してください。
このままSWISS-DOCKで設定する分子の上限よりも少し大きいので、末端のアミノ酸を少し削ります。
Tool→Sequence→Sequenceから配列を出し、IETVPVKLKPGMDGまでを選択し、削除してください。(以下ではIETVPVKLKまで選択されています。)
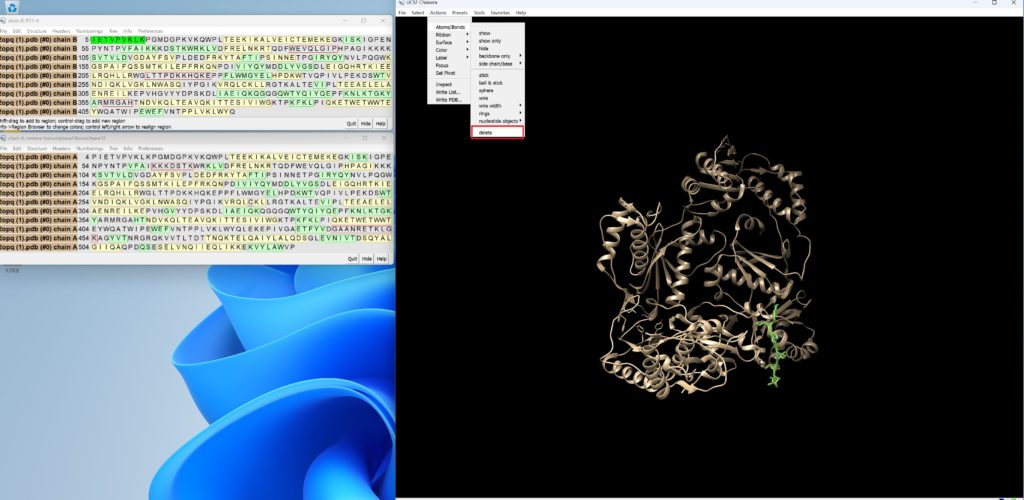
その後、左上のFile→Save PDBからこのPDBファイルを保存してください。
以上でタンパク質の準備は終了です。
リガンドの準備
それでは続いて、リガンドの準備に移っていきます。前回のスクリーニングで取れた化合物はTolnaftateです。
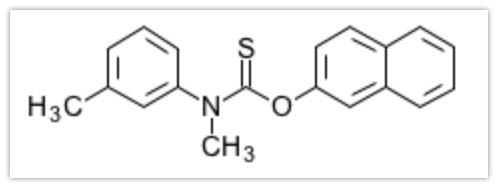
それではswiss-dock用のファイルを作成していきます。
draw molecule onlineというサイトに行き、Tolnaftateを書き、Click to Convertを押します。
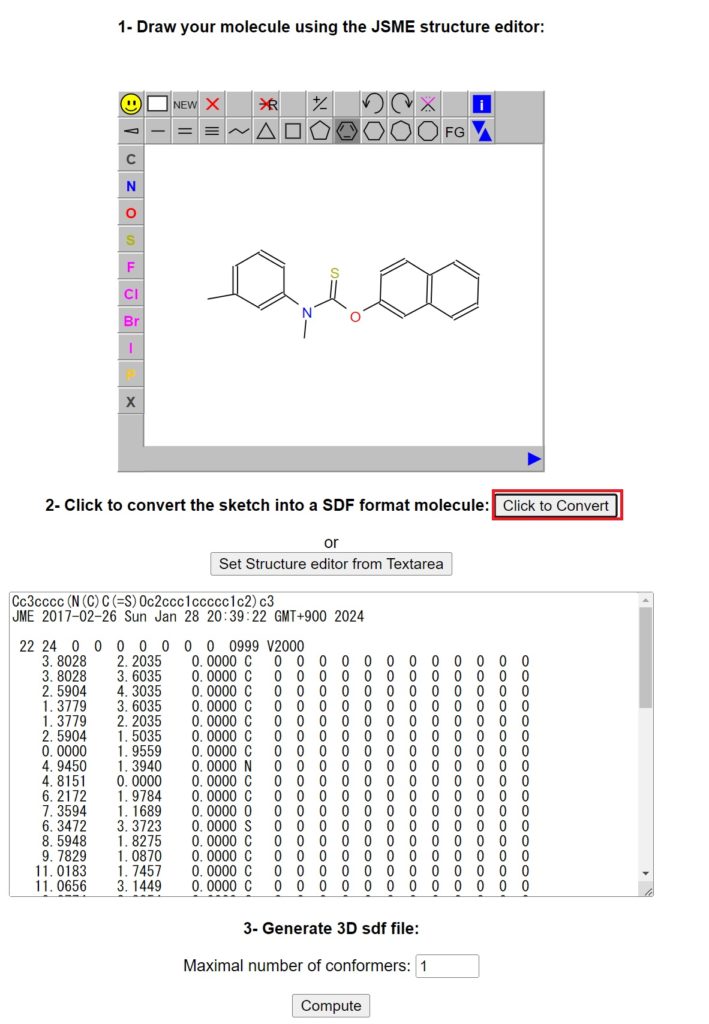
そして、Compute を押してください。
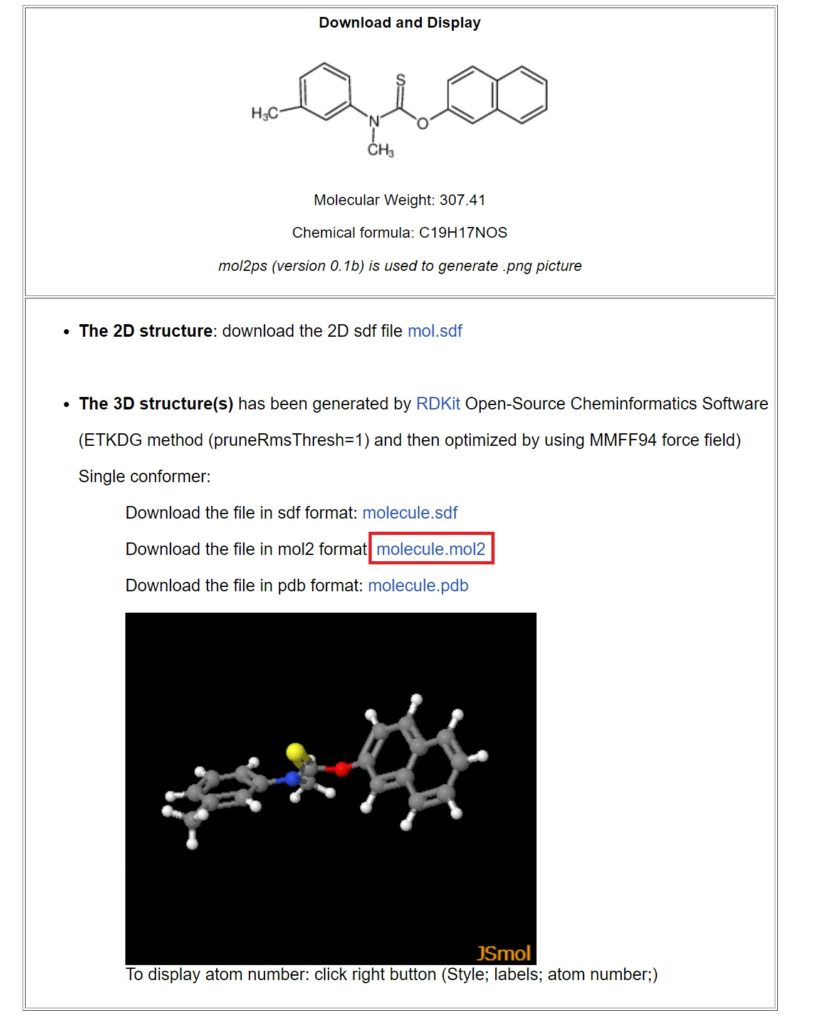
molecule.mol2 を押すと、molファイルがダウンロードされます。Tolnaftate.mol2として保存します。
これでリガンドの設定は終了です。
Swiss-Dockによる分子ドッキング
それではSwiss-Dockによる分子ドッキングを行っていきましょう。ここにアクセスすると以下の画面になると思います。
Submit Dockingタブに行き、準備したファイルのアップロードとJob Name, E-mail addressを書いてください。
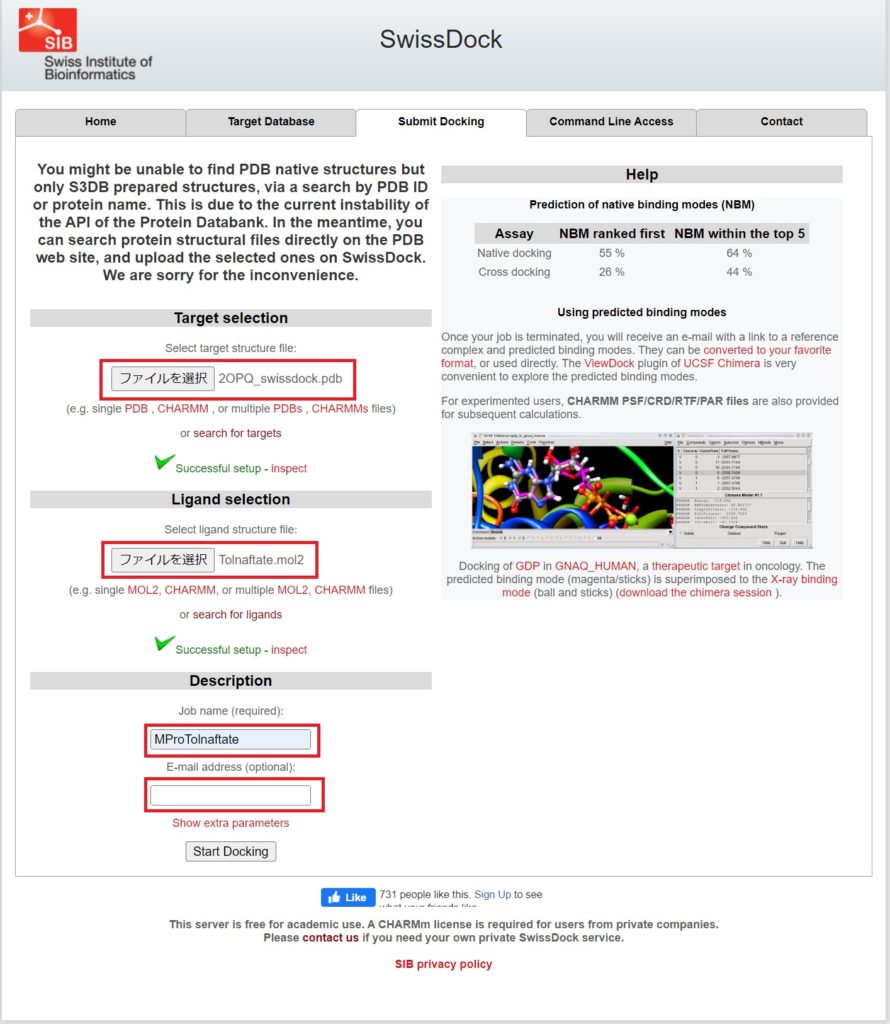
Start Dockingを押すと、分子ドッキングが開始されます。
終了するとメールが届くので、開いてみましょう。
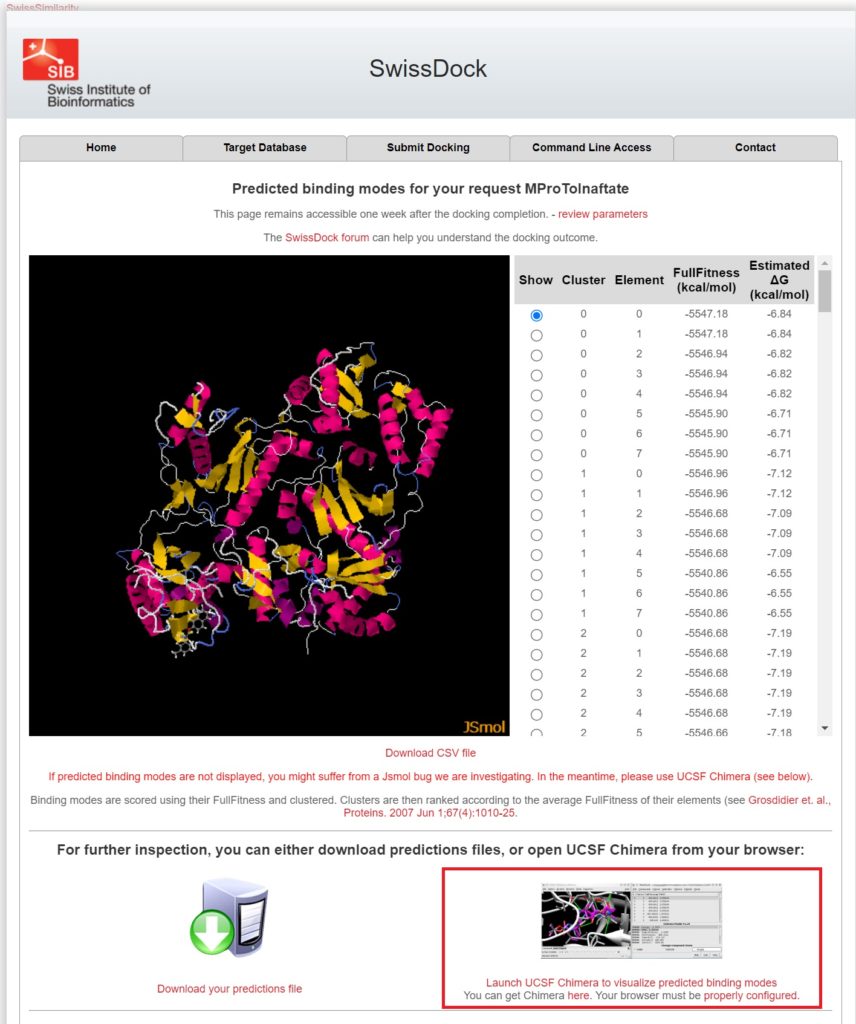
結合モードはFullFitness(kcal/mol)とEstimated ΔG(kcal/mol)という2つの値でスコアリングされ、クラスタリングされています。これらの値はドッキングの結果の質を示すためのもので、値が低いほど予測される結合が強いことを意味しています。リストには複数のクラスターとそれぞれのエネルギースコアが表示されています
さらに詳細に解析するために、右下のLaunch UCSF Chimera to visualize predicted binding modes
をクリックして、UCSF Chimeraで開いてみてください。
UCFS Chimeraによる解析
開くと以下のような画面になっているかと思います。
各結合サイトと情報を細かく見えます。
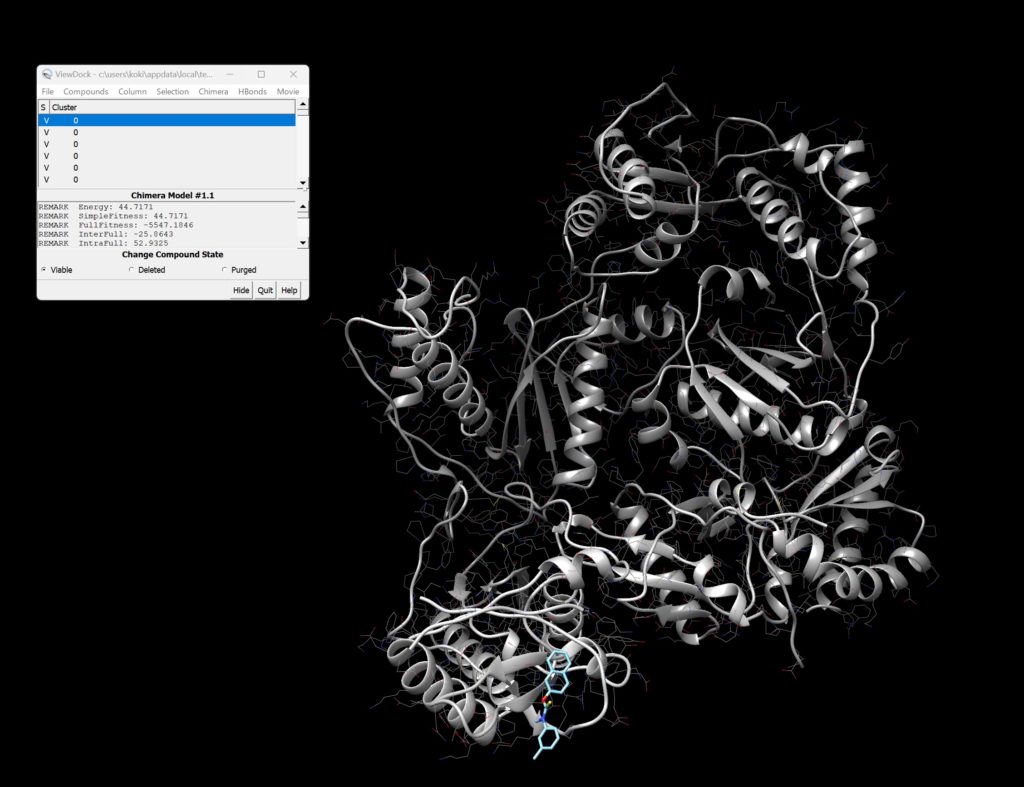
さらに右上のMovie→Playを押すと、結合様式一覧がMovieでみれます。
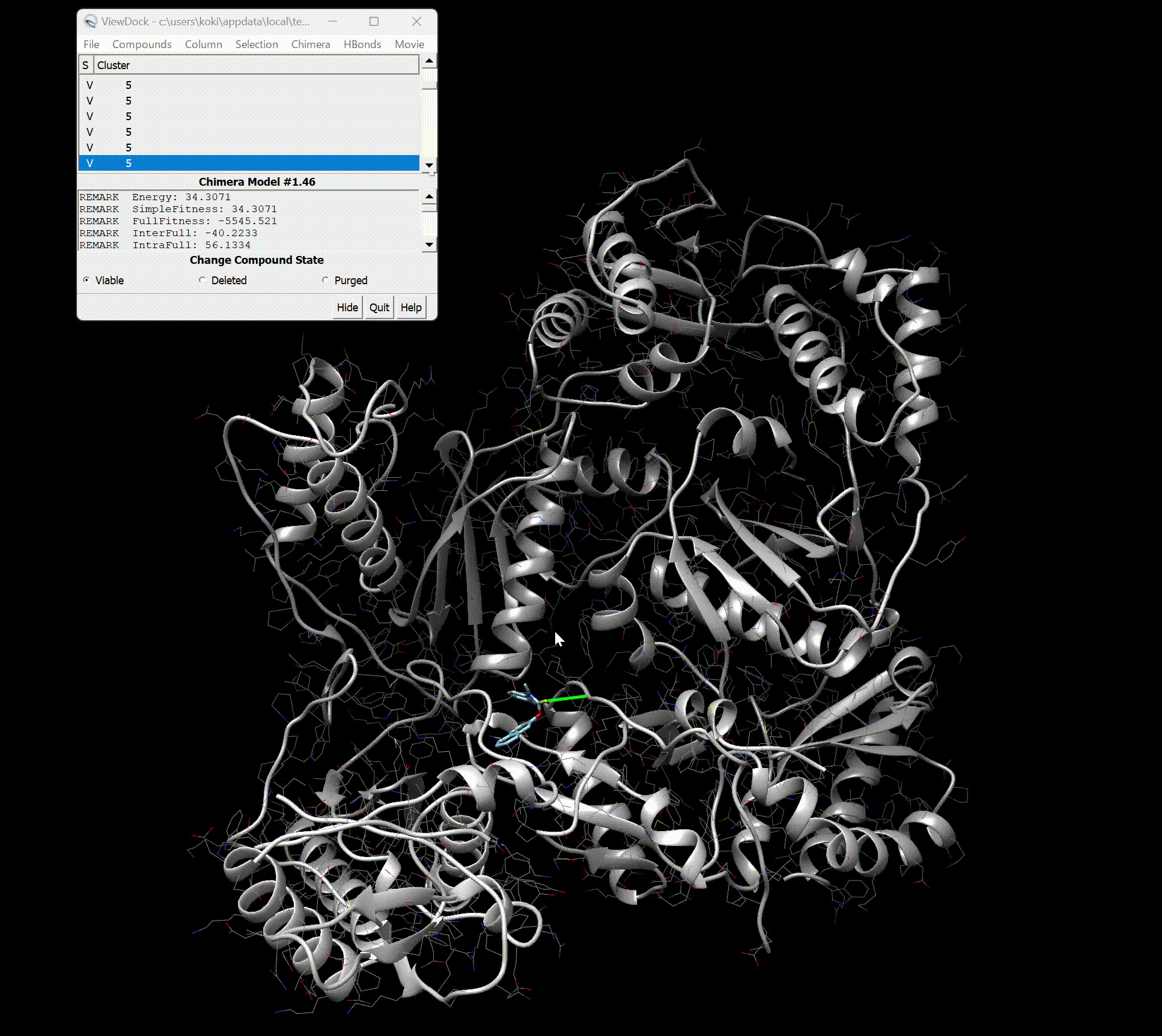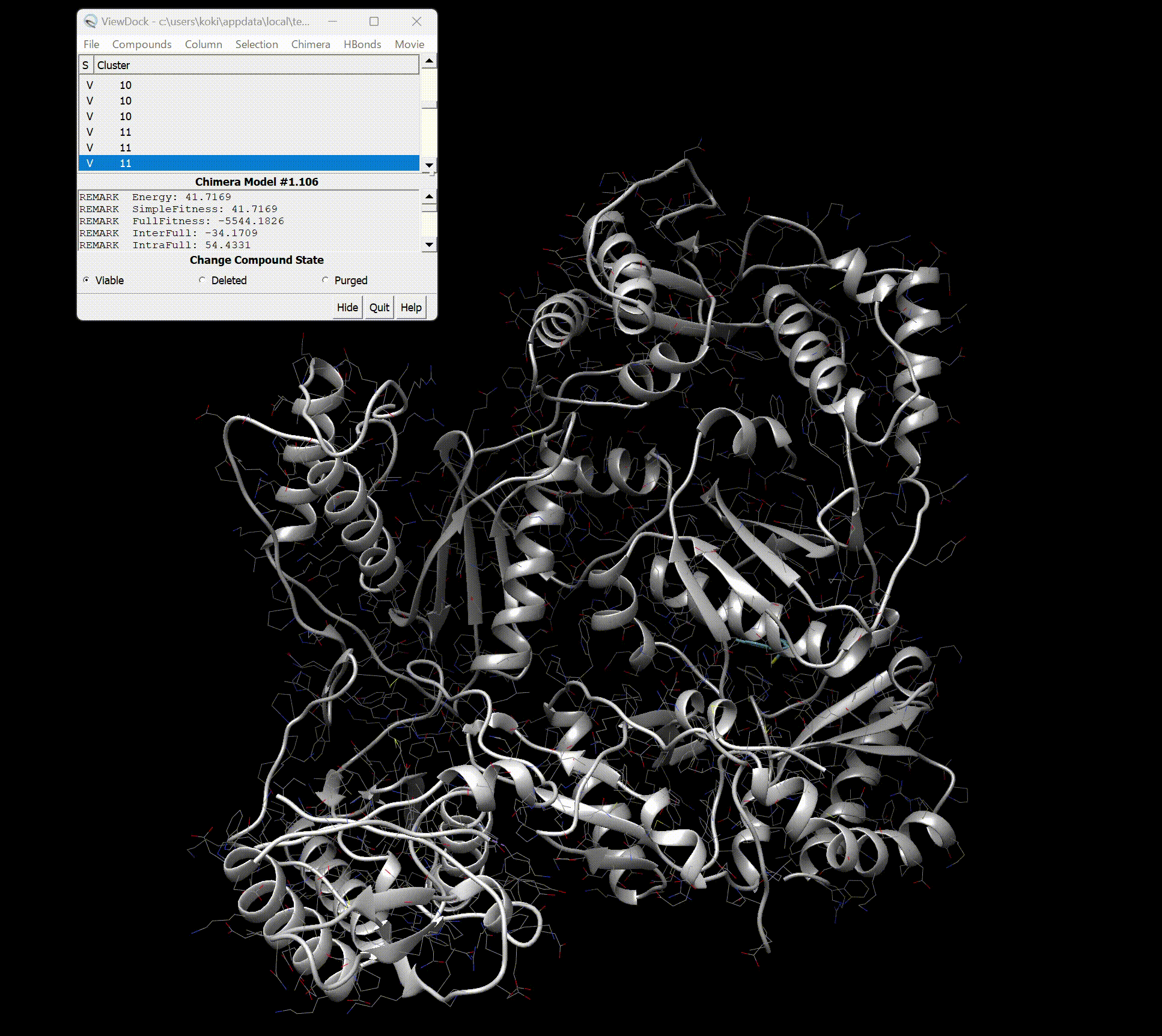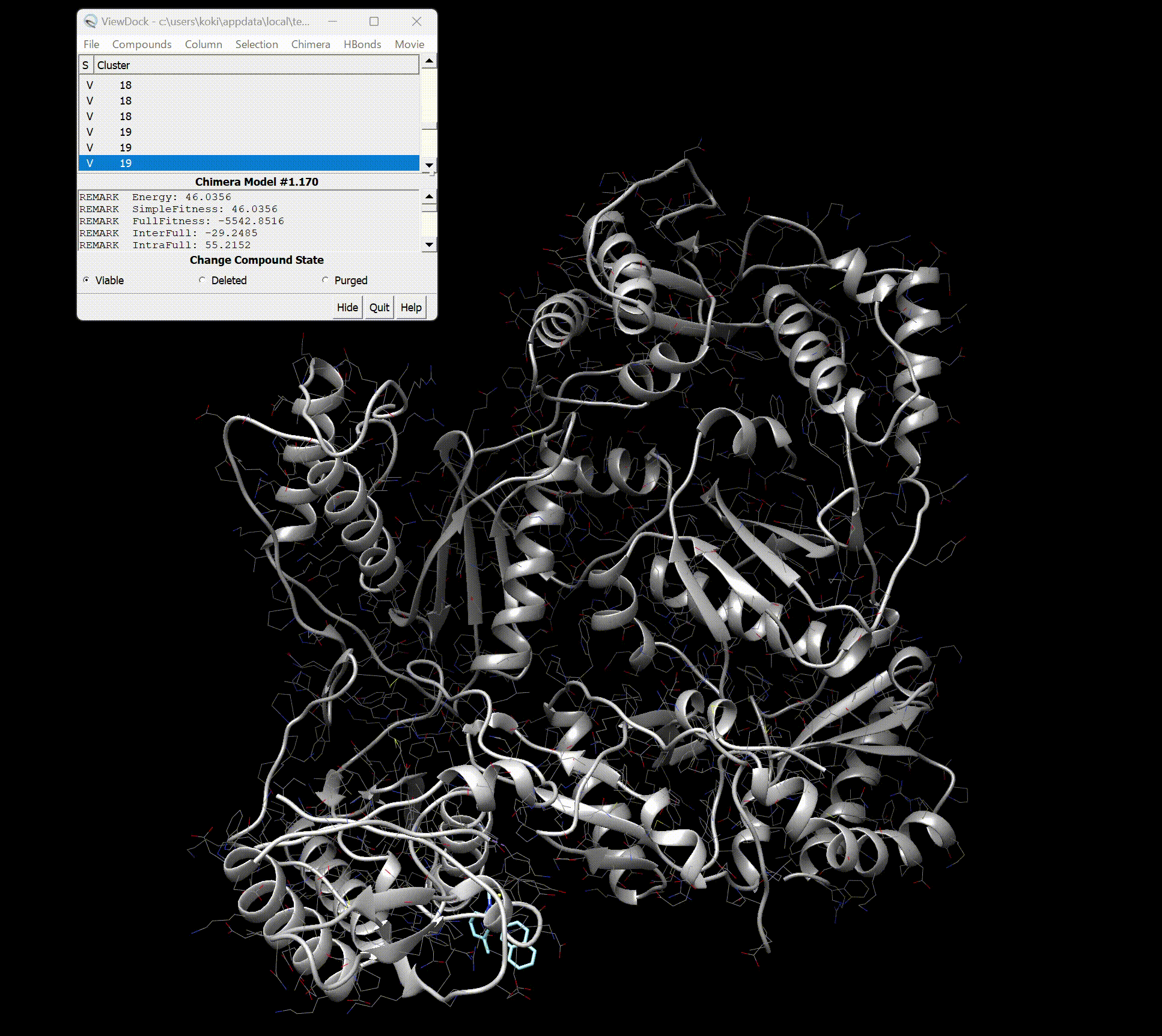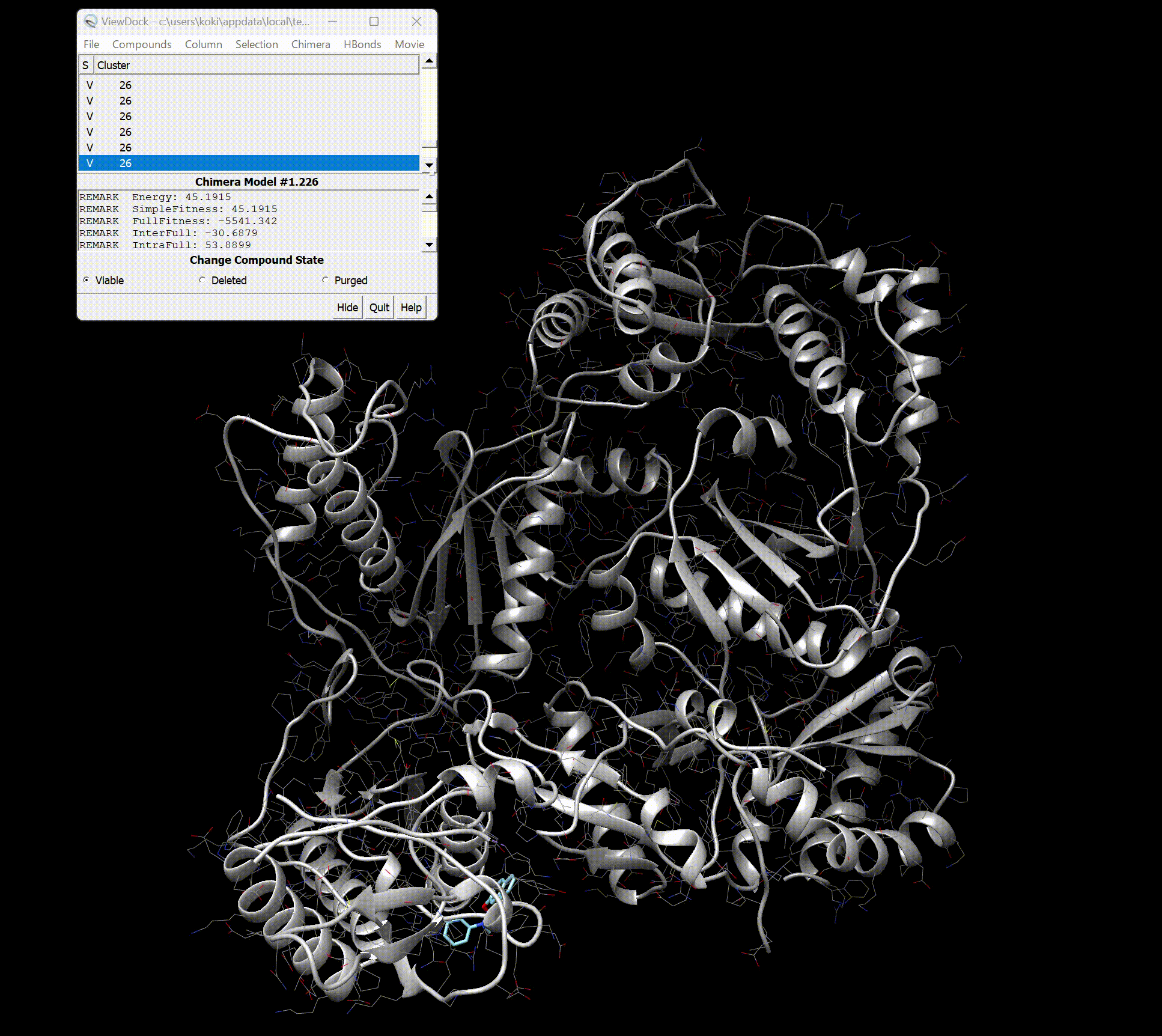
最後に
いかがでしたでしょうか?これまでAuto DockやPyRx, 機械学習など様々なスクリーニング技術を紹介してきました!それらで見つけてきた化合物を今度は分子Dockingさせてみて、もっと詳細なDockingを試してみて下さい!
参考文献
バイオインフォマティクス: Swissdock タンパク質リガンドのドッキング |バイオコード株式会社
Lesson 12: Protein docking with SwissDock and UCSF-Chimera
SwissDock, a protein-small molecule docking web service based on EADock DSS.
Fast docking using the CHARMM force field with EADock DSS.
自宅でできるin silico創薬の技術書を販売中
¥3,200 → ¥1500 今なら50%OFF!!
新薬探索を試したい方必読!
ITエンジニアである著者の視点から、wetな研究者からもdryの創薬研究をわかりやすく身近に感じられるように解説しています
自宅でできるin silico創薬の技術書を販売中
¥3,200 → ¥1,500 今なら50%OFF!!
分子ドッキングやMDシミュレーションなど、
自宅でできるin silico創薬の解析方法を解説したものになります!





