この記事では、分子ドッキングというコンピュータシミュレーション手法とその具体例であるPatchDockについて説明しています。分子ドッキングは薬物とタンパク質の相互作用を予測し、新薬開発やタンパク質研究に役立ちます。PatchDockは分子の形状マッチングを利用して結合を予測する手法です。記事ではPatchDockの使い方や結果の解釈について詳しく解説しており、分子ドッキングの基本的な原理と応用のメリットを学ぶことができます。ぜひトライしてみてください!
macOS Ventura(13.2.1), PyMOL 2.5.4
自宅でできるin silico創薬の技術書を販売中
¥3,200 → ¥1500 今なら50%OFF!!
新薬探索を試したい方必読!
ITエンジニアである著者の視点から、wetな研究者からもdryの創薬研究をわかりやすく身近に感じられるように解説しています
自宅でできるin silico創薬の技術書を販売中
¥3,200 → ¥1,500 今なら50%OFF!!
分子ドッキングやMDシミュレーションなど、
自宅でできるin silico創薬の解析方法を解説したものになります!
本記事を進むにあたって、PyMOLのダウンロードをお願いします。
大阪大学の蛋白研究所からインストールの仕方が解説されています。
分子ドッキングとは?
分子ドッキングとは、薬品や生体分子(例えばタンパク質)がどのように相互作用するかを予測するコンピュータシミュレーションのことです。特定のタンパク質と薬物がどのように結合するか、どの部分が互いに触れ合うかを計算し、その結果から薬物の効果を予測します。これは新薬開発などに非常に役立ちます。現在様々なwebで使うことのできる分子ドッキング手法があります。今回はその中でも、PatchDockについて紹介していきます。
タンパク質の下準備
まず各手法の解析の前にドッキングするタンパク質の下準備をしていきたいと思います。今回はモデルのタンパク質としてProtein Data Bank(PDB)の番号8DHAであるレプチンとその受容体を使います。
レプチンは、脂肪組織に由来するタンパク質ホルモンであり、体重とエネルギーバランスの調節に重要な役割を果たしています。レプチンは脂肪細胞から血液中に放出され、脳の視床下部に存在する特定の受容体に結合することで、食欲を抑制し代謝を調節する作用を持っています。
薬物としてのレプチンの魅力は、肥満治療における可能性です。レプチンの欠乏または抵抗は、肥満の原因となることが知られています。そのため、レプチンの補充や受容体の活性化を促進する薬剤の開発が進められています。
レプチン薬物の魅力は、食欲を抑制する効果と体重減少をもたらす可能性です。これにより、肥満者やメタボリックシンドロームの患者にとって、体重管理や疾患リスクの低減に役立つことが期待されています。さらに、レプチンは食欲を制御する神経回路にも関与しているため、食欲をコントロールする新たな治療法の開発にもつながる可能性があります。
最近ではインスリンを用いずに、レプチンを使って、血糖値を下げることにも成功しており、糖尿病への治療へも期待されています。
(PDB)の番号8DHAであるレプチンとその受容体は結合している箇所のみを実際に取り出したもので、全体は8DH8に登録されています。今回は計算量を少なくするために、番号8DHAであるレプチンとその受容体を使います。
まずPymolのタブのFile→Get PDB…に8DHAと打ち、レプチンとその受容体を表示させます。
続いてDisplay→Sequenceより全体の配列を表示します。
/C/C/1から始まる部位がレプチンに該当するので、この配列を選択します。
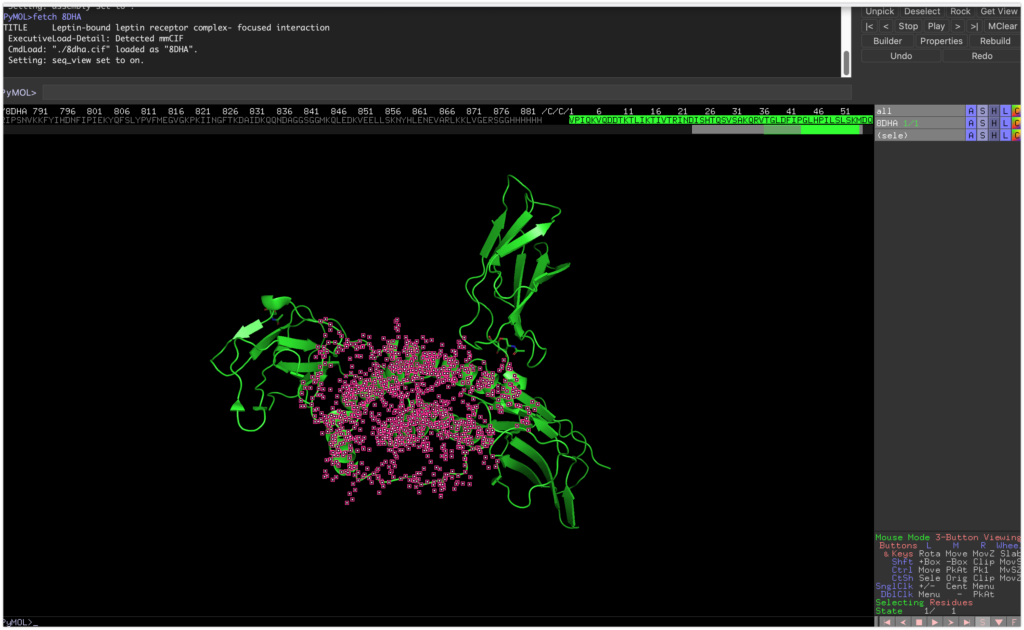
続いて、以下のコードを打ち、実行します。
save leptin.pdb, sele
これによって、選択部位がleptin.pdbとして保存されます。
続いてレプチン以外の表示されているレプチン受容体の部分を選択し、
save focus_leptinR.pdb, sele
として保存します。2-acetamido-2-deoxy-beta-D-glucopyranoseも一緒に結合していますが、こちらの部分は選択しないでください。
これによって、選択部位がfocus_leptinR.pdbとして保存されます。
これらのデータはMacintosh HD/ユーザ/自分のパソコンの名前
の階層に保存されていると思います。
以上でタンパク質の下準備は終了です。
PatchDockとは?
PatchDockとは、分子がどのように組み合わさるかを予測するためのツールです。例えば、タンパク質やDNA、薬物などの分子がどのように組み合わさるかを予測します。
このツールの使い方は、ジグソーパズルを解くのに似ています。ジグソーパズルでは、一つのピースを選んで、それに合うピースを探しますよね。PatchDockも同じように、一つの分子(ピース)を選んで、それに合う分子を探します。
早速PatchDockのサイトに行ってみましょう。
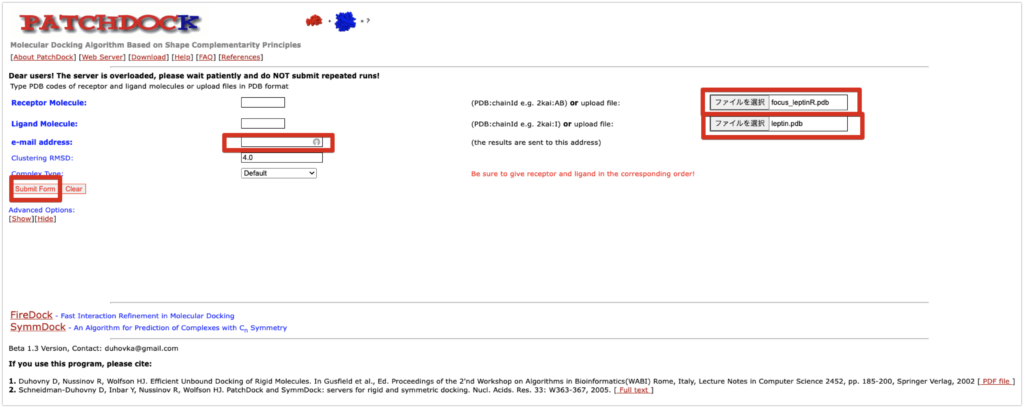
以下を設定してください。
- Receptor Moleculeに
leptin.pdbを選択します。 - Ligand Molecule:に
focus_leptinR.pdbを選択します。 - emailを書きます。
- 他の設定はそのままにしておきます。
- しばらくすると、入力したメールに結果が送られてきます。
PatchDockの原理
2つの分子が与えられると、その表面は表面形状に従ってパッチに分割されます。これらのパッチは、パズルのピースを視覚的に区別するパターンに対応します。パッチが特定されると、形状マッチングアルゴリズムを使用して重ね合わせることができます。アルゴリズムは主に3つのステージから構成されています:
- 分子形状の表現:このステップでは、分子の表面を計算します。次に、幾何学的なパッチ(凹部、凸部、平面部分)を検出するためのセグメンテーションアルゴリズムを適用します。パッチはフィルタリングされ、’ホットスポット’残基を含むパッチのみが保持されます。
- 表面パッチマッチング:このステップでは、前のステップで検出されたパッチをマッチさせるために、幾何学的ハッシングとポーズクラスタリングマッチング技術のハイブリッドを適用します。凹部のパッチは凸部と、平面のパッチは任意のタイプのパッチとマッチします。
- フィルタリングとスコアリング:このステップでは、前のステップからの候補複合体を検査します。受容体の原子がリガンドの原子に不適切に侵入しているすべての複合体を破棄します。最後に、残りの候補を幾何学的な形状補完性スコアによってランク付けします。
結果
結果は次のようになりました。
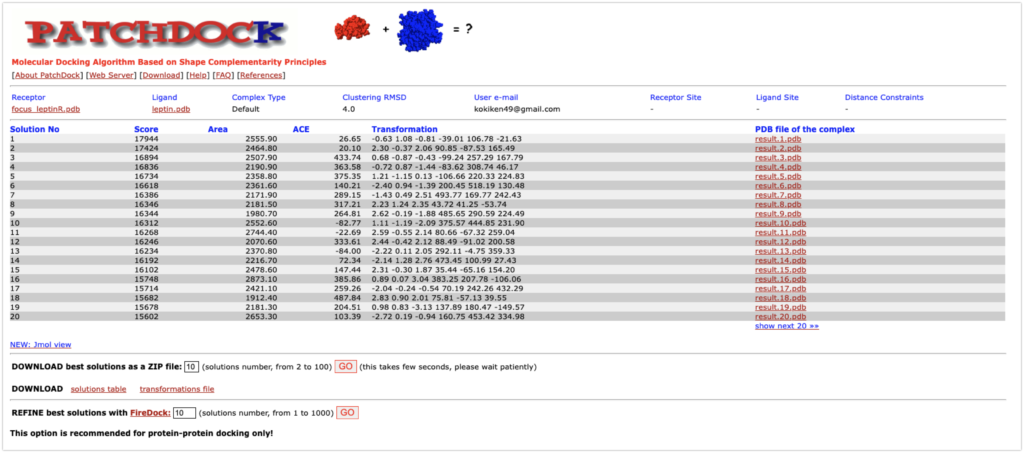
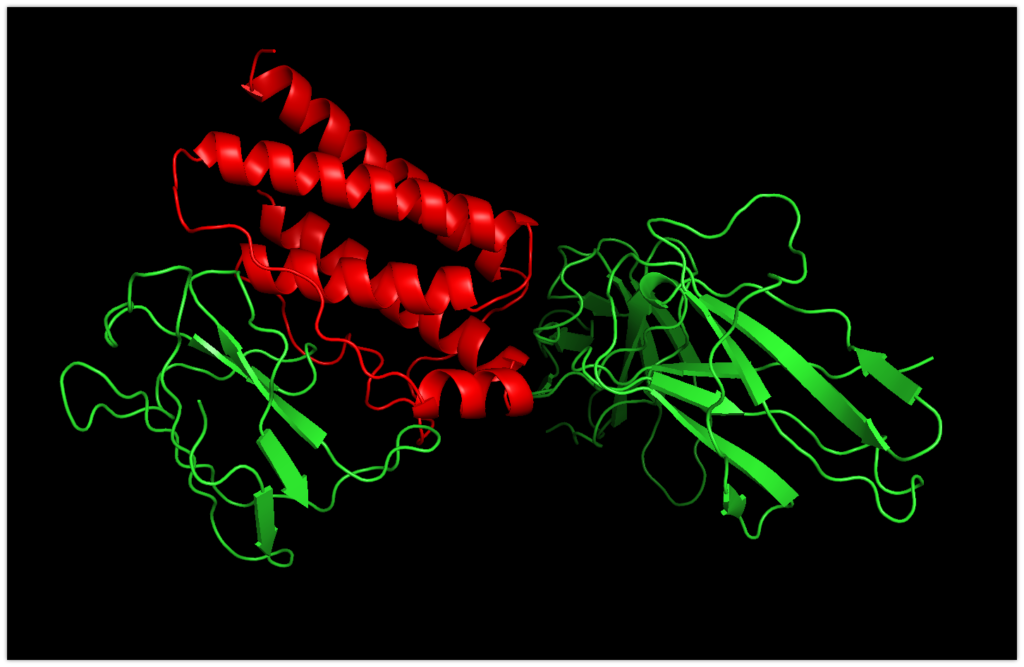
最も高いスコアを示したもの(Solution No.1)をpymolで表示します。
赤がleptinで緑がleptinレセプターです。
さて、これを元々の構造と重ねてみましょう。
マゼンダ色のものが元々の配列です。
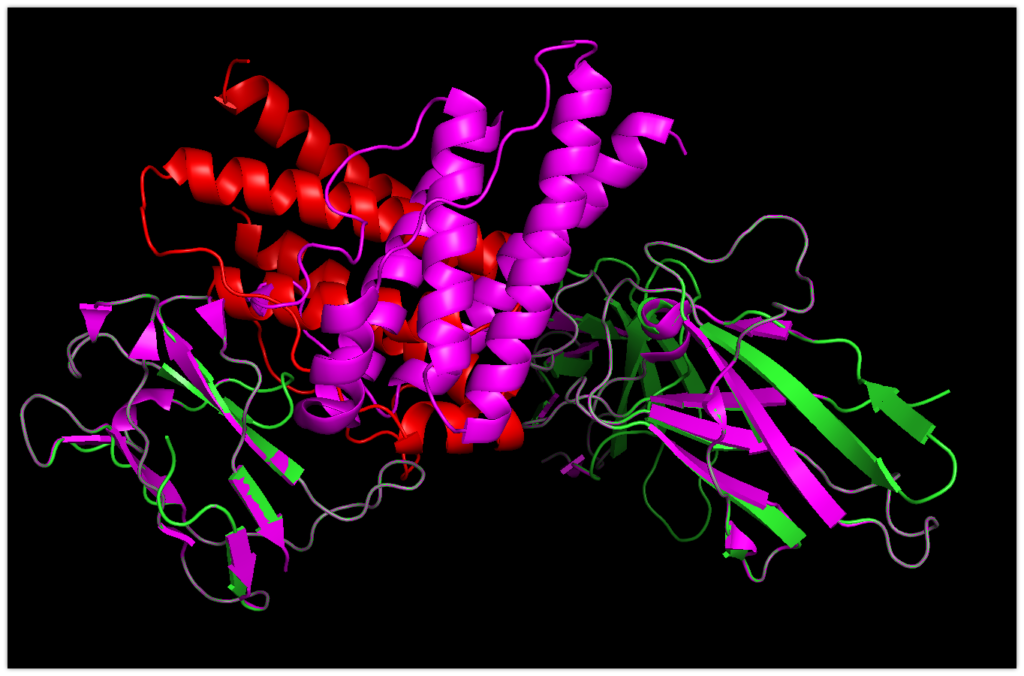
おっと、意外と違う形で結合してそうですね。。。
今回のPatchDockは形状的にうまくハマることでドッキングしているので、結果的に相互作用面が大きい方に重点がいき、赤色のPatchDockのleptinのように結合したのだと思われます。また今回のレセプター分子は二分子あるので、両方の形状にハマることは難しかったのかもしれません。事実、赤色のPatchDockのLeptinは左側のレセプターの方に相互作用面が大きく重なっています。
最後に
いかがでしたでしょうか?本日はタンパク質ータンパク質ドッキング法の中でPatchDockについて取り上げてみました。こちらはサーバーで簡単にドッキングを試すことができるので、ぜひ皆さんも試してみてください。以前の記事ではClusProを取り上げましたが、こちらとは原理が少し違います。興味がある方はこちらもどうぞ。今後もタンパク質-タンパク質ドッキング法を取り上げていくので、ぜひ楽しみにしておいてください!
参考文献
Protein – Protein Docking Beginner Tutorial
PatchDock and SymmDock: servers for rigid and symmetric docking





