本記事では、プログラミング言語Rを利用した生存時間解析のやり方を紹介しています。
特に利用実績のあるRパッケージsurvivalとsurvminerを利用し、前回の記事で行ったKaplan-Meier曲線(KM曲線)やログランク検定に加えて、複数の変数が生存に与える影響を同時に評価できる Cox比例ハザードモデル(Cox回帰)を実行して行きます。
macOS Sequoia (15.5)
公共データを用いたSingle Cell RNA-seq解析に関する初心者向け技術書を販売中
プログラミング初心者でも始められるわかりやすい解説!
RとSeuratで始めるSingle Cell RNA-seq解析!
公共データを用いたシングルセル ダイナミクス解析に関する初心者向け技術書を販売中
シングルセルデータの高度な解析であるTrajectory解析、RNA Velocity解析、空間トランスクリプトーム解析が環境構築方法から詳しく解説されています!
TCGAデータ x R を用いた生存時間解析に関する技術書を販売中
R言語による生存時間解析の手法をステップバイステップで解説!自分のPCとRさえあれば、費用をかけずに本格的なサバイバル解析を体験できます
Cox回帰とは?
Cox比例ハザードモデル(Cox回帰) とは、生存時間データにおいて、共変量$x_1, x_2, …, x_n$とイベント発生の関係性を解析するための統計モデルです。 共変量とは、性別・年齢・身長・体重など、イベント発生に関係しそうな患者ごとの特徴を指します。 オミックス解析では遺伝子発現や遺伝子の変異の有無なども共変量となります。 Cox回帰では、ハザード関数(単位時間あたりのイベント発生率)を、回帰モデル
$$ \eta = \beta_1 x_1 + \beta_2 x_2 + …, \beta_n x_n $$
として表現し、各変数の影響を推定します。 イベントに強く関与する変数(例:$x_i$)ほど、対応する係数(例:$\beta_i$)は0から大きく離れた値となります。
この線型結合の値$\eta$はリスクスコア、またはPrognostic Index(PI)などと呼ばれ、値が高い患者ほど、イベント(例: 死亡)が発生するリスクが高いと判断されます。 そのため、患者での$\eta$の平均値、中央値など、何かしらの閾値を設定して、$\eta$の値が高い高リスク群と、値が低い低リスク群に層別化し、各々の群でのKM曲線の推定や、ログランク検定による二群間の比較が行われます。
なぜRを使うのか?
前回は、TCGAの公共オミックスデータを用いた生存時間解析を手軽に実施できるGDC Data Portal、UCSC Xena、KM-PlotterといったWebアプリを紹介しました。 これらのツールは、プログラミングの知識がなくても即座に使えるという点で非常に優れており、初学者がデータの可視化や初期的な仮説検証を行うには有用な手段です。
一方で、本格的な解析や研究成果としての再現性・柔軟性を重視する場合には、Rなどのプログラミング環境を用いて解析を行うことが強く推奨されます。その理由は以下の通りです。
- 解析の透明性と再現性: GUIでは、クリック操作で得られた結果の「内部的な処理」がブラックボックスになりがちです。一方、Rでは実行したコードがそのまま解析の記録となるため、他者が同じ処理を再現できます。これは論文執筆やレビュー時にも大きな強みとなります。
- 柔軟なカスタマイズ: GUIでは層別の方法、時点ごとの検定、図のデザインなどに制限があります。Rでは、任意の特徴量に基づく層別化、生存時間のカットオフ、サブグループ解析、可視化のスタイル変更などを自在に行うことができます。特にRは標準のグラフィック機能に加え、
ggplot2やplotlyなど、各種最先端の可視化ツールが利用可能です。 - 解析対象の拡張性: GUIツールの多くは単一遺伝子ごとの解析を想定していますが、Rを使えば多変量Cox回帰やLASSO、機械学習モデルの利用、リスクスコアの構築、クロスバリデーションによる検証、予測モデルの構築といった発展的解析まで実装可能です。
- 自動処理・スケーラブルな解析: 複数の遺伝子に対して一括でCox回帰を行ったり、100件以上の条件で生存曲線を生成するといった作業は、GUIでは手作業の繰り返しになってしまいます。Rではループや関数化により、自動かつ効率的に大量の解析が可能です。
- パイプラインへの組み込み: 生存時間解析は、通常は前後のデータ前処理や他の統計解析と連携して行われます。Rで解析を組むことで、前後の処理(正規化・欠損処理・可視化・結果出力)を一貫して統合できます。
そのため以降では、R言語を利用し、生存時間解析の定番パッケージであるsurvivalと、survivalの結果のグラフ描画をサポートするsurvminerで、生存時間解析を行います。
ここでは、本格的なオミックスデータでのCox回帰を行う前の準備として、まず生存曲線の作成やログランク検定、Cox回帰の実行方法を確認します。
まずは用意されたサンプルデータで単変量解析(1つの要因による生存曲線の層別化・比較)を行い、次に多変量Cox回帰によるリスクスコア解析を行います。
survival / survminerのインストール
R言語やR-studio(またはPositron)は事前にインストールされているものとします。
各OSにインストールされたR(WindowsならR.exe, MacならR.app)のコンソール画面か、もしくはR-Studio/Positronを起動し、以下のコマンドを入力することで、survivalやsurvminerの内部で実装された関数や、内部データを利用できるようになります。
# survival / survminerがない場合は、インストール
if (!require("survival", quietly = TRUE)) install.packages("survival")
if (!require("survminer", quietly = TRUE)) install.packages("survminer")
# パッケージをロード
library(survival)
library(survminer)KM曲線とログランク検定
Rのsurvivalパッケージには生存時間解析のデモ用データがいくつか含まれています。その一つに肺がん患者のデータセットlungがあります。
まず、このデータを使って性別ごとのKM曲線を描き、ログランク検定で差の有無を確認してみましょう。
head(lung) # データの先頭部分を確認
# inst time status age sex ph.ecog ph.karno pat.karno meal.cal wt.loss
#1 3 306 2 74 1 1 90 100 1175 NA
#2 3 455 2 68 1 0 90 90 1225 15
#3 3 1010 1 56 1 0 90 90 NA 15
#4 5 210 2 57 1 1 90 60 1150 11
#5 1 883 2 60 1 0 100 90 NA 0
#6 12 1022 1 74 1 1 50 80 513 0lungデータのtimeが生存時間(日数)、statusがイベント発生の有無です。
ここでstatusは1=censored(打ち切り)、2=dead(死亡)となっているため、そのままでは「2=イベント発生」を意味します。survivalパッケージのSurv()関数では、イベント発生を示す引数は通常0/1を想定しています。
そのため、status==2 とすることで「死亡の場合TRUE(=1)、打ち切りの場合FALSE(=0)」の論理値ベクトルを作り、生存オブジェクトを生成します。
# 生存オブジェクトの作成(status==2 がイベント発生を示す)
surv_obj <- Surv(time = lung$time, event = (lung$status == 2))
surv_obj[1:5] # 最初の5例を確認
#> [1] 306 455 1010+ 210 883上記の出力では、数値の後ろに+が付いているものは打ち切り(生存で観察終了)を示します。
例えば1010+は1010日で打ち切られたことを意味します。
次に、この生存オブジェクトを用いてKM曲線を推定します。survfit()関数に「~ 性別」のような式を書けば、性別ごとの生存曲線をフィッティングできます。
(fit_sex <- survfit(surv_obj ~ sex, data = lung))
# Call: survfit(formula = surv_obj ~ sex, data = lung)
#
# n events median 0.95LCL 0.95UCL
#sex=1 138 112 270 212 310
#sex=2 90 53 426 348 550ここでmedianは、中央値生存時間を意味しており、男性群では270日、女性群は426日、といった具合に読み取れます。
また、survminerパッケージのggsurvplot()関数を使うと、美しく整形された生存曲線をプロットできます。
プロットにはp値(ログランク検定)や生存人数(リスクセット)の表を付けるオプションもあり、論文やレポートにそのまま利用できるクオリティのグラフが得られます。
# 生存曲線を描画
ggsurvplot(fit_sex, data = lung, pval = TRUE, risk.table = TRUE, surv.median.line = "hv", legend.labs = c("Male", "Female"), xlab = "Time (days)", ylab = "Survival probability", conf.int = TRUE)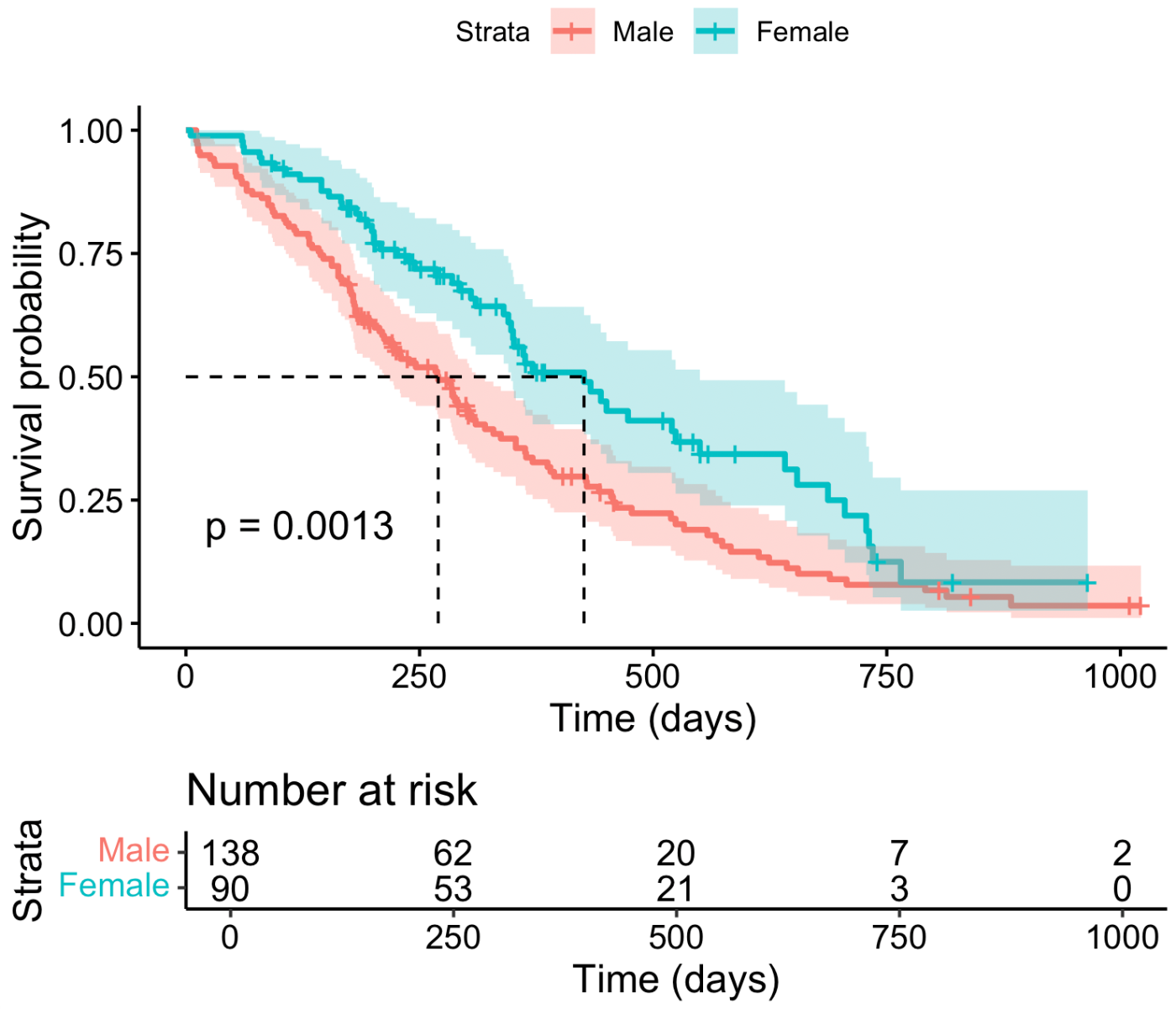
上図は男性(赤)と女性(青)のKM曲線を示しています。 陰影部分は95%信頼区間で、下段に時点ごとの生存人数(risk table)が表示されています。中央値生存時間に水平線・垂直線が引かれています。 ログランク検定のp値も自動で付加されます。 縦線は打ち切りが発生したタイミングです。
2つの生存曲線を見比べると、赤線が青線よりも早く値が降下しており、ログランク検定のp値も0.0013であり、有意水準を0.05と設定した場合、2群間で有意な生存期間の差があると判断できます。
なお、ログランク検定の結果だけを数値で確認したい場合は、survdiff()関数を用います。
その出力からカイ二乗値や自由度を取得し、p値を計算できます。
(test_sex <- survdiff(surv_obj ~ sex, data = lung))
#Call:
#survdiff(formula = surv_obj ~ sex, data = lung)
#
# N Observed Expected (O-E)^2/E (O-E)^2/V
#sex=1 138 112 91.6 4.55 10.3
#sex=2 90 53 73.4 5.68 10.3
#
#Chisq= 10.3 on 1 degrees of freedom, p= 0.001上記の結果から、自由度1のカイ自乗統計量は10.3で、対応するp値は約0.001となっています。 そのため確かに性別で層別化した二群での生存曲線間には有意差があることがわかります。
Cox回帰によるリスクスコアと生存曲線
続いて、多変量のCox回帰モデルを用いてみましょう。 上記の性別で層別化した例のような単一因子の解析では、他の要因の影響を無視していますが、現実には年齢や他の臨床指標が生存に影響を与えている可能性があります。 Coxモデルならそれらを同時に扱うことができます。 また、Coxモデルの結果からリスクスコアを計算し、患者を予後の良し悪しで層別化するといった応用も可能です。
ここでは例として、lungデータから年齢(連続変数)と性別(カテゴリ変数)を説明変数にしたCox回帰を行ってみます。
そして各患者について、Coxモデルから算出されるリスクスコアを用いて高リスク群と低リスク群に分け、生存曲線を比較します。
# 多変量Cox比例ハザードモデルの適用(年齢と性別を共変量に設定)
cox_fit <- coxph(Surv(time, status == 2) ~ age + sex, data = lung)
summary(cox_fit)
#Call:
#coxph(formula = Surv(time, status == 2) ~ age + sex, data = lung)
#
# n= 228, number of events= 165
#
# coef exp(coef) se(coef) z Pr(>|z|)
#age 0.017045 1.017191 0.009223 1.848 0.06459 .
#sex -0.513219 0.598566 0.167458 -3.065 0.00218 **
#---
#Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
#
# exp(coef) exp(-coef) lower .95 upper .95
#age 1.0172 0.9831 0.9990 1.0357
#sex 0.5986 1.6707 0.4311 0.8311
#
#Concordance= 0.603 (se = 0.025 )
#Likelihood ratio test= 14.12 on 2 df, p=9e-04
#Wald test = 13.47 on 2 df, p=0.001
#Score (logrank) test = 13.72 on 2 df, p=0.001上記のCoxモデル結果では、年齢のハザード比(exp(coef))は約1.0172、性別(sexは男性=1, 女性=2と符号化)のハザード比は約0.5986となっています。
性別については、男性を基準としたとき、女性の死亡リスクは約40%低い(ハザード比0.586)と解釈されます。
これは統計的にも有意であり(p = 0.00218)、生存において性別が独立した予測因子である可能性が高いことを示唆します。
一方、年齢のハザード比は1.0172であり、年齢が1歳上がるごとに死亡リスクが約1.7%上昇することを意味します。 ただし、この効果のp値は0.06459と有意水準0.05をわずかに超えており、統計的に明確な差とまでは言えません。 傾向としては年齢が高いほどリスクが高まる可能性があるものの、慎重な解釈が必要です。
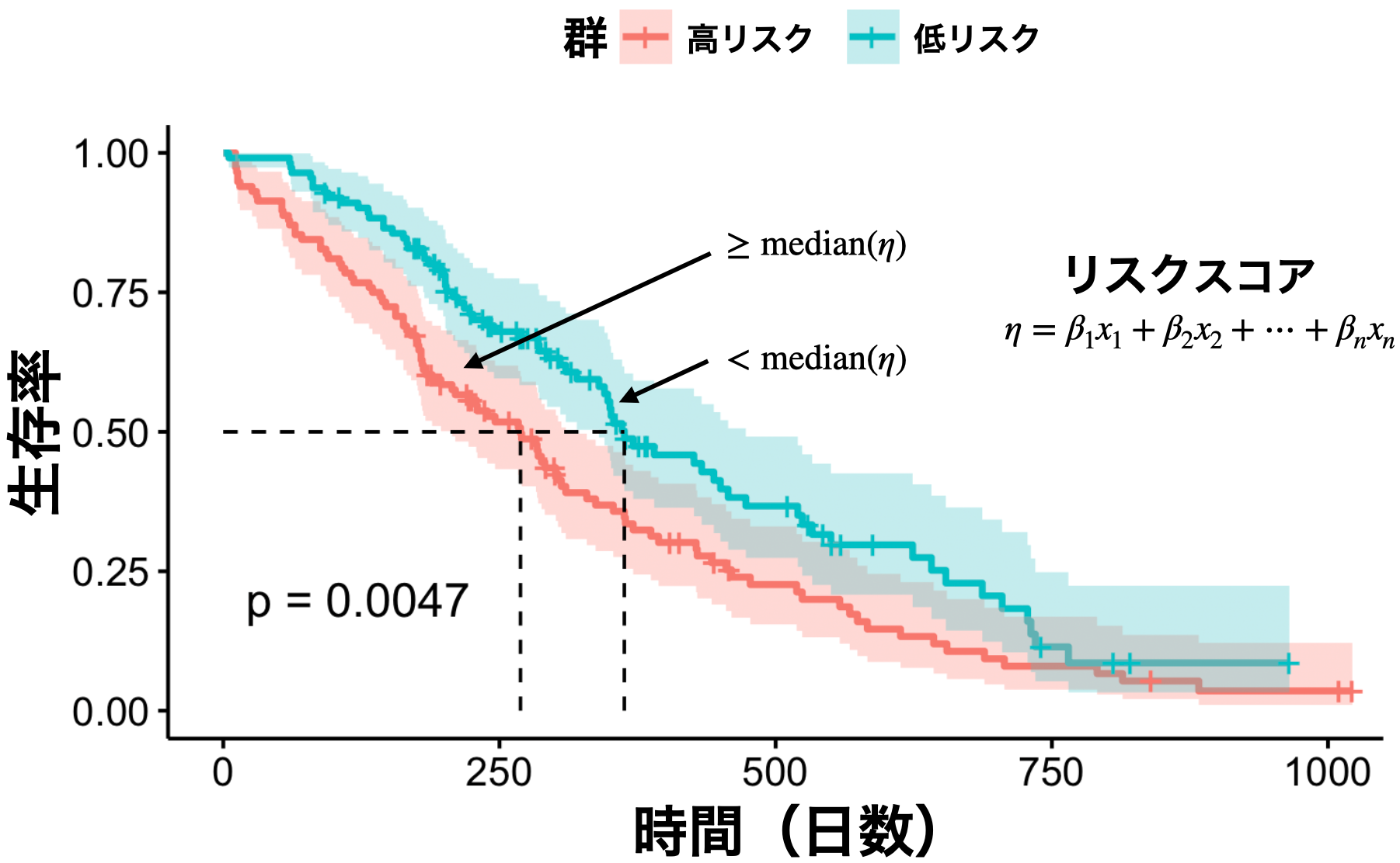
モデルが得られたら、次に各患者についてリスクスコアを計算します。
Rでは、predict(cox_fit, type="risk")で各患者の相対的なリスク(指数スケール)を計算できますが、ここでは線形予測子そのもの(type="lp")を用いても順位付けは同じです。
簡単のため、リスクスコアの中央値で区切って高リスク群(スコアが高い=予測されるハザードが高い)と低リスク群に2分割します。
# 各患者のリスクスコア(線形予測子)を計算
risk_score <- predict(cox_fit, type = "lp")
# リスクスコアの中央値で高リスク/低リスクに分類
median_score <- median(risk_score, na.rm = TRUE)
risk_group <- ifelse(risk_score >= median_score, "High risk", "Low risk")
table(risk_group)
#risk_group
#High risk Low risk
# 116 112リスク群に分けられたので、この2群間で生存曲線の差を見てみます。
再びsurvfit()と ggsurvplot()を用いてKaplan-Meier曲線をプロットし、ログランク検定のp値も確認します。
# リスク群ごとの生存曲線
fit_risk <- survfit(Surv(time, status == 2) ~ risk_group, data = lung)
ggsurvplot(fit_risk, data = lung, pval = TRUE, risk.table = TRUE, surv.median.line = "hv", legend.labs = c("High risk", "Low risk"), xlab = "Time (days)", ylab = "Survival probability", conf.int = TRUE)
上記のグラフにより、高リスク群は生存率の低下が早く、低リスク群は緩やかである、といった結果が視覚的に得られます。
ログランク検定のp値が有意であれば、「年齢と性別を考慮したCoxモデルによるリスク評価で、高リスク群と低リスク群の生存期間に有意差が認められた」という解釈になります。 このようにCox回帰を用いることで、複数因子を統合した予後予測指標を作成し、生存曲線として提示することが可能です。
最後に
本記事では、GUIツールを用いず、プログラミング言語Rを用いた生存時間解析の基本操作を体験しました。 Rを利用することで、前回のGUIで行っていたKM曲線の作成やログランク検定を、コマンドベースで実行できるようになり、より柔軟で再現性の高い解析が可能となります。
さらに、本記事では複数の共変量と生存時間の関係をモデル化するCox回帰も紹介しました。 肺がん患者データを用いた解析では、リスクスコアに基づいて2群に分けた際、生存に有意な差が見られることを確認しました。
次回は、Rを用いてTCGA(The Cancer Genome Atlas)の大規模がんゲノムデータにアクセスし、実際の解析に活用する方法を紹介します。 ぜひ引き続きご覧ください。
公共データを用いたSingle Cell RNA-seq解析に関する初心者向け技術書を販売中
プログラミング初心者でも始められるわかりやすい解説!
RとSeuratで始めるSingle Cell RNA-seq解析!
公共データを用いたシングルセル ダイナミクス解析に関する初心者向け技術書を販売中
シングルセルデータの高度な解析であるTrajectory解析、RNA Velocity解析、空間トランスクリプトーム解析が環境構築方法から詳しく解説されています!
TCGAデータ x R を用いた生存時間解析に関する技術書を販売中
R言語による生存時間解析の手法をステップバイステップで解説!自分のPCとRさえあれば、費用をかけずに本格的なサバイバル解析を体験できます









