Network pharmacologyとは、薬物、標的、疾患の相互関係をネットワークとして捉え、複雑な生体システム内での薬の作用を網羅的に解析する創薬手法です。この手法を使えば、漢方薬の成分がどの標的に効果があるかについて知ることができます。この記事ではまず漢方薬の成分について、PubChemから抽出してみます。
【この記事のまとめ】
ネットワーク薬理学の初学者やin silico創薬の研究者を対象に、PubChemから漢方薬(黄芩)の成分情報をPythonを用いて効率的に抽出・自動収集する具体的手順を解説します。
- ネットワーク薬理学のワークフロー提示: 漢方薬のような「多成分・多標的」の作用機序を解明するため、成分取得から標的予測、ネットワーク可視化に至る一連の解析フローを紹介。
- Seleniumによる自動スクレイピング: 手動でのダウンロードが困難な大量の化合物情報を、Python(Selenium)を用いてPubChemのTaxonomyセクションからCID(化合物ID)として自動取得するコードを公開。
- API連携とドラッグライクネス評価:
pubchempyを利用してCIDからSMILESやIUPAC名を一括取得し、さらにRDKitを用いて「Lipinski’s Rule of Five」などの基準に基づき薬物性の高い成分のみを抽出するフィルタリング手法を詳説。
この記事を読むことで、ライセンス制限のあるデータベースに頼らず、公共のPubChemデータから解析に必要な「化合物リスト」を自力で構築する技術を習得できます。
Mac M1, Sequoia 15.3
自宅でできるin silico創薬の技術書を販売中
新薬探索を試したい方必読!
ITエンジニアである著者の視点から、wetな研究者からもdryの創薬研究をわかりやすく身近に感じられるように解説しています
自宅でできるin silico創薬の技術書を販売中
タンパク質デザイン・モデリングに焦点を当て、初めてこの分野に参入する方向けに、それぞれの手法の説明から、環境構築、実際の使い方まで網羅!
Network pharmacologyとは?
Network pharmacology(ネットワーク薬理学)は、漢方薬や機能性食品に含まれる複数の成分が、体内のさまざまな標的(タンパク質、遺伝子など)に同時に作用し、複雑な生理的効果をもたらす仕組みを「ネットワーク」として解析する手法です。
従来の薬理学が「1成分=1標的」の考え方に基づいていたのに対し、ネットワーク薬理学は「多成分=多標的=多経路」の全体像をとらえます。たとえば、漢方薬「黄芩」に含まれるバイカリンなどの成分が、乳がんに関与する複数の遺伝子やシグナル経路に作用している可能性を、各種データベースを用いて可視化できます。
これにより、伝統的処方の有効性を科学的に裏付けたり、新たな疾病への応用可能性を探索したりすることができます。
Network pharmacologyの流れ
- 成分取得:PubChemなど各種データベースを使って、漢方や食品中の有効成分を調査する。
- 標的予測:SwissTargetPrediction、ChEMBL Multitask Neural Network modelなどを用いて、成分が結合する可能性のある標的タンパク質を予測する。
- 疾患関連遺伝子の収集:Open Targetsを使って、対象とする疾患に関係する遺伝子やタンパク質の情報を収集する。
- 共通ターゲットの抽出:成分の標的と疾患関連遺伝子を照合し、共通の遺伝子(ターゲット)を見つける。
- ネットワーク構築・可視化:成分、標的、疾患の関係性をCytoscapeなどでネットワーク図として可視化する。
- (in silico screening):これまで得られた化合物とタンパク質に対して、分子ドッキングを行います。
- (分子動力学シミュレーション(MD)):6の結果、結合が良かった化合物とタンパク質をMDしていきます。
以下の論文を参考にし、漢方のScutellaria baicalensis(オウゴン (黄芩))の成分と乳がんの標的タンパク質を明らかにし、黄芩のどの成分が、乳がんの標的に結合するか明らかにしていきます。
今回は1. 成分取得について、PubChemを使いながら、ご説明します。
論文ではTCMSP platform (http://tcmspw.com/tcmsp.php)も使われておりますが、ライセンスの関係上今回は使用しません。アカデミアの方は使用されると良いと思います。
Pubchemによる成分抽出
以下の手順で漢方のScutellaria baicalensis(オウゴン (黄芩))の成分を取得してみましょう!
- まずはPubChemにアクセスし、
Scutellaria baicalensisを検索して下さい。
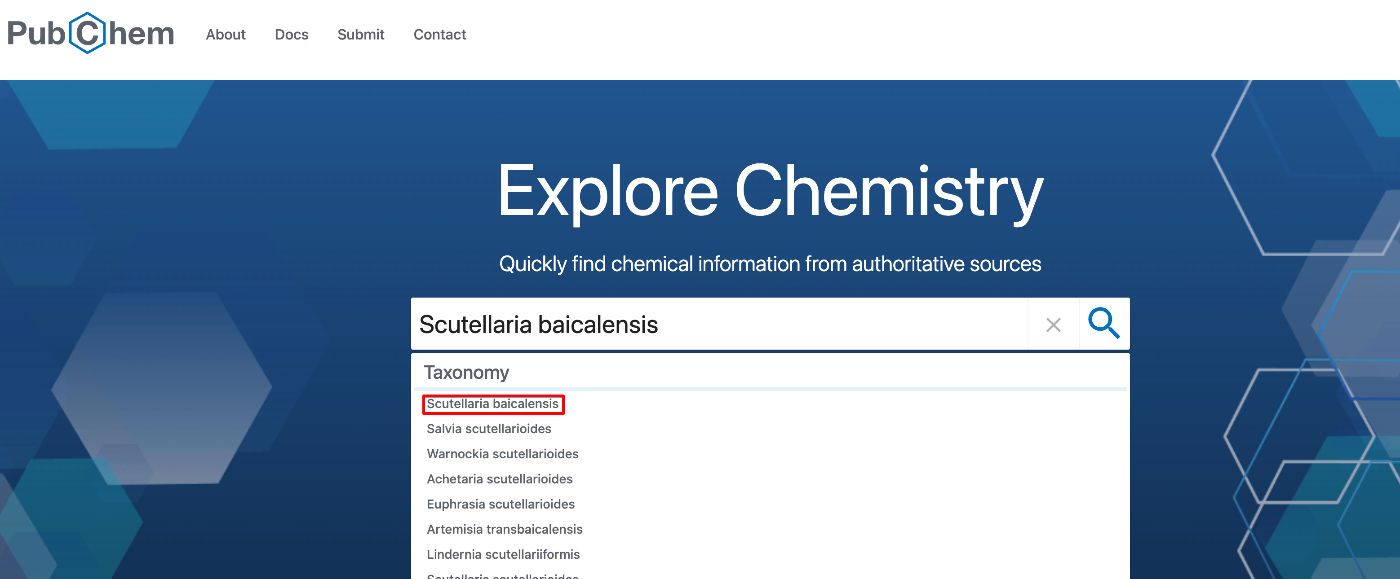
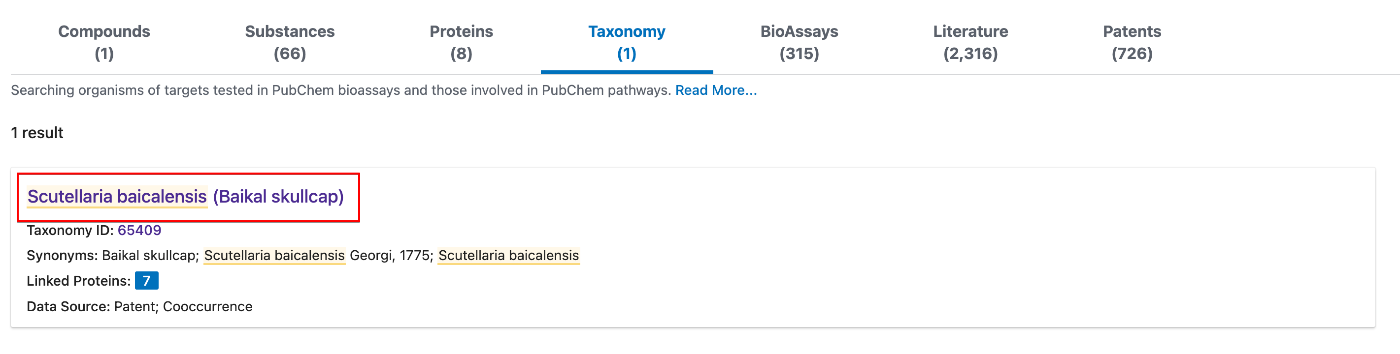
- Taxonomyのの箇所に、
Scutellaria baicalensisがあるので、こちらをクリックして下さい。
- 下の方にNatural Productsがあるので、ここのダウンロードを押して下さい。csv形式で良いと思います。
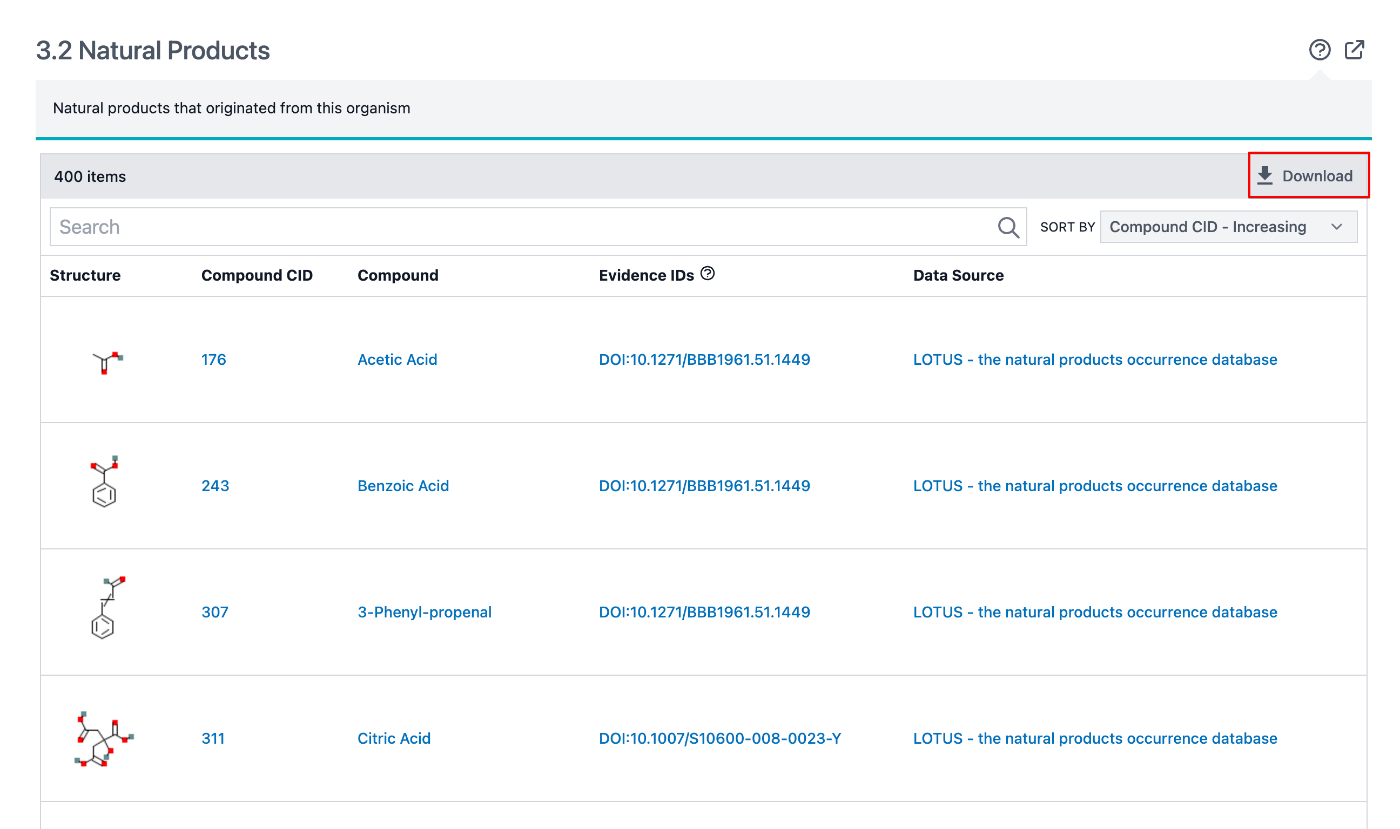
一つ一つダウンロードするのも良いですが、時間がかかるので、スクレイピングとAPIを使った化合物情報の取得で取得していきましょう!
スクレイピングとAPIを使った化合物情報の取得
まずは以下でスクレイピングとフィルタリングの環境を構築しましょう。
環境構築
まずは以下で環境構築をしていきます。
conda create -n scraping python=3.11
conda activate scraping
pip install pandas requests beautifulsoup4 selenium
brew install chromedriver
pip install selenium pandas tqdm
Cursor、VSCodeをお使いの方は、以下で環境を選べるようになります。
詳細な説明
1. 仮想環境の作成
conda create -n scraping python=3.11
このコマンドは、新しい仮想環境を作成します。仮想環境とは、特定のプロジェクトごとにライブラリやPythonのバージョンを分けて管理できる作業空間のことです。
n scraping:この仮想環境の名前をscrapingに設定します。python=3.11:この環境で使用するPythonのバージョンを 3.11 に指定しています。
これにより、他の環境と影響を与え合わずに安全にスクレイピングの開発ができます。
2. 仮想環境の有効化
conda activate scraping
作成した scraping 仮想環境に「入る」コマンドです。これを実行することで、以降の操作がこの環境内で行われるようになります。
仮想環境を有効にしないと、インストールしたライブラリが意図した環境に入らない可能性があります。
3. 必要なライブラリのインストール①
pip install pandas requests beautifulsoup4 selenium
スクレイピングに必要な基本ライブラリをインストールします。
| ライブラリ | 説明 |
|---|---|
pandas |
表形式のデータを扱う(CSVの読み書きなど) |
requests |
WebページにアクセスしてHTMLを取得する |
beautifulsoup4 |
HTML構造を解析し、特定の要素を取り出す |
selenium |
ブラウザを自動で開いて操作する(ログインなど) |
4. ChromeDriverのインストール(Macユーザー向け)
brew install chromedriver
このコマンドは Mac のユーザーが selenium を使うために必要なドライバ chromedriver をインストールします。
brewはMac用のパッケージ管理ソフトです。chromedriverは、SeleniumがGoogle Chromeを操作するために必要な「仲介役」です。- 注意:Chrome本体のバージョンとドライバのバージョンを合わせる必要があります。
Windowsの場合は、公式サイトからChromeDriverをダウンロードしてパスを通す必要があります。
5. 必要なライブラリのインストール②
pip install selenium pandas tqdm
一部重複していますが問題ありません(すでに入っていればスキップされます)。ここでは tqdm が新しく登場します。
tqdm:進捗バーを表示するライブラリ。大量のページをスクレイピングするときに、どれくらい処理が進んでいるかを可視化できます。
スクレイピングによるCIDの取得
コード全文
CID(Compound ID)とは、PubChemで化合物を一意に識別するための番号です。次にこれを取得し、その後にAPIで化合物の情報を取得していきます。スクレイピングのやりすぎはサーバーに負荷がかかるので、 やりすぎには注意して下さい。時間をおくなどの対応をお願いします。
from selenium import webdriver
from selenium.webdriver.common.by import By
from selenium.webdriver.support.ui import WebDriverWait
from selenium.webdriver.support import expected_conditions as EC
import time
driver = webdriver.Chrome()
#化合物によりここのURLを変更する
search_url = "https://pubchem.ncbi.nlm.nih.gov/taxonomy/65409#section=Natural-Products"
driver.get(search_url)
wait = WebDriverWait(driver, 10)
all_cids = set()
page = 1
while True:
print(f"\n📄 ページ {page} の処理中...")
try:
natural_products_section = wait.until(
EC.presence_of_element_located((By.ID, "Natural-Products"))
)
rows = natural_products_section.find_elements(By.XPATH, ".//div[contains(@class, 'sm:table-row')]")
for i, row in enumerate(rows):
try:
link = row.find_element(By.XPATH, ".//a[contains(@href, '/compound/')]")
cid = link.get_attribute("href").split("/")[-1]
if cid not in all_cids:
print(f"✅ Row {i}: CID = {cid}")
all_cids.add(cid)
except Exception as e:
print(f"❌ Row {i} エラー: {e}")
except Exception as e:
print(f"❌ Natural Products セクションが見つかりません: {e}")
break
# 次ページボタンを「Natural-Products」セクション内だけから取得してクリック
try:
next_button = WebDriverWait(natural_products_section, 5).until(
EC.element_to_be_clickable((
By.XPATH, ".//button[.//span[text()='Next'] and not(@disabled)]"
))
)
next_button.click()
page += 1
time.sleep(2)
except Exception:
print("⛔️ 次のページが存在しないか、ボタンが無効になっています。終了します。")
break
driver.quit()
print(f"\n🔚 全取得CID数: {len(all_cids)} 件")
from selenium import webdriver
from selenium.webdriver.common.by import By
from selenium.webdriver.support.ui import WebDriverWait
from selenium.webdriver.support import expected_conditions as EC
import time
driver = webdriver.Chrome()
#化合物によりここのURLを変更する
search_url = "https://pubchem.ncbi.nlm.nih.gov/taxonomy/65409#section=Natural-Products"
driver.get(search_url)
wait = WebDriverWait(driver, 10)
all_cids = set()
page = 1
while True:
print(f"\n📄 ページ {page} の処理中...")
try:
natural_products_section = wait.until(
EC.presence_of_element_located((By.ID, "Natural-Products"))
)
rows = natural_products_section.find_elements(By.XPATH, ".//div[contains(@class, 'sm:table-row')]")
for i, row in enumerate(rows):
try:
link = row.find_element(By.XPATH, ".//a[contains(@href, '/compound/')]")
cid = link.get_attribute("href").split("/")[-1]
if cid not in all_cids:
print(f"✅ Row {i}: CID = {cid}")
all_cids.add(cid)
except Exception as e:
print(f"❌ Row {i} エラー: {e}")
except Exception as e:
print(f"❌ Natural Products セクションが見つかりません: {e}")
break
# 次ページボタンを「Natural-Products」セクション内だけから取得してクリック
try:
next_button = WebDriverWait(natural_products_section, 5).until(
EC.element_to_be_clickable((
By.XPATH, ".//button[.//span[text()='Next'] and not(@disabled)]"
))
)
next_button.click()
page += 1
time.sleep(2)
except Exception:
print("⛔️ 次のページが存在しないか、ボタンが無効になっています。終了します。")
break
driver.quit()
print(f"\n🔚 全取得CID数: {len(all_cids)} 件")
コードの詳細説明
使用ライブラリとその役割
from selenium import webdriver
from selenium.webdriver.common.by import By
from selenium.webdriver.support.ui import WebDriverWait
from selenium.webdriver.support import expected_conditions as EC
import time
selenium: WebブラウザをPythonで自動操作するための主要ライブラリ。webdriver.Chrome(): Chromeブラウザを操作するためのドライバを起動。WebDriverWait: 条件が満たされるまで指定時間待機する機能(例:要素の読み込み)。expected_conditions as EC: 具体的な待機条件を指定するためのモジュール(例:要素のクリック可能状態)。time: 手動での待機処理に使用(例:ページ遷移後の短い遅延)。
対象ページへのアクセス
driver = webdriver.Chrome()
search_url = "https://pubchem.ncbi.nlm.nih.gov/taxonomy/65409#section=Natural-Products"
driver.get(search_url)
- PubChemの該当ページへアクセスし、「Natural Products」セクションを表示。
taxonomy/65409はScutellaria baicalensis(オウゴン)のTaxonomy ID。#section=Natural-Productsで対象セクションにスクロール。
初期設定(CID記録用セットとページ番号)
all_cids = set()
page = 1
all_cids:重複を避けるための集合型データ構造(set())。page:現在処理しているページ番号を管理。
メインループ(ページを繰り返し処理)
while True:
print(f"\n📄 ページ {page} の処理中...")
- 無限ループでページを順番に処理。
- 処理中のページ番号をログとして出力。
Natural Products セクションの取得と行の抽出
natural_products_section = wait.until(
EC.presence_of_element_located((By.ID, "Natural-Products"))
)
rows = natural_products_section.find_elements(By.XPATH, ".//div[contains(@class, 'sm:table-row')]")
wait.untilで「Natural Products」セクションがDOM上に現れるまで最大10秒待機。- セクション内の
table-row相当の行(化合物情報)をXPathで抽出。
各行からCIDの抽出
for i, row in enumerate(rows):
try:
link = row.find_element(By.XPATH, ".//a[contains(@href, '/compound/')]")
cid = link.get_attribute("href").split("/")[-1]
if cid not in all_cids:
print(f"✅ Row {i}: CID = {cid}")
all_cids.add(cid)
except Exception as e:
print(f"❌ Row {i} エラー: {e}")
- 各行に含まれるリンクから
/compound/を含むURLを取得。 - CID(PubChem Compound ID)はリンク末尾の数字部分。
- 初出のCIDのみ記録(
setを利用)し、重複を回避。
Natural Products セクション内の「Next」ボタンをクリック
try:
next_button = WebDriverWait(natural_products_section, 5).until(
EC.element_to_be_clickable((By.XPATH, ".//button[.//span[text()='Next'] and not(@disabled)]"))
)
next_button.click()
page += 1
time.sleep(2)
natural_products_section内からのみNextボタンを探すため、他セクションのボタンと誤って干渉しない。not(@disabled)を指定して、無効な(= 最終ページの)ボタンを除外。- ページ遷移後の読み込みを待つために
time.sleep(2)を挿入。
次ページが存在しない場合の処理
except Exception:
print("⛔️ 次のページが存在しないか、ボタンが無効になっています。終了します。")
break
Nextボタンが見つからない、または無効(disabled)な場合はbreakでループ終了。
処理終了後の後始末と結果出力
driver.quit()
print(f"\n🔚 全取得CID数: {len(all_cids)} 件")
- Webブラウザを自動終了し、メモリ解放。
- 重複除去されたCIDの総数を表示。
APIを使ったSMILESの取得
ここでは先ほど取得したCIDを使って、APIで化合物情報を取得し、csvとして保存する処理を行います。
まず以下を使って、pubchempyをインストールしておいて下さい。
!pip install pubchempy
コード全文
import csv
import time
import pubchempy as pcp
output_file = "compound_data.csv"
compound_data = []
cid_list = sorted(all_cids) # ← 事前に定義されたCIDリストまたは集合
for i, cid in enumerate(cid_list):
try:
# ConnectivitySMILES と IUPACName を明示的に指定して取得
props_list = pcp.get_properties(['ConnectivitySMILES', 'IUPACName'], cid, namespace='cid')
if props_list:
props = props_list[0]
smiles = props.get('ConnectivitySMILES', 'NOT_FOUND')
name = props.get('IUPACName', 'NOT_FOUND')
else:
smiles = "NOT_FOUND"
name = "NOT_FOUND"
compound_data.append({
"CID": cid,
"SMILES": smiles,
"IUPACName": name
})
print(f"✅ {i+1}/{len(cid_list)} CID {cid} → ConnectivitySMILES: {smiles} → IUPACName: {name}")
except Exception as e:
print(f"❌ {i+1}/{len(cid_list)} CID {cid} → エラー: {e}")
compound_data.append({
"CID": cid,
"SMILES": "ERROR",
"IUPACName": "ERROR"
})
time.sleep(0.2) # API負荷軽減
# CSVとして保存
if compound_data:
with open(output_file, "w", newline="", encoding="utf-8") as f:
writer = csv.DictWriter(f, fieldnames=["CID", "SMILES", "IUPACName"])
writer.writeheader()
writer.writerows(compound_data)
print(f"\n📄 完了: {output_file} に {len(compound_data)} 件のデータを保存しました。")
else:
print("⚠️ データが空なので、CSVは作成されませんでした。")
詳細コード
必要なモジュールのインポート
import csv
import time
import pubchempy as pcp
csv:CSVファイルへの保存に使います。time:APIへのアクセスの間隔を調整するために使用。pubchempy:PubChem REST API を使いやすくするPythonラッパー。
初期設定
output_file = "compound_data.csv"
compound_data = []
cid_list = sorted(all_cids) # all_cids は事前に定義された CID の集合またはリスト
- 出力ファイル名を定義。
- 取得した化合物データを保存するリスト
compound_dataを初期化。 all_cids(取得済みのCID一覧)をソート。
各CIDに対して情報取得ループ
for i, cid in enumerate(cid_list):
各CIDに対して以下の処理を順に行います:
PubChem APIから情報取得
props_list = pcp.get_properties(['ConnectivitySMILES', 'IUPACName'], cid, namespace='cid')
ConnectivitySMILESとIUPACNameを一度に取得します。namespace='cid'により、CIDをキーとして検索します。
成功時の処理
if props_list:
props = props_list[0]
smiles = props.get('ConnectivitySMILES', 'NOT_FOUND')
name = props.get('IUPACName', 'NOT_FOUND')
- データが取得できた場合、最初の辞書から
ConnectivitySMILESとIUPACNameを取り出します。 - 取得できなければ
'NOT_FOUND'を代入。
データを保存
compound_data.append({
"CID": cid,
"SMILES": smiles,
"IUPACName": name
})
- CID、SMILES、IUPAC名を辞書にしてリストへ追加。
エラーハンドリング
except Exception as e:
print(f"❌ {i+1}/{len(cid_list)} CID {cid} → エラー: {e}")
compound_data.append({
"CID": cid,
"SMILES": "ERROR",
"IUPACName": "ERROR"
})
- 例外発生時には
"ERROR"として記録。 - これにより、処理が途中で止まらず、すべてのCIDを処理できます。
APIアクセスの間隔
time.sleep(0.2)
- PubChem APIへのアクセス頻度を制限(1秒間に5回=安全圏)。
結果をCSVファイルに保存
with open(output_file, "w", newline="", encoding="utf-8") as f:
writer = csv.DictWriter(f, fieldnames=["CID", "SMILES", "IUPACName"])
writer.writeheader()
writer.writerows(compound_data)
compound_dataリストの内容をCSVとして保存。- UTF-8で保存するので日本語でも問題なし。
データがなかった場合の処理
else:
print("⚠️ データが空なので、CSVは作成されませんでした。")
compound_dataが空だった場合はCSVファイルを作成しない。
化合物のフィルタリング
上記で得られた化合物をdrug-likeness(薬になりやすさ)に基づいてフィルタリングします。
コード全文
#Filtering
from rdkit import Chem
from rdkit.Chem import Descriptors
import pandas as pd
# データフレームに読み込み直す(または compound_data を使ってもOK)
df = pd.DataFrame(compound_data)
df = df[df["SMILES"] != "ERROR"]
# プロパティ計算関数
def calculate_properties(smiles):
mol = Chem.MolFromSmiles(smiles)
if mol is None:
return None
return {
"MW": Descriptors.MolWt(mol),
"LogP": Descriptors.MolLogP(mol),
"HBD": Descriptors.NumHDonors(mol),
"HBA": Descriptors.NumHAcceptors(mol),
"TPSA": Descriptors.TPSA(mol),
"RotB": Descriptors.NumRotatableBonds(mol),
"RingCount": Chem.rdMolDescriptors.CalcNumRings(mol)
}
# 各ルール判定
def passes_lipinski(p): return p["MW"] < 500 and p["LogP"] < 5 and p["HBD"] <= 5 and p["HBA"] <= 10
def passes_veber(p): return p["TPSA"] <= 140 and p["RotB"] <= 10
def passes_egan(p): return p["TPSA"] <= 131 and p["LogP"] <= 5.88
def passes_muegge(p): return (200 <= p["MW"] <= 600 and -2 <= p["LogP"] <= 5 and p["HBD"] <= 5 and
p["HBA"] <= 10 and p["TPSA"] <= 150 and p["RingCount"] <= 7 and p["RotB"] <= 15)
# 判定+結果格納
results = []
for _, row in df.iterrows():
props = calculate_properties(row["SMILES"])
if props:
results.append({
**row,
**props,
"Lipinski": passes_lipinski(props),
"Veber": passes_veber(props),
"Egan": passes_egan(props),
"Muegge": passes_muegge(props)
})
df_results = pd.DataFrame(results)
# すべてのルールを満たす化合物を抽出
df_filtered = df_results[df_results[["Lipinski", "Veber", "Egan", "Muegge"]].all(axis=1)]
# 保存
df_results.to_csv("compound_with_DL_rules.csv", index=False)
df_filtered.to_csv("compound_DL_filtered.csv", index=False)
print(f"\n✅ Drug-likenessルール通過化合物を保存しました:")
print(f" - 全体データ: compound_with_DL_rules.csv")
print(f" - フィルタ後 : compound_DL_filtered.csv({len(df_filtered)} 件)")
コード詳細
使用ライブラリ
from rdkit import Chem
from rdkit.Chem import Descriptors
import pandas as pd
rdkit: 化学構造の操作や記述子計算を行うPythonライブラリ(創薬やケモインフォマティクス分野で必須)pandas: データフレーム操作(CSVファイルとの連携やフィルタリングなど)に使用
SMILESデータをDataFrameとして読み込み
df = pd.DataFrame(compound_data)
df = df[df["SMILES"] != "ERROR"]
compound_dataはSMILES取得で作成されたリスト- エラーが含まれる化合物(SMILES = “ERROR”)は除外します
RDKitを使って分子記述子を計算
def calculate_properties(smiles):
mol = Chem.MolFromSmiles(smiles)
if mol is None:
return None
return {
"MW": Descriptors.MolWt(mol),
"LogP": Descriptors.MolLogP(mol),
"HBD": Descriptors.NumHDonors(mol),
"HBA": Descriptors.NumHAcceptors(mol),
"TPSA": Descriptors.TPSA(mol),
"RotB": Descriptors.NumRotatableBonds(mol),
"RingCount": Chem.rdMolDescriptors.CalcNumRings(mol)
}
- SMILESから分子構造を生成し、以下の記述子を取得:
MW: 分子量LogP: 脂溶性(疎水性)HBD: 水素供与体の数HBA: 水素受容体の数TPSA: 極性表面積RotB: 回転可能結合の数RingCount: 環の数
代表的なDrug-likenessルールを関数化
def passes_lipinski(p): ...
def passes_veber(p): ...
def passes_egan(p): ...
def passes_muegge(p): ...
- LipinskiのRule of 5:
- MW < 500, LogP < 5, HBD ≤ 5, HBA ≤ 10
- Veberルール:
- TPSA ≤ 140, RotB ≤ 10
- Eganルール:
- TPSA ≤ 131, LogP ≤ 5.88
- Mueggeルール:
- 幅広い物理化学的条件を組み合わせた厳しめのルール
全化合物に対して記述子計算とルール評価を実行
results = []
for _, row in df.iterrows():
props = calculate_properties(row["SMILES"])
if props:
results.append({
**row,
**props,
"Lipinski": passes_lipinski(props),
"Veber": passes_veber(props),
"Egan": passes_egan(props),
"Muegge": passes_muegge(props)
})
- 各化合物に対して記述子を計算し、各ルールを
True/Falseで評価 - ルールの結果をDataFrameに追加
評価結果を保存
df_results = pd.DataFrame(results)
df_filtered = df_results[df_results[["Lipinski", "Veber", "Egan", "Muegge"]].all(axis=1)]
df_results.to_csv("compound_with_DL_rules.csv", index=False)
df_filtered.to_csv("compound_DL_filtered.csv", index=False)
compound_with_DL_rules.csv:- 各化合物の記述子と各ルールの合否を含む全データ
compound_DL_filtered.csv:- すべてのルールに合格した「drug-like」な化合物のみを抽出
実行結果の出力
print(f"\n✅ Drug-likenessルール通過化合物を保存しました:")
print(f" - 全体データ: compound_with_DL_rules.csv")
print(f" - フィルタ後 : compound_DL_filtered.csv({len(df_filtered)} 件)")
終了すると、全102件の化合物が取得できていると思います!
最後に
本記事では、Network pharmacology(ネットワーク薬理学)という考え方をベースに、漢方薬「黄芩(Scutellaria baicalensis)」の有効成分をPubChemから取得し、drug-likeness(薬になりやすさ)に基づくフィルタリングを行うまでの一連の流れをご紹介しました。
特に、
- スクレイピングによるCIDの取得
- PubChem APIを用いたSMILES・IUPAC名の抽出
- RDKitによるLipinskiやVeberなどのフィルタリング
といったステップは、誰でも再現可能なスクリプト形式で紹介しましたので、研究や業務の中でもすぐに応用いただけると思います。次回は化合物から標的タンパク質の予測を行っていきます!
自宅でできるin silico創薬の技術書を販売中
新薬探索を試したい方必読!
ITエンジニアである著者の視点から、wetな研究者からもdryの創薬研究をわかりやすく身近に感じられるように解説しています
自宅でできるin silico創薬の技術書を販売中
タンパク質デザイン・モデリングに焦点を当て、初めてこの分野に参入する方向けに、それぞれの手法の説明から、環境構築、実際の使い方まで網羅!










