今回は前回の記事でRFPeptideで作成した環状ペプチド骨格に対して、その骨格に一致するアミノ酸配列をProteinMPNNで探索していきます。また、MPNNで予測したアミノ酸配列の構造をAlphaFold2を利用して確認することまで行います。
この記事を読むことで、ProteinMPNNを最新のアミノ酸配列推定方法とAlphaFold2の実行方法を知ることが出来ます。
【この記事のまとめ】
本記事では、創薬研究者やバイオインフォマティクス初学者に向けて、最先端の配列設計AI「ProteinMPNN」を駆使して、高い構造安定性を持つ環状ペプチドの配列を精密にデザインする方法を解説します。
- 高精度な配列最適化:骨格構造(バックボーン)に最適なアミノ酸配列を数秒で生成し、設計の成功率を従来の物理化学的手法よりも大幅に向上させます。
- 環状構造への特化設定:N末端とC末端を連結させる環状ペプチド特有のパラメータ設定や、計算を効率化するための「タイリング(Tying)」の手法を習得できます。
- 実践的な実行フロー:Google Colab等での環境構築から、実際のコマンド入力、設計結果の評価まで、ステップバイステップで詳しくガイドします。
この記事を読むことで、最新のAI技術を自身の研究に取り入れ、標的タンパク質に強く結合する「次世代の環状ペプチド候補」を効率的に創出できるようになります。
Windows 11, WSL2(Ubuntu 20.04),bash(Micromamba )
PCスペック
CPUメモリ: 16GB
GPU: NVIDIA GeForce RTX 2070 SUPER(8GB)
GPUドライバー: 581.29-desktop-win10-win11-64bit-international-dch-whql
自宅でできるin silico創薬の技術書を販売中
新薬探索を試したい方必読!
ITエンジニアである著者の視点から、wetな研究者からもdryの創薬研究をわかりやすく身近に感じられるように解説しています
自宅でできるin silico創薬の技術書を販売中
タンパク質デザイン・モデリングに焦点を当て、初めてこの分野に参入する方向けに、それぞれの手法の説明から、環境構築、実際の使い方まで網羅!
ProteinMPNNとは?
ProteinMPNN は、タンパク質の立体構造(バックボーン座標)からアミノ酸配列を設計するための深層学習モデルです。簡単に言うと、「構造からその構造にマッチする最適な配列を予測するAI」です。
Message Passing Neural Network (MPNN)を用いることで、従来法(Rosetta Design等)に比べ、速く、正確で、構造に忠実な配列設計ができる可能性があるツールです。こちらも前回のRFPeptide同様GPUを利用するツールです。もしもGPUドライバーがインストールされていない場合は、前回の記事を参考にインストールしておきましょう。
以下で、少しだけこのツールの仕組みを深堀します。MPNNとはグラフ構造データを扱うためのニューラルネットワークの一種で、簡単に言うと、ノード(点)とエッジ(線)で表される構造情報をもとに、各ノードの状態を更新して特徴を学習する仕組みです。 このMPNNで、ノードをアミノ酸、エッジを残基間の距離や結合に見立てて、グラフ構造としてタンパク質の立体構造を扱うことで、遠くのアミノ酸から付近のアミノ酸までを自然な形で重みをつけて考慮することが可能になっています。
ProteinMPNNでは、このMPNNによってアミノ酸配列と立体構造の関係が学習されており、これをタンパク質設計に応用しています。
ProteinMPNNの実装方法
では早速、ProteinMPNNでこちらの記事で作成した環状ペプチド骨格に最適なアミノ酸配列を予測していきます。
こちらを参考に環境構築から行っていきます。今回もWindowsのWSLを利用していきます。WSLの利用方法がわからない場合は、こちらの記事に記載してあるのでそちらを参考にしてください。
前回の記事と同様に、環境構築はmicromambaで行います。WSLで以下を実行してください。途中で [Y/n] の入力を求められたら、Yを入力してください。
# GitHub から ProteinMPNN をクローン
git clone https://github.com/dauparas/ProteinMPNN.git
cd ProteinMPNN
# micromambaで環境を用意
micromamba create -n mlfold python=3.10 -y
# 環境の有効化
micromamba activate mlfold
# 必要なパッケージをインストール
micromamba install pytorch torchvision torchaudio cudatoolkit=11.3 -c pytorch
# 学習済みのモデルをダウンロード
wget https://github.com/dauparas/ProteinMPNN/blob/main/vanilla_model_weights/v_48_002.pt
wget https://github.com/dauparas/ProteinMPNN/blob/main/vanilla_model_weights/v_48_010.pt
wget https://github.com/dauparas/ProteinMPNN/blob/main/vanilla_model_weights/v_48_020.pt
wget https://github.com/dauparas/ProteinMPNN/blob/main/vanilla_model_weights/v_48_030.ptプログラムを実行する
これでProteinMPNNを実行する用意が整いました。実際にこれから実行をしていきます。
私は前回の記事で作成したdesign_0.pdbファイルの構造に対して、ProteinMPNNを実行します。そのため、pdbファイルを保存していたフォルダ内にMPNNoutputというフォルダを作成し、そのフォルダにProteinMPNNの結果が保存されるように指定して実行しました。もしも、別のファイルを対象にProteinMPNNを実行したい場合や、別の保存場所を利用したい場合は、--pdb_pathと--out_folder に関して、ご自身の保存場所や指定したいファイルに合わせて変更してください。
前回の記事を参考に進めていただいている場合は、上記のパス設定はそのままで、アウトプットの保存先として、/Documents/RFPeptide/samplesフォルダ内にMPNNoutputという名前のフォルダを作成してから、以下を実行してください。
# ProteinMPNNを実行
python ProteinMPNN/protein_mpnn_run.py \ --pdb_path documents/RFdiffusion/samples/design_0.pdb \ --pdb_path_chains "B" \ --out_folder /documents/RFdiffusion/samples/MPNNoutput \ --model_name v_48_020 \ --num_seq_per_target 2 \ --sampling_temp 0.1 \ --seed 42 \ --batch_size 1実行結果
MPNNoutputフォルダに/documents/RFPeptide/samples/MPNNoutputdesign_0.faseqsというフォルダが出来、その中にdesign_0.faというファイルができてその中身がいれば成功です!
このファイルはメモ帳などで開くことができます。実際に確認してみると以下の様になっていました。指定通りに、“ESGIIDIETGERIP”と“ESGIIDIETGEKIP”の2種類のアミノ酸配列が予想されたことが分かります。ESGIIDIETGERIPこのファイルをメモ帳などで開くと以下の様になっているはずです。この3行目以降がProteinMPNNで予測された、骨格構造に沿ったアミノ酸配列です。指定通りに2種類の配列が予測されました。
design_0, score=3.7456, global_score=1.9113, fixed_chains=['B'], designed_chains=['A'], model_name=v_48_020, git_hash=8907e6671bfbfc92303b5f79c4b5e6ce47cdef57, seed=42
GGGGGGGGGGGGGG
T=0.1, sample=1, score=0.8211, global_score=1.6212, seq_recovery=0.1429
ESGIIDIETGERIP
T=0.1, sample=2, score=0.7738, global_score=1.6172, seq_recovery=0.1429
ESGIIDIETGEKIPAlphaFold2での実行結果の確認
では、ここからAlphaFold2でのMPNNの出力物の確認を行っていきます。AlphaFold2を自身のPCで実行するのは要求されるPCスペックが非常に高いことから現実的ではないのですが、今回はブラウザ環境でAlphaFold2が実行できるGoogleColab環境を利用することで実行していきます。また、通常のAlphaFoldは環状ペプチドの構造予測には対応していないので、GoogleColabを使って環状ペプチドの構造予測にAlphaFoldを対応させてあるこちらで公開されているツールを利用していきます。
先ずはこちらのリンクへ移動してください。ここから先ほどのツールによるGoogleColabでのAlphaFold2による環状ペプチドの構造予測を簡単に行うことが出来ます。リンク先のブラウザ画面からは環状ペプチドの構造予測の設定をいじることが出来ますが、今回変更が必要なのは3か所のみです。
先ずは、①**query_sequence:** のカラム内に今回ProteinMPNNで作成した配列を入力してください。さらに②**jobname:** カラム内はアウトプットファイルの名前を指定できるので、お好きな名前を入力してください。
私は**query_sequence:** にはMPNNが予測した1番目の配列”ESGIIDIETGERIP”を、jobname: には”Sample1”と入力しておきました。
最後に、③ページを下にスクロールしたところにある**Save settings**のチェックボックスにチェックをつけて、実行結果を保存できるようにしておきましょう。(上記の設定箇所はスクリーンショット内に赤枠でマークしてありますので、見つかりにくい場合の参考にしてください。)
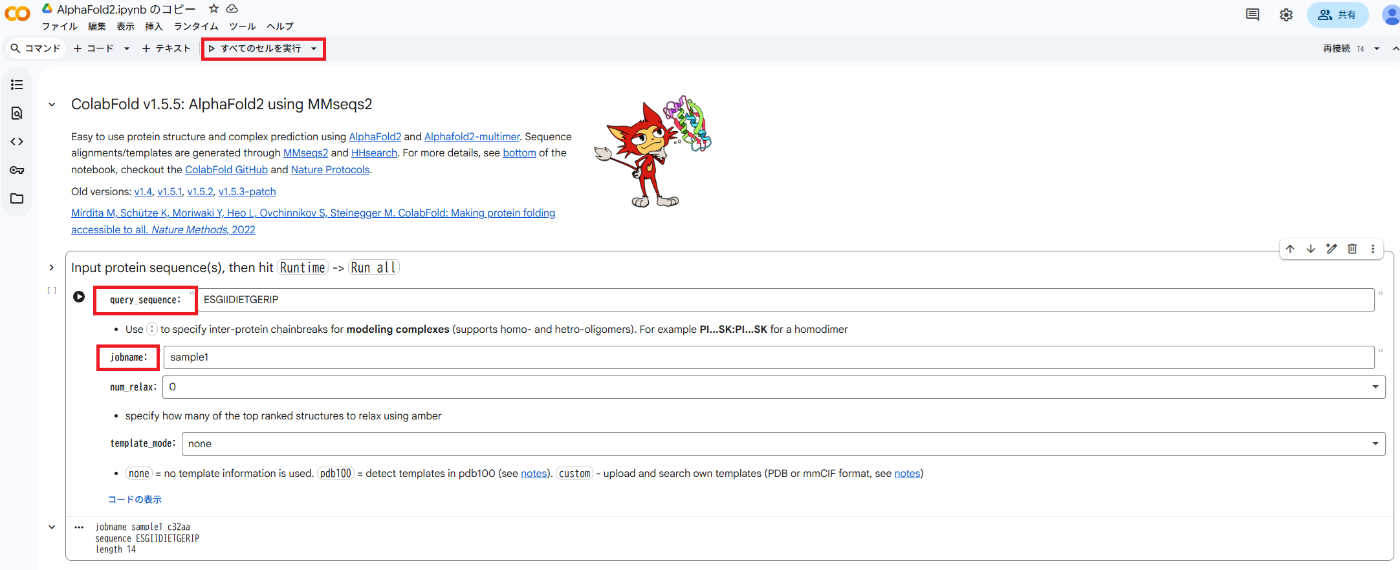
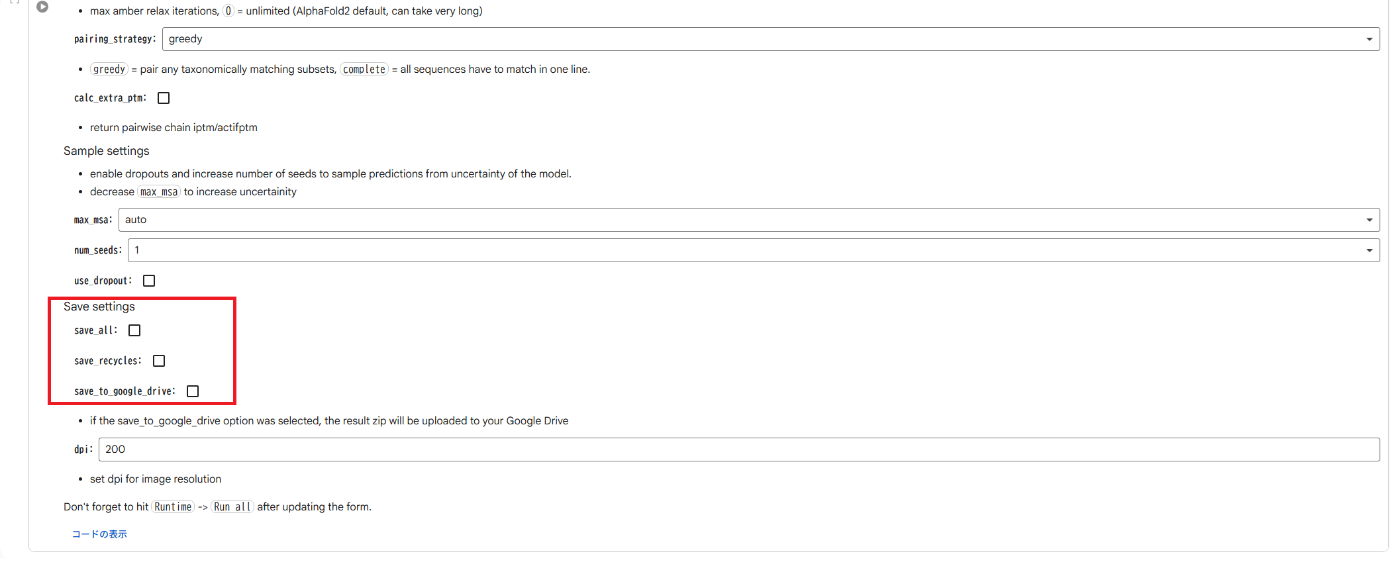
上記の設定が終わったらページ左上の”全てのセルを実行”(スクリーンショット1枚目の赤枠)をクリックすると、AlphaFold2が実行できます。この時に以下の警告が表示されると思いますが、気にせず実行してしまって構いません。

すると、しばらくするとページをスクロールした先に、このように予測された立体構造が表示されます。
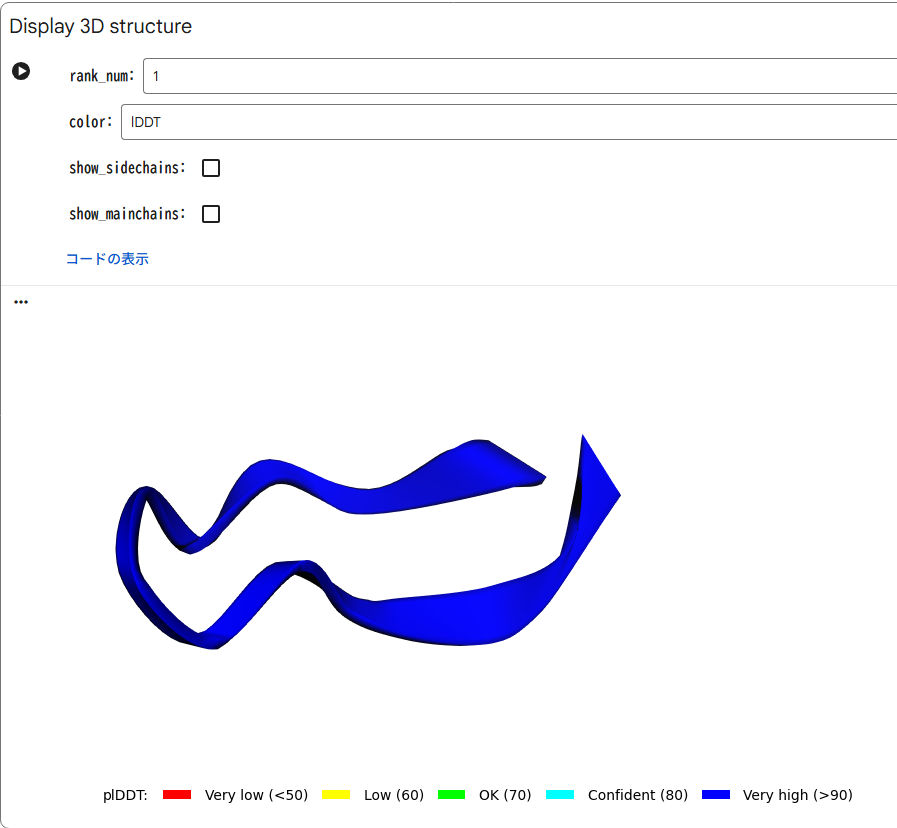
今回のアミノ酸配列がplDDTが90–100という高い精度で、狙い通りの環状ペプチド構造を形造ることが確認できました。
上記は切れているように見えますが、Pymolで読み込み、Sticksで表示すると、きちんとした環状ペプチドになっています!
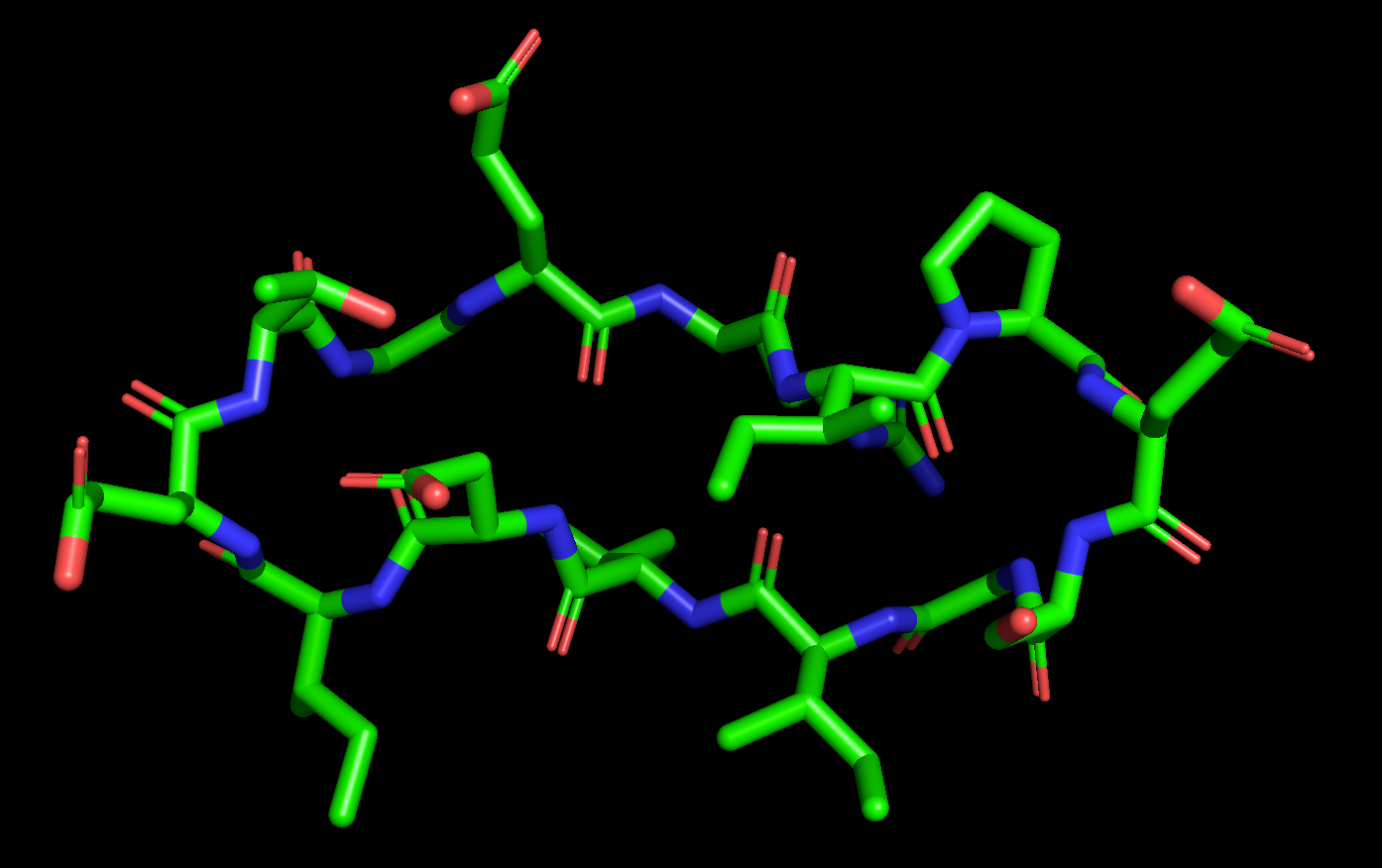
コードの解説
上に書いたソースコードの解説をしていきます。
# GitHub から ProteinMPNN をクローン
git clone https://github.com/dauparas/ProteinMPNN.git
cd ProteinMPNN
# micromambaで環境を用意
micromamba create -n mlfold python=3.10 -y
# 環境の有効化
micromamba activate mlfold
# 必要なパッケージをインストール
micromamba install pytorch torchvision torchaudio cudatoolkit=11.3 -c pytorch
# 学習済みのモデルをダウンロード
wget https://github.com/dauparas/ProteinMPNN/blob/main/vanilla_model_weights/v_48_002.pt
wget https://github.com/dauparas/ProteinMPNN/blob/main/vanilla_model_weights/v_48_010.pt
wget https://github.com/dauparas/ProteinMPNN/blob/main/vanilla_model_weights/v_48_020.ptgit cloneは、GitHub にあるプロジェクト(この場合は ProteinMPNN)を自分のパソコンにコピーするコマンドです。https://github.com/dauoaras/ProteinMPNN.gitはコピー元の URL です。実行すると、カレントディレクトリ(今いるフォルダ)に ProteinMPNN という名前のフォルダが作られ、その中にプロジェクトのファイルが全部入ります。
イメージ: 「インターネット上の本棚(GitHub)から、本を丸ごと借りて自分の机に置く」ような感じです。
cd ProteinMPNNで先ほどクローンしたディレクトリへ移動します。micromamba create -n mlfold python=3.10 -yでは、micromambaを使って、mfoldという名前の環境を作成し、pythonをインストールしています。環境の名前はなんでもOKです。micromamba activate mlfoldで先ほど作った環境を有効化します。これで先ほどインストールしたpythonが入っている環境が立ち上がりました。micromamba install pytorch torchvision torchaudio cudatoolkit=11.3 -c pytorchで、立ち上がった環境内にさらに必要なパッケージをインストールしていきます。wgetはオンライン上のファイルをダウンロードするコマンドです。ここではGitHub上にある、ProteinMPNNで利用する学習済みモデルの重みを3種類ダウンロードしています。これでProtein MPNNが利用可能になりました。
# ProteinMPNNを実行
python ProteinMPNN/protein_mpnn_run.py \ --pdb_path documents/RFdiffusion/samples/design_0.pdb \ --pdb_path_chains "B" \ --out_folder /documents/RFdiffusion/samples/MPNNoutput \ --model_name v_48_020 \ --num_seq_per_target 2 \ --sampling_temp 0.1 \ --seed 42 \ --batch_size 1- このコードで実際にProteinMPNNを実行しています。各オプションの意味は以下の通りです。
-pdb_path:デザインの元になるタンパク質構造(PDBファイル)の場所を指定。
-pdb_path_chains "B" :PDBの中のどのチェーンをデザイン対象にするか指定。
今回のファイルには、環状ペプチドと結合ターゲットとなるタンパク質の2種類の鎖が含まれているため、環状ペプチドのチェーンの方(私のファイルの場合はB)を指定した。
-out_folder :MPNN が生成した配列をどこに保存するかを指定。
-model_name :使いたい学習済みモデルの重みを指定。
- ProteinMPNN には複数モデルがあり、どのモデルを利用するかを指定できる。
今回はvanilla_model_weights/v_48_020.ptで一般的な ProteinMPNN の基本モデルを利用。その他には、水溶性の大型タンパク質のためのモデルや、より簡易的な予測をするための軽量モデルなどが選択できる。
-num_seq_per_target 2 :1つの構造に対して何本のアミノ酸配列を生成するかを指定。今回は、簡易に2本の配列を予測するように指定。多すぎても、計算に時間がかかる為、10本ほど予測させておくと良いかもしれない。
-sampling_temp 0.1 :生成モデルの温度パラメーターを指定する。温度パラメーターとは、“ランダム性(探索の広さ)” を決めるパラメータ。
- 0.1:保守的(モデルがアミノ酸配列を1アミノ酸ずつ予測する際に、より確率の高いもアミノ酸候補の中から選択する)
- 1.0:ランダム性高い(比較的確率が低いアミノ酸も選択肢に入れて、アミノ酸候補を選択する。予想されるアミノ酸配列に多様な配列が生まれる。)
最適な数値は、実際に試しながら探っていく必要があるが、今回の用途で使い場合は0.1~0.3ぐらいの保守的な予測が適切な可能性が高い。
-seed 42 :実行ごとのランダム性を固定するためのシード設定。今回のような生成モデルは、たとえ同様のpdbファイルを対象にした場合でも実行するごとに予測結果が変動する。シード設定として適当な値を設定しておくことで、再度プログラムを実行したときに前回と同様のシード値を設定することで、前回と同じ結果を予測することができる。予測ファイルを誤って消去してしまった際などや、結果の再現性を確保したい際のために設定しておくと便利。
-batch_size 1 :一度にいくつの構造を処理するかを指定する。余程の高性能マシンでない限り、 1 のままでOK。
最後に
ProteinMPNNの解説は以上になります。相変わらずGPUは必要ですが、ツール自体は意外と簡単に利用できたのではないでしょうか。AlphaFoldも加えて、構造もきちんと確認できるととても面白いですね!
参考文献
Protein MPNN
https://github.com/dauparas/ProteinMPNN?tab=MIT-1-ov-file MIT License
AlphaFold2
https://github.com/google-deepmind/alphafold?tab=Apache-2.0-1-ov-file Apache License
環状ペプチド用のAlphaFold2
くろたんくLab様より開発
https://colab.research.google.com/github/blacktanktoplab/ColabFold/blob/cyclic/AlphaFold2.ipynb
自宅でできるin silico創薬の技術書を販売中
新薬探索を試したい方必読!
ITエンジニアである著者の視点から、wetな研究者からもdryの創薬研究をわかりやすく身近に感じられるように解説しています
自宅でできるin silico創薬の技術書を販売中
タンパク質デザイン・モデリングに焦点を当て、初めてこの分野に参入する方向けに、それぞれの手法の説明から、環境構築、実際の使い方まで網羅!










