本記事は、ファーマコフォアを用いて化合物を効率的に探索するツール、Pharmitについての解説です。創薬において、タンパク質の特定の機能(例えば、酵素活性を阻害する、特定の受容体に結合するなど)を誘発する化合物を探索する際、膨大な数の化合物ライブラリの中から、有望な候補を絞り込む必要があります。ファーマコフォアをうまく使用することで、有望な候補を簡単に絞ることができます。今回は、ファーマコフォアの概念から、その作成、そしてPharmitを用いた検索方法までを解説します。
Mac M1, Sequoia 15.6
自宅でできるin silico創薬の技術書を販売中
新薬探索を試したい方必読!
ITエンジニアである著者の視点から、wetな研究者からもdryの創薬研究をわかりやすく身近に感じられるように解説しています
自宅でできるin silico創薬の技術書を販売中
タンパク質デザイン・モデリングに焦点を当て、初めてこの分野に参入する方向けに、それぞれの手法の説明から、環境構築、実際の使い方まで網羅!
ファーマコフォアとは?
ファーマコフォア(Pharmacophore)とは、特定の生体分子と相互作用するために不可欠な、分子の3次元的な特徴の集合体のことです。簡単に言うと、「薬として働くために必要な、化合物の形と化学的性質のパターン」を抽象的に表現したものです。
例として、薬とタンパク質が結合する際に、水素結合の供与体・受容体、疎水性相互作用、正の電荷、負の電荷といった、特定の化学的性質を持つ原子の配置が重要になります。ファーマコフォアは、これらの「重要な特徴」と「特徴間の距離」をモデル化することで、構造が全く異なる化合物であっても、同じファーマコフォアを持つものを効率的に探し出すことを可能にします。
Pharmitとは?
Pharmitは、ファーマコフォアモデルに基づいて、化合物の探索を高速に行うためのツールです。タンパク質の立体構造情報から、リガンドが結合するポケット(結合サイト)の特徴を抽出し、その特徴を満たす化合物をデータベースから見つけ出すことができます。
Pharmitの利用法(ブラウザからの利用)
PharmitはWebサーバーとして公開されているため、環境構築なしでブラウザからすぐに利用できます。これにより、初心者でも手軽にファーマコフォア検索を体験することができます。
Pharmitを使ったファーマコフォア検索の実行
PharmitのWebサイトで、実際にファーマコフォアを作成し、化合物を探索する手順を解説します。
今回は、PDBからダウンロードしたタンパク質–リガンド複合体(例: PDB ID 2OQV、DPP4とその阻害薬)の構造を例に、リガンドの構造を基にファーマコフォアを作成します。
まず、Pharmitのサイトにアクセスします。お好きなタンパク質のPDB ID(例: PDB ID 2OQV)を入れます。その横にはリガンドの名前(ここではMA9があります)
サイト中央のSubmit ボタンをクリックします。
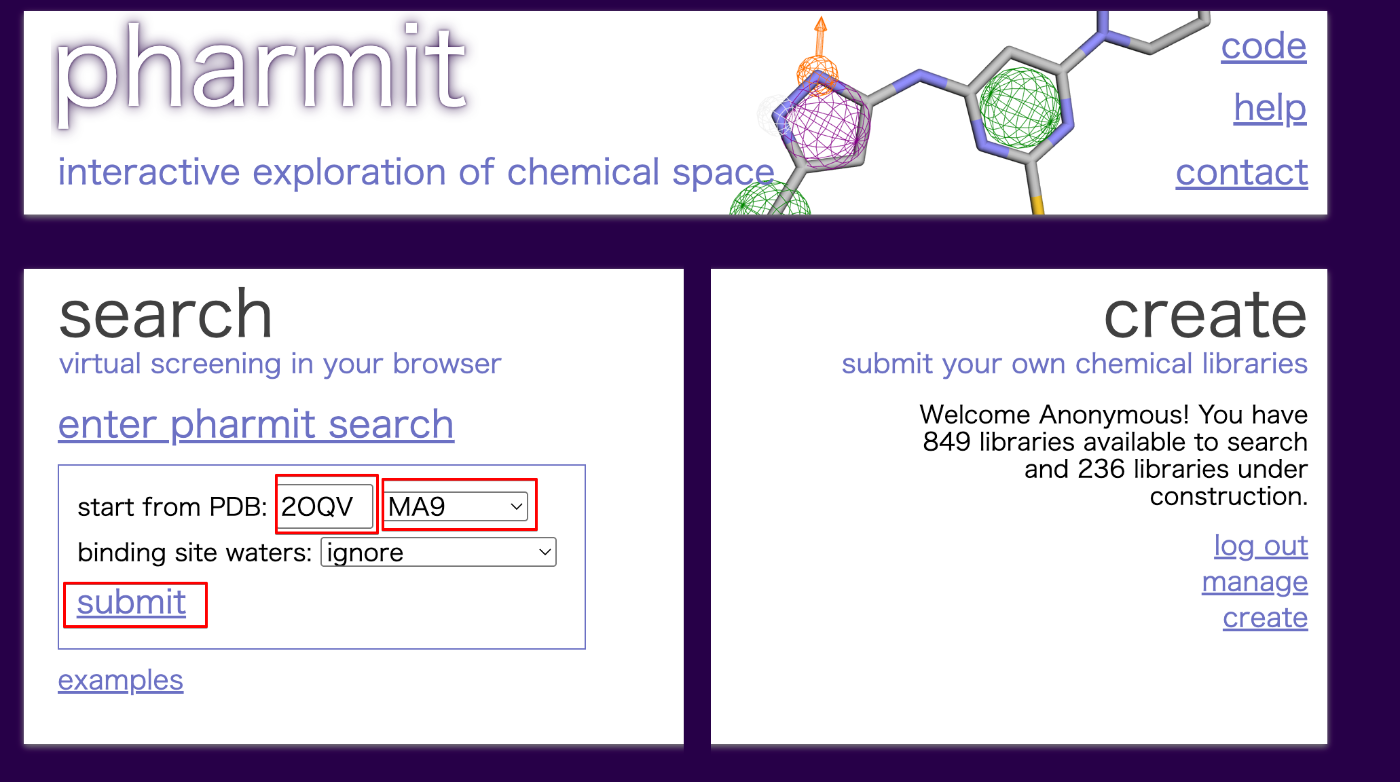
読み込みが完了すると、タンパク質とリガンド(阻害剤)の複合体が表示されます。
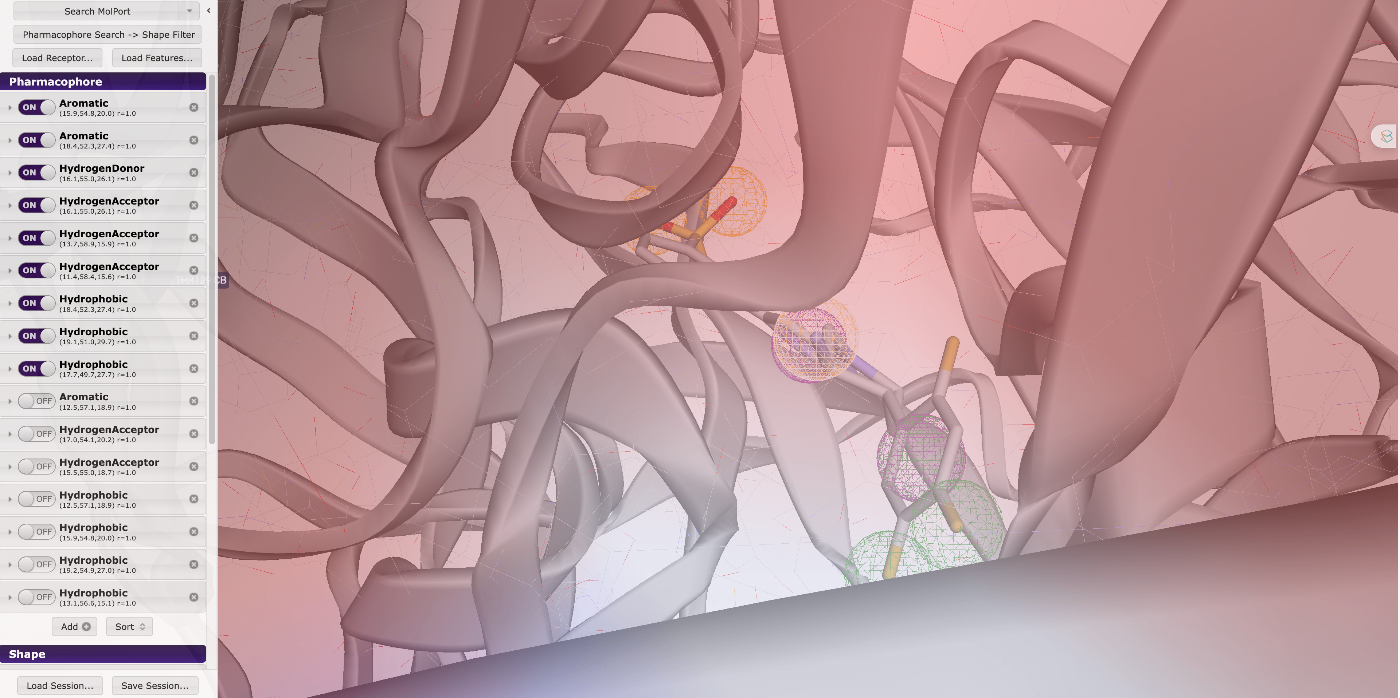
スクロールして、タンパク質の全体を見ることもできます。
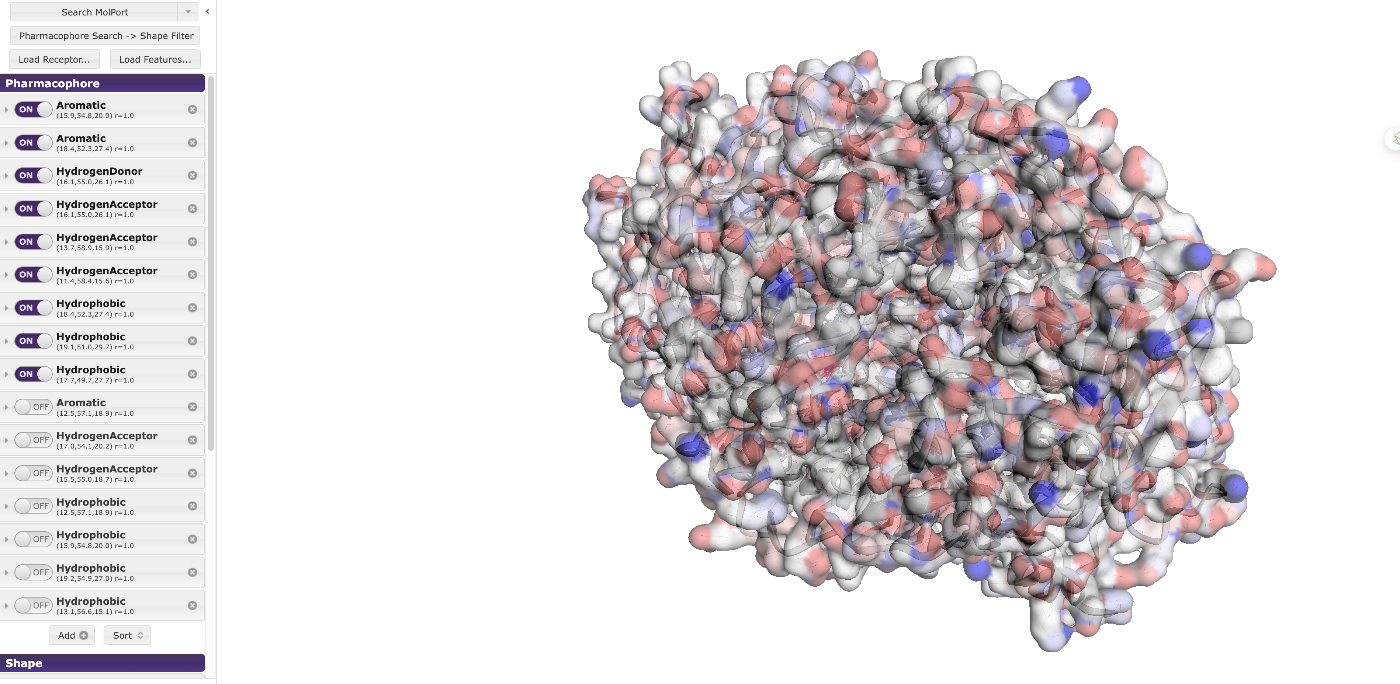
見えにくいので、受容体表面は見えやすいようにします。Receptor Surface Ppacityの値を小さくしてください。
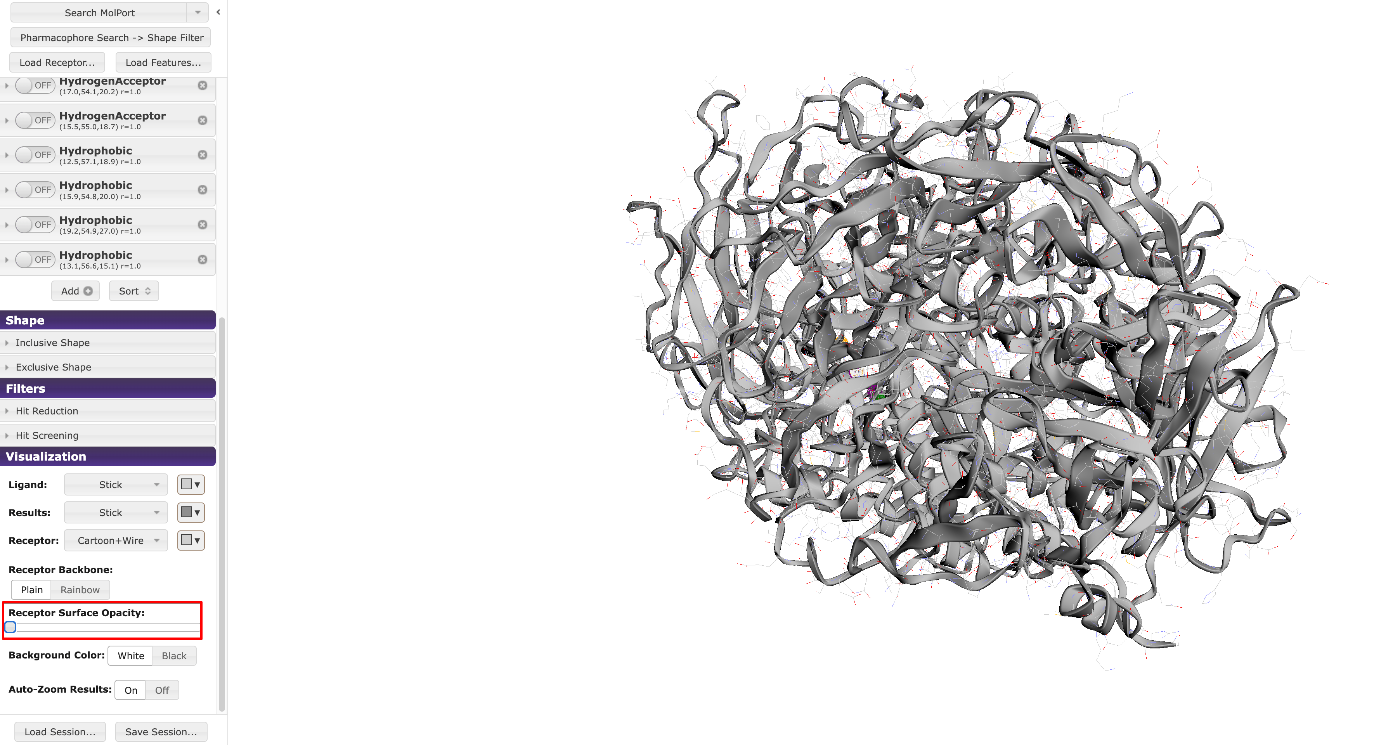
左側のPharmacophoreの部分から残しておきたいPharmacophoreをONしておきます。これにより、リガンドの化学的特徴が自動的に抽出され、ファーマコフォアの点(feature points)として可視化されます。
- 水素結合供与体(Hydrogen Bond Donor, HBD)
- 水素結合受容体(Hydrogen Bond Acceptor, HBA)
- 疎水性(Hydrophobic)
- 芳香環(Aromatic) といった特徴点が自動的に認識されます。リガンドの中の緑や紫の丸の部分です。
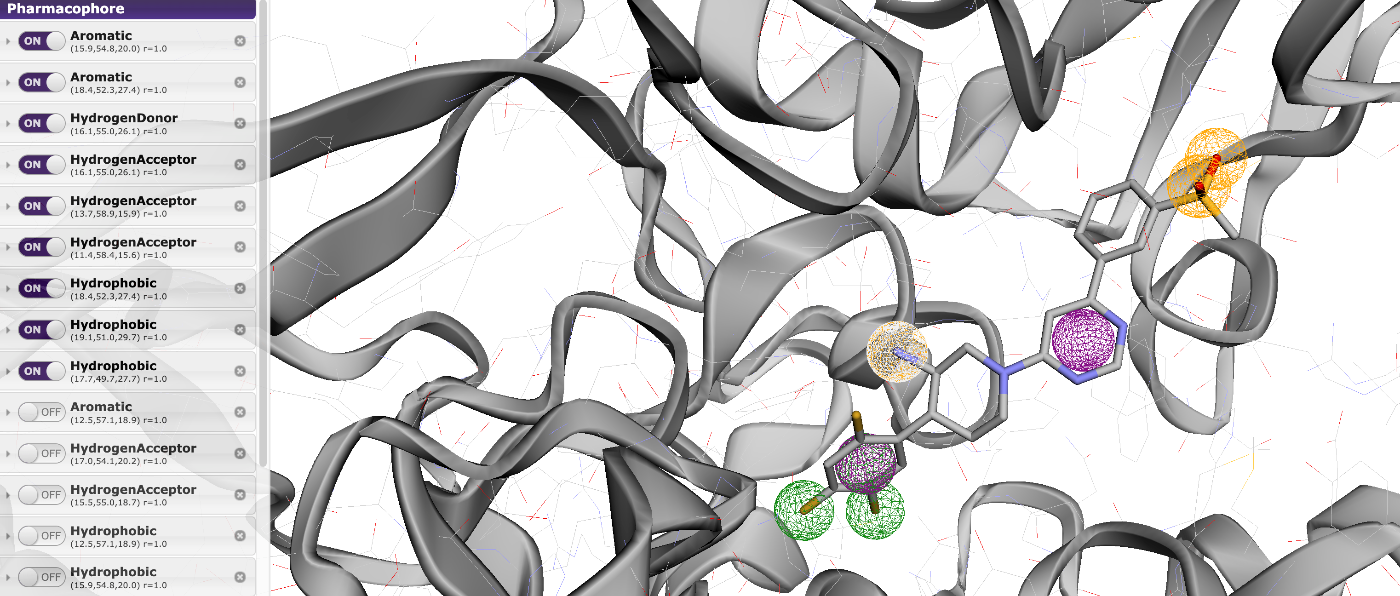
ファーマコフォアが作成できたら、Search タブに移動します。
Databases では、検索対象となる化合物データベースを選択できます。ここでは、ZINC データベースの購入可能なものを使用します。
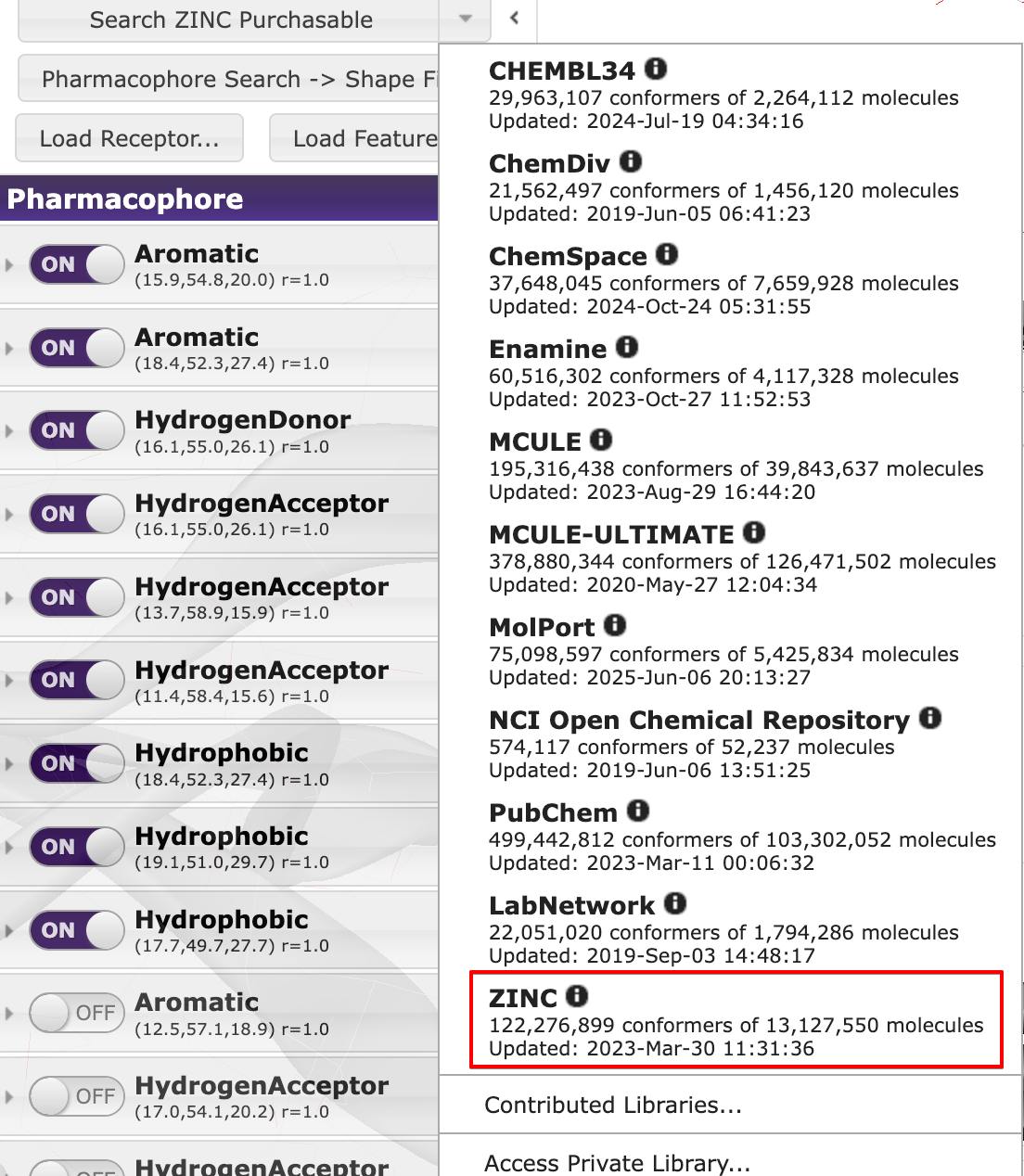
Search ZINC Purchasable ボタンをクリックして、検索を開始します。
検索が完了すると、ファーマコフォアに適合する化合物が結果として表示されます。表示された化合物のリストから、それぞれの3D構造を確認し、同じような場所に同じようなファーマこフォアがあることが確認できます。この名前をZINCで検索し、化合物を取得してみてください。
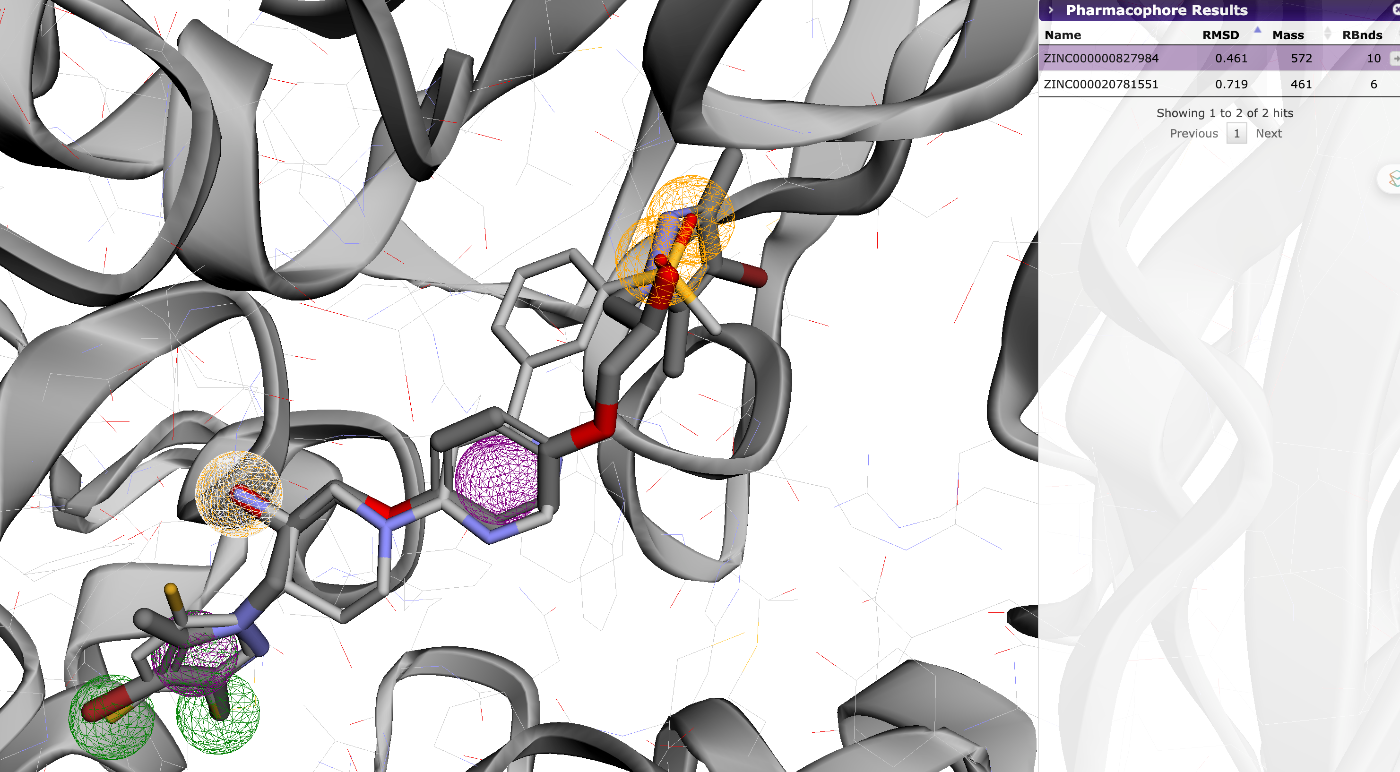
※上記では設定しておいたファーマコフォアの数が多かったので、あまり化合物がヒットしませんでしたが、もう少しファーマコフォアの数を少なくすると、化合物ライブラリも構築可能です。
検索結果のフィルタリング
Pharmitの検索結果には、作成したファーマコフォアに適合した化合物がリストアップされ、それぞれに以下の項目が表示されます。
| 項目名 | 意味 | 補足 |
|---|---|---|
| Name | 化合物の識別子 | ZINCデータベースにおける化合物のIDです。このIDを使って、ZINCデータベースから化合物の詳細情報を確認できます。 |
| RMSD | 重ね合わせのずれの度合いを示す値 | Root-Mean-Square Deviationの略で、ファーマコフォアの点と、化合物の対応する化学的特徴の点の位置的なずれ(ばらつき)を数値化したものです。値が小さいほど、ファーマコフォアモデルと化合物の立体配置がよく一致していることを意味します。 |
| Mass | 化合物の質量(分子量) | 化合物の分子量(単位:Da, ダルトン)です。医薬品開発においては、一般的に分子量が500 Da以下であることが望ましいとされています(リピンスキーの法則)。 |
| RBnds | 回転可能な結合の数 | Rotatable Bondsの略で、分子内の自由に回転できる結合の数を示します。この数が多いと、分子の柔軟性が高まり、ターゲットタンパク質に結合する際に多くのコンフォメーション(立体配置)を取り得ます。柔軟性が高すぎると、エントロピー的に結合が不利になることがあります。 |
RMSDについて
RMSDは、検索結果を評価する上で特に重要な指標です。ファーマコフォア検索では、化合物のどの部分がファーマコフォアの各点(水素結合受容体、疎水性部位など)に適合しているかを判断します。RMSDが小さい化合物は、ファーマコフォアの形状と化学的性質を高い精度で満たしているため、より有望な候補であると考えられます。
図の例では、ZINC000000827984 のRMSDが0.461であるのに対し、ZINC00020781551 は0.719であり、前者の方がファーマコフォアとの適合性が高いことが分かります。
また以下のMinimizeからも検索できます。

以下のように出てきます。

| 項目名 | 意味 |
|---|---|
| Name | 化合物の識別子。ZINCデータベースのIDです。 |
| Score | 結合エネルギーの予測値(Autodock Vina)。Pharmitが計算した、化合物とタンパク質の間の結合親和性を示すスコアです。スコアは負の値がより安定な結合を示し、値が小さいほど結合親和性が高い(=強力に結合する)と予測されます。 |
| mRMSD | ファーマコフォア検索で見つかった化合物の3D構造が、結合ポケット内で最適化された後でも、どれだけ元のファーマコフォアモデルに忠実であるかを示す指標です。 |
ライブラリ構築の際はScoreが低いもの順やmRMSDが低いもの順で上位何個かを取ってスクリーニングすると良いでしょう。
以下のような論文に使用されているので、ご参考にしてみてください。
最後に
Pharmitは、ファーマコフォアという概念を視覚的に理解し、実際に探索を体験する上で非常に優れたツールです。環境構築が不要で、初心者でも直感的に操作できるため、in silico創薬の一歩として、ぜひ活用してみてください。
本記事で解説したファーマコフォア検索は、創薬研究の初期段階で非常に重要な役割を果たします。効率的な化合物探索を通じて、新たな医薬品の発見に貢献できることを願っています。
参考文献
- Pharmit公式サイト: https://pharmit.csb.pitt.edu/
- 論文: Pharmit: A Web-Based Platform for Interactive Pharmacophore Search
Pharmit: interactive exploration of chemical space
GitHub – dkoes/pharmit: Open-source online virtual screening tools for large databases
the BSD 3-clause license and the GNU Public License version 2
自宅でできるin silico創薬の技術書を販売中
新薬探索を試したい方必読!
ITエンジニアである著者の視点から、wetな研究者からもdryの創薬研究をわかりやすく身近に感じられるように解説しています
自宅でできるin silico創薬の技術書を販売中
タンパク質デザイン・モデリングに焦点を当て、初めてこの分野に参入する方向けに、それぞれの手法の説明から、環境構築、実際の使い方まで網羅!









