今回は注目の環状ペプチド探索・生成法であるRFPeptideについて解説をしていきます。RFPeptideの概要・インストール方法・使い方を合わせて説明します。
RFPeptideを使えるようになると、ニューラルネットワークを使った非常に高精度な環状ペプチドのDe novo生成が可能になります。一緒にマスターしていきましょう!
【この記事のまとめ】
創薬研究者やバイオインフォマティクスに関心がある方に向けて、拡散モデル(Diffusion Model)を活用して高精度な環状ペプチドを新規設計(De novo生成)できるツール「RFPeptide」の使い方を解説します。
- 拡散モデルによる高度な構造最適化:ノイズ除去のプロセスを通じて、ターゲットタンパク質に最適に結合するペプチド骨格を、従来の物理シミュレーションより高速かつ高精度に生成します。
- 環状ペプチドに特化した設計:創薬において安定性が高く重要な「環状構造」の生成にフォーカスし、具体的なパラメータ設定や実行手順を詳しく紹介します。
- WSL2/GPU環境での実践ガイド:環境構築から実際のPDBファイルを用いた実行、結果の確認まで、ハンズオン形式で迷わず進められるワークフローを網羅しています。
この記事を読むことで、最先端の生成AI技術を創薬ワークフローに取り入れ、PC一台で独自の高品質なペプチドライブラリを創出するスキルが身につきます。
Windows 11, WSL2(Ubuntu 20.04),bash(Micromamba 初期化済)
PCスペック
CPUメモリ: 16GB
GPU0: NVIDIA GeForce RTX 2070 SUPER(専用メモリ 8GB)
GPU1: Intel UHD Graphics 630(内蔵 GPU)
GPUドライバー: 581.29-desktop-win10-win11-64bit-international-dch-whql
自宅でできるin silico創薬の技術書を販売中
新薬探索を試したい方必読!
ITエンジニアである著者の視点から、wetな研究者からもdryの創薬研究をわかりやすく身近に感じられるように解説しています
自宅でできるin silico創薬の技術書を販売中
タンパク質デザイン・モデリングに焦点を当て、初めてこの分野に参入する方向けに、それぞれの手法の説明から、環境構築、実際の使い方まで網羅!
RFPeptideとは?
RFPeptideとは、ディープラーニングを用いてペプチドの立体構造や結合を設計・予測するためのツールです。ターゲットタンパクの構造を渡すと、その構造に対して結合する最適化されたペプチド骨格を生成してくれます。
以下でもう少し詳しく説明します。
RFPeptideは、拡散モデル(diffusion model)に基づく生成技術を利用して開発された手法で、ペプチドの構造最適化(標的タンパク質に対する結合ペプチドの構造設計)を目的としています。
従来の物理シミュレーションやランダム探索に比べ、拡散モデルを活用することで高速かつ高精度に候補を生成できる点が特徴であり、このRFPeptideはペプチドに特化(短いアミノ酸配列の立体構造予測・最適化)しています。
仕組みについて(より詳しく知りたい方向け)
先ほどの説明では物足りなかった方向けにこのツールの内部をより詳しくご説明します。
RFPeptideは、拡散モデル(diffusion model)に基づく生成技術を利用する手法と言いましたが、拡散モデルという言葉は初めて聞いた方も多いのではないでしょうか。
拡散モデルとは、確率的なノイズ除去の過程を利用してデータを生成する深層生成モデルです。
もう少し詳しく説明すると、拡散モデルは、以下の2stepに分けられます。
- 前向き過程(noising process)
- 元のデータ(例:ランダムなペプチド構造)に少しずつガウスノイズを加えていき、最終的に「純粋なノイズ」に変換するステップ。
- 逆過程(denoising process)
- 学習済みのニューラルネットワーク(NN)を用いて、ノイズから徐々に元のデータを復元していくステップ。(この過程で多様な復元像が生成されうる。)
RFPeptideではこの「ノイズを段階的に入れて壊してから、逆に段階的に直す」プロセスを利用し、ノイズから無数の新しいペプチド構造(ペプチドライブラリ)が生み出され、その中からより最適な立体構造に収束する形でノイズが除去されていきます。
拡散モデルを利用することのはメリットは、
①破壊と修復を段階的に行う点と、②ライブラリ生成に学習済みNNを用いる点です。
まず、1stepではなく段階的に破壊と修復を行うことで、より安定的に多様なペプチドライブラリを探索できます。さらに、逆過程において既存のペプチド構造を学習したNNモデルを利用することで、物理的に妥当で自然なペプチドライブラリを探索でき、またNNでの条件付き生成という形でユーザーが指定した条件(ターゲットタンパクに結合しやすい様にや、特定構造を含む様になど)を自然とライブラリ探索に活かせるために、より高品質なライブラリを高速で探索できるというメリットがあります(ランダム変異のような形でペプチドライブラリを生成する場合と比較して)。
RFPeptideはこれらのメリットから、最近注目されるペプチド構造探索法となっています。
RFPeptideの実装方法
では早速、注目のペプチド骨格構造探索法であるRFPeptideを動かしていきます。
こちらを参考に環境構築から行っていきます。今回もWindowsのWSLを利用していきます。WSLの利用方法はこちらの記事に記載してあるので、そちらを参考にしてください。
今回のRFPeptideはGPUを利用するツールのため、まずはこちらのサイトでお使いのPCに搭載されているGPUに 対応したドライバ(Windows11用・Linux用ではありません)をDLして、Windows 上でインストーラを実行し、再起動して有効化してください。また、どのドライバーが良いのか分からない場合は、こちらのNDIVIA appをインストールすると最適なドライバーをインストールしてくれるようです(既にインストール済みの場合は不要です。)
次に、WSLのターミナルで以下のコードを実行して、環境構築をしていきます。
# micromamba をインストールするディレクトリを作成
mkdir -p ~/micromamba_env
# micromamba をインストール(-b: バッチモード, -p: インストール先指定)
curl -Ls https://micro.mamba.pm/install.sh | bash -s -- -b -p ~/micromamba_env
# bash 設定ファイルを読み込み直す(PATH 設定などを反映)
source ~/.bashrc
# GitHub から RFPeptide をクローン
git clone https://github.com/RosettaCommons/RFdiffusion.git
# 学習済みモデルの重みをダウンロード
cd RFdiffusion/scripts
bash download_models.sh ../models
# フォルダを移動
cd ~/RFdiffusion
# micromambaで環境を用意
micromamba env create -f env/SE3nv.yml --channel-priority flexible
# 環境の有効化
micromamba activate SE3nv
# GPU利用に必要なパッケージをインストール
micromamba install -c nvidia -c pytorch -c conda-forge pytorch torchvision torchaudio pytorch-cuda=12.1 -y
# SE3Transformer のインストール
cd env/SE3Transformer
pip install --no-cache-dir -r requirements.txt
python setup.py install
cd ../..
# RFPeptide のインストール
pip install -e .
プログラムを実行する
まずは、作業したいフォルダを決めます(私はWindowsのdocuments下に作ったRFPeptideというフォルダで作業します)。
次に、RFPeptideに渡すターゲットタンパクの立体構造のPDBファイルを用意します。利用する立体構造はご興味に合わせてどのようなものでも良いですが、私はこちらの論文内で利用されていたタンパクファイル(7zkr_GABARAP.pdb)をダウンロードして、ターゲットタンパクとしました。 (既に目的のタンパク構造ファイルがある方も同様に、ここで立体構造ファイルをコピペで作業フォルダ内に置いておきましょう)。
7zkr_GABARAP.pdbのファイルはこちらからダウンロードできます。ダウンロードしたファイル(rfd_macro.tar.gz)のexamples>input_pdbs内にあるので、こちらを作業フォルダ内に移動しておきましょう。
次は、以下のコマンドで作業したいフォルダに移動します
# 作業フォルダへ移動
cd /mnt/c/users/(あなたのWondowsのユーザー名)/documents/RFPeptide
/documents/RFPeptideは、ご自身が作業したいフォルダの名前に合わせて変更して下さい。あとは以下のコマンドを実行するだけです。
/home/(あなたのWSLのユーザー名)/RFdiffusion/scripts/run_inference.py\
inference.input_pdb=7zkr_GABARAP.pdb\
'contigmap.contigs=[12-18 A3-117/0]'\
inference.num_designs=5\
inference.cyclic=true\
diffuser.T=25
実行結果
作業していたフォルダにoutputとsampleというフォルダが出来ているはずです。

outputには実行時のログが入っており、生成された環状ペプチドはsamplesに保存されています。
samplesフォルダ内はこのようになっていますdesign_0から4までの5種類の立体構造ファイル(pdb)と生成条件に関するファイル(trb)、生成過程の様子に関するファイルが入っているフォルダ(traj)が作成されています。
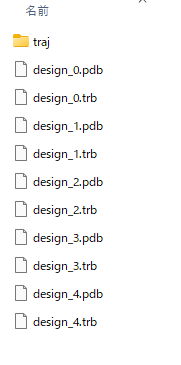
この様にオプションで指定した通りに、5つの環状ペプチド構造を作成することに成功しました!
早速、Mol* Viewerを利用してdesign_0と標的タンパク質を可視化してみましょう!
Mol*Viewerを開き、以下からdesign_0.pdbを開いてみてください。
茶色の受容体タンパクと緑の環状ぺプチドが、確かに結合されていることがわかりますね!
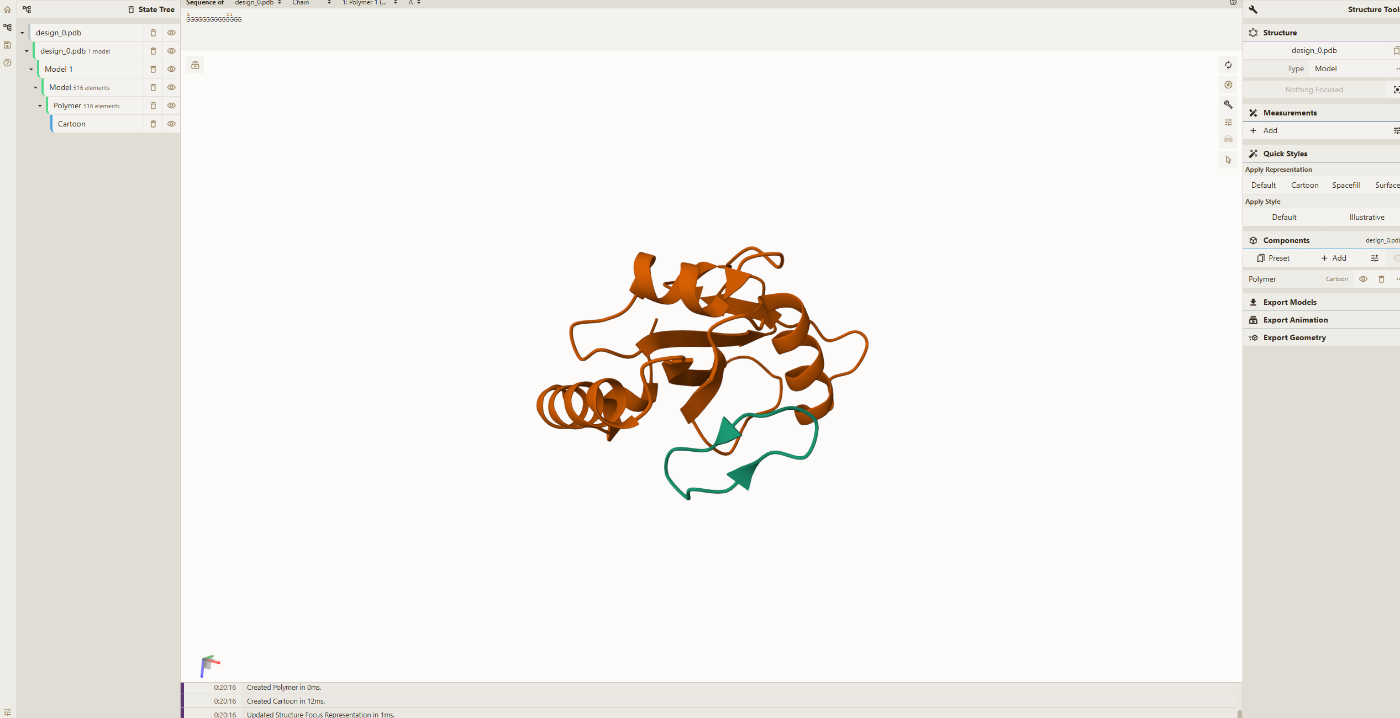
これら立体構造ファイルをこちらで紹介したAutoDockなどの結合評価ツールで利用すれば、そのデザインの性能を評価出来るでしょう。
また、今回作成した環状ペプチドの配列自体は全てグリシンから構成されていることもわかるかと思います。これは、あくまでRFeptideは立体構造のみを生成するツールであり、配列までは決定できないためです。配列の決定自体は別のツール(ProteinMPNN)を利用してやる必要があります(配列の決定に関しては、次回の記事で解説していきます)。
コードの解説
上に書いたソースコードの解説をしていきます。
# micromamba をインストールするディレクトリを作成
mkdir -p ~/micromamba_env
# micromamba をインストール(-b: バッチモード, -p: インストール先指定)
curl -Ls https://micro.mamba.pm/install.sh | bash -s -- -b -p ~/micromamba_env
# bash 設定ファイルを読み込み直す(PATH 設定などを反映)
source ~/.bashrc
今回もmicromambaを使った環境構築をしました。こちらはそのmicromambaをインストールするコマンドです。すでにインストールしている場合は、不要です。
# GitHub から RFPeptide をクローン
git clone https://github.com/RosettaCommons/RFdiffusion.git
# 学習済みモデルの重みをダウンロード
cd RFdiffusion/scripts
bash download_models.sh ../models
-
git cloneは、GitHub にあるプロジェクト(この場合は RFdiffusion(RFpeptideのもとになるツールです))を自分のパソコンにコピーするコマンドです。 -
https://github.com/RosettaCommons/RFdiffusion.gitはコピー元の URL です。実行すると、カレントディレクトリ(今いるフォルダ)に RFdiffusion という名前のフォルダが作られ、その中にプロジェクトのファイルが全部入ります。
イメージ: 「インターネット上の本棚(GitHub)から、本を丸ごと借りて自分の机に置く」ような感じです。
-
cdは「Change Directory(ディレクトリを移動)」の略です。 -
RFdiffusion/scriptsは、先ほどクローンしたフォルダの中の scripts というサブフォルダに移動します。 -
bashは Linux/Mac の「シェル」というコマンド実行環境でスクリプトを動かす命令です。 -
download_models.shは、学習済みのモデルの重み(RFPeptide のニューラルネットが計算に使うデータ)をダウンロードするためのスクリプトです。 -
../modelsは「1つ上のフォルダに models という名前で保存してね」という意味です。..は「1つ上の階層」を指します。なのでRFdiffusion/scriptsの上の階層(つまりRFdiffusionフォルダ)に models フォルダが作られます。
# フォルダを移動
cd ~/RFdiffusion
# micromambaで環境を用意
micromamba env create -f env/SE3nv.yml --channel-priority flexible
# 環境の有効化
micromamba activate SE3nv
# GPU利用に必要なパッケージをインストール
micromamba install -c nvidia -c pytorch -c conda-forge pytorch torchvision torchaudio pytorch-cuda=12.1 -y
# SE3Transformer のインストール
cd env/SE3Transformer
pip install --no-cache-dir -r requirements.txt
python setup.py install
cd ../..
# RFPeptide のインストール
pip install -e .
ここでは、RFPeptide用の環境を作成し、その中に必要なパッケージやライブラリをインストールしています。
-
micromanmba env createは新しい仮想環境を作るコマンドです。 -
f env/SE3nv.ymlは、先ほどGitHubからコピーしたプロジェクトの中に用意されていた「env/SE3nv.yml という設定ファイル」を使って環境を作るという意味です。この yml ファイルには、必要な Python のバージョンやライブラリが書かれています。
-
-channel-priority flexibleは「依存関係を柔軟に解決してくれる」という意味です。イメージとしては「RFPeptide 専用の別の作業部屋(環境)を作って、その中に必要な道具(ライブラリ)を揃える」感じです。
-
micromamba install -c nvidia -c pytorch -c conda-forge pytorch torchvision torchaudio pytorch-cuda=12.1 -yこちらは、GPU を使って計算するために必要なパッケージをインストールします。これらのパッケージは、通常は先ほどのenv/SE3nv.ymlからの環境構築でインストールされているはずですが、私の場合なぜか上手くいっていなかったので、こちらでやり直しています。皆様も念のためやり直しておくとよいと思います。
-
-cは「このチャンネルからライブラリを取ってきてね」という意味です。nvidia→ GPU 関連pytorch→ PyTorch(ディープラーニング用ライブラリ)conda-forge→ その他便利ライブラリ -
pytorch torchvision torchaudio pytorch-cuda=12.1はインストールするライブラリです。pytorch-cuda=12.1は CUDA 12.1 という GPU 計算用のバージョンを指定しています。 -
-yは「途中で確認しなくても自動で Yes にする」という意味です。 -
cd env/SE3Transformerこれは RFPeptide が使う特別なモジュールです。
SE3Transformer というサブフォルダに移動します。
-
pip install --no-cache-dir -r requirements.txtrequirements.txtに書かれた必要な Python ライブラリをインストールします。-no-cache-dirは「古いキャッシュを使わずに新しくインストールする」という意味です。 -
python setup.py installSE3Transformer モジュールを Python 環境にインストールします。
-
cd ../..元の RFdiffusion フォルダに戻ります。
-
pip install -e .-eは editable(編集可能)モード の意味です。このモードでインストールすると、フォルダ内のファイルを直接編集しても、そのまま環境に反映されます。
.は「今いるフォルダ(RFdiffusion)をインストール対象にする」という意味です。つまり、RFPeptide 自体を installして、利用できるように読み込んでいます。
# 作業ファルダへ移動
cd /mnt/c/users/(あなたのWondowsのユーザー名)/documents/RFPeptide
ここのコードでは、実際に作業したいWindows上のフォルダ(私の場合はdocuments/RFPeptide)に移動しています。
/home/(あなたのWSLのユーザー名)/RFdiffusion/scripts/run_inference.py\
inference.input_pdb=7zkr_GABARAP.pdb\
'contigmap.contigs=[12-18 A3-117/0]'\
inference.num_designs=5\
inference.cyclic=true\
diffuser.T=25
先ほどインストールしたRFPeptideのメインスクリプトであるrun_inference.pyを呼び出して、構造生成を行っています。
-
inference.input_pdb=7zkr_GABARAP.pdb設計対象の タンパク質構造ファイル を指定しています。
7zkr_GABARAP.pdbは先ほどダウンロードした PDB ファイルです。ここで指定したタンパク質構造に対してペプチド結合設計が行われます。 -
'contigmap.contigs=[12-18 A3-117/0]’ターゲットタンパクのうちのどのアミノ酸領域に、何塩基のペプチドを結合させたいかを指定しています。意味:
[結合させたい部分のペプチドの塩基数の範囲/ターゲットタンパク上の結合させたい場所/結合させない部分のペプチドの塩基数の範囲][12-18 A3-117/0]は:ターゲットタンパク質の A鎖の3〜117番目残基に対して長さ12〜18のペプチドを設計するという意味です。 -
inference.num_designs=5作成するペプチドデザインの数を指定しています。この場合 5種類のペプチドデザイン が出力されます。
-
inference.cyclic=true設計するペプチドを 環状(ループ状)にするかどうか を指定しています。true→ 環状ペプチドを設計、false→ 直鎖ペプチドを設計 -
diffuser.T=25ペプチド設計の際に使われる 拡散モデルのステップ数 を指定しています。数字が大きいほど設計精度が上がる場合がありますが、計算時間も長くなります。環状ペプチドの場合は25スッテプが良いとの論文もありますが、調整の余地があると思われます。
その他には設計に関わる以下のようなオプションが存在し、細かく指定することでより詳細な構造探索が行えます。
| オプションの使用例 | 意味 | 補足 |
|---|---|---|
| ppi.hotspot_res=[A30,A33,A34] | contigmap.contigsで設定した範囲うちで、特に結合してほしい箇所を示す。 | contigmap.contigsで大まかな範囲を、こちらでより細かい範囲(特に結合してほしい塩基)を指定できます。 |
| inference.cyc_chains=’a’ | 環状にしたい鎖の指定。 | chain aを環状にするという意味 |
| ‘contigmap.contigs=[150-150]’ | 環状ペプチドの塩基数のみを指定するやり方。(結合させたいタンパクを指定しない場合) | 150塩基を指定する場合は ‘150-150’。’10-20’の場合は10~20塩基のいずれかで作成。 |
| inference.output_prefix=~~~ | 結果の出力フォルダの名前を指定できる | なしだとoputputというフォルダ名になる |
最後に
今回は、最新の強力なペプチド構造探索法である RFPeptide を利用しました。GPU前提の計算量を必要とするツールですが、使用するとより大規模かつ多様なペプチドライブラリから高速かつ高精度な探索が可能です。
実践的には、よくあるのがタンパク質ータンパク質相互作用面に結合するペプチドをデザインし、その相互作用を阻害する利用法です。その際はターゲット部位を見つけることがミソになりそうです。
今後より一層使われていくであろうツールの一つだと思いますので、ぜひ一度お試しください。
参考文献
RF Peptide
https://github.com/RosettaCommons/RFdiffusion?tab=readme-ov-file BSD License
自宅でできるin silico創薬の技術書を販売中
新薬探索を試したい方必読!
ITエンジニアである著者の視点から、wetな研究者からもdryの創薬研究をわかりやすく身近に感じられるように解説しています
自宅でできるin silico創薬の技術書を販売中
タンパク質デザイン・モデリングに焦点を当て、初めてこの分野に参入する方向けに、それぞれの手法の説明から、環境構築、実際の使い方まで網羅!










