この記事ではAlphafold Analysisを利用した立体構造予測結果の分析を扱います。この記事を理解することでAlphafoldやlocalcolabfoldなどの立体構造予測モデルを定量的に解析できます。
【この記事のまとめ】
AlphaFold 2やLocalColabFoldによるタンパク質立体構造予測の結果を定量的に評価し、複数の指標(pLDDT, PAE等)を用いて可視化・解析するツール「af_analysis」の実装手順を解説します。
- 初心者でも容易な一括解析: 従来、複数のスクリプト実行が必要だった複雑な構造解析を、Google Colaboratory上でライブラリ(
af_analysis)を用いることで簡単に実行・可視化できるワークフローを提示。 - 主要な予測精度指標の解釈: 残基ごとの信頼度を示す
pLDDT、相対的な位置誤差を示すPAE、さらに多量体の相互作用を評価するipTMやpDockQなどのスコアを定量的に算出し、予測構造の妥当性を検証する手法を詳説。 - インタラクティブなデータ分析: ヘモグロビン(1A3N)の解析例を通じ、Google Driveと連携したデータのロードから、信頼度マップのプロット、スコアに基づく最良・最悪構造の比較までをPythonコードと共に紹介。
この記事を読むことで、AIが生成した膨大な構造データの中から、どの予測結果が最も信頼に値し、どのドメインが安定しているかを科学的根拠に基づいて客観的に判断できるようになります。
macOS Montery(12.4), python3.7.10, Jupyter Notebook
自宅でできるin silico創薬の技術書を販売中
新薬探索を試したい方必読!
ITエンジニアである著者の視点から、wetな研究者からもdryの創薬研究をわかりやすく身近に感じられるように解説しています
自宅でできるin silico創薬の技術書を販売中
タンパク質デザイン・モデリングに焦点を当て、初めてこの分野に参入する方向けに、それぞれの手法の説明から、環境構築、実際の使い方まで網羅!
Alphafold Analysisとは?
Alphafold Analysisとは、Alphafold 2による立体構造予測の結果を分析するためのツール群です。Alphafold 2はアミノ酸配列から立体構造を予測するAIツールです。Alphafold 2によって生成された構造を分析するために、いくつかの指標が提案されているが、それらを利用するには複数のスクリプトを実行する必要があり、初心者に取って難しい課題があります。Alphafold Analysisでは、これを解決するため、複数の指標による評価や可視化を簡単に実行できるライブラリを提供します。
Alphafold Analysisを利用した構造予測結果の分析
早速Alphafold Analysisを用いて構造予測結果の分析を行ってみましょう!
こちらからGoogle Colaboratoryのページに飛んでください。
また、今回はlocalcolabfoldによって出力されたヘモグロビン(1A3N)の結果を利用します。
具体的な利用方法に関しては後ほど説明します。
ではコードを実行していきます。詳細なコード説明については本ページの最後にあります。
パッケージのインストール
まず、以下のコードを実行し、必要なパッケージをインストールします。
# @title Install `af_analysis` library {"display-mode":"form"}
%%bash
git clone https://github.com/samuelmurail/af_analysis;
cd af_analysis;
pip install .;
pip install ipympl;
以下の画像のように、エラーによって処理が中断せずに実行できれば問題ないです。

場合によってはこちらのようなエラーが発生します。
ERROR: pip's dependency resolver does not currently take into account all the packages that are installed. This behaviour is the source of the following dependency conflicts.
google-colab 1.0.0 requires notebook==6.5.7, but you have notebook 7.4.3 which is incompatible.
jupyter-kernel-gateway 2.5.2 requires jupyter-client<8.0,>=5.2.0, but you have jupyter-client 8.6.3 which is incompatible.
jupyter-kernel-gateway 2.5.2 requires notebook<7.0,>=5.7.6, but you have notebook 7.4.3 which is incompatible.
一部のパッケージのバージョンが衝突していることが原因です。その場合は、先ほどのコードを以下のように書き換えて実行してください。
# @title Install `af_analysis` library {"display-mode":"form"}
%%bash
git clone https://github.com/samuelmurail/af_analysis;
cd af_analysis;
pip install .;
pip install ipympl;
pip install notebook==6.5.7 jupyter-client<8.0; # 修正部分
ログの設定
続いて、以下のコードを実行してログの設定を行います。追加部分によってGoogle Colablatory上でインタラクティブにグラフ操作ができます。
# @title notebook configuration
import logging
from google.colab import output
output.enable_custom_widget_manager()
logging.basicConfig(filename='app.log',
level=logging.DEBUG,
force=True, # Resets any previous configuration
)
%matplotlib ipympl
from google.colab import output # 追加部分
output.enable_custom_widget_manager() # 追加部分
場合によってはこちらのようなエラーが発生します。

その場合は、%matplotlib ipympl をコメントアウトして先ほどのコードを再度実行してください。
パッケージとデータのロード
まず、こちらからLabCodeで用意したlocalcolabfoldの出力結果をダウンロードします。
右上にある「すべてダウンロード」をクリックすると、ダウンロードが始まります。

Hemoglobin-20250529T100200Z-1-001.zip のようなzipファイルがダウンロードされます。
ファイル内に1A3N_unrelaxed_rank_005_alphafold2_multimer_v3_model_1_seed_000.pdb のようなpdbファイルが5個あります。これは、localcolabfoldで生成した5個の立体構造予測結果です。
こちらzipファイルを解凍すると、Hemoglobinというフォルダが展開されます。
このHemoglobinフォルダをご自身のGoogle Driveにアップロードしてください。
では、コードの実行に戻ります。
以下のコードを実行して、Google Driveとの連携を行います。こちらは追加コードです。
from google.colab import drive # 追加部分
drive.mount('/content/drive') # 追加部分
場所はimporting Alphafold Data の下とかでいいと思います。コードはctrl+M+Bで追加できます。

以下のようなポップアップが表示されるので、「Google ドライブに接続」をクリックしてください。
ご自身のGoogleアカウントをクリックしてください。

「次へ」を選択してください。

「続行」を選択してください。

以下のコードを実行して、パッケージをロードしてください。
import af_analysis
import seaborn as sns
import matplotlib.pyplot as plt
以下のコードを実行してデータをロードしてください。ロードするデータのパスを先ほどアップロードしたものに変更しています。
DATA_PATH = '/content/drive/My Drive/Hemoglobin' # 修正部分
my_data = af_analysis.Data(DATA_PATH)
では、以下のコードを実行してロードされたデータを確認します。
my_data.df.head(2)
my_data.chain_length
my_data.chains
こちらのようにエラーなく表示できていたら成功です。ロードしたデータはDataframeとして管理されます。1A3Nには複数のサブユニットが含まれています。具体的には、A、B、C、Dの4つのチェーンが存在し、それぞれの残基数は141、141、146、146であることが確認できます。

この特徴はこちらのページで表示できる、1A3Nの立体構造図の見た目とも一致しており、データと構造可視化結果の整合性が取れていることが分かります。

スコアによる分析
このツールでは、Alphafoldの出力結果などに対して、以下のスコアを用いた分析が可能です。
- pLDDT: アミノ酸残基の局所的な構造予測の信頼度を示すスコアで、0~100の範囲で表される
- PAE: 2つのアミノ酸残基間の相対的な位置関係に対する予測誤差(Å単位)。
- pTM: タンパク質全体の構造予測の精度を評価するスコア
- ipTM: タンパク質複合体におけるサブユニット間の相互作用部位の予測精度を評価するスコア
- ranking_confidence: AlphaFold Multimerの出力構造を比較してランク付けするための指標。
以下のコードを実行すると、ipTMスコアが最大、最小のものを選択できます。
best_ipTM_index = my_data.df['ipTM'].idxmax()
worst_ipTM_index = my_data.df['ipTM'].idxmin()
以下のコードを実行すると、ipTMスコアが最も良いもの詳細を見ることができます。
my_data.df.iloc[best_ipTM_index]
こちらのようにエラーなく表示できていたら成功です。

プロットによる分析
このツールでは、Alphafoldの出力結果などに対して、図表にプロットして分析することが可能です。
以下のコードを実行すると、ipTMスコアが最も良いもののpLDDTスコアをプロットすることができます。
my_data.plot_plddt([best_ipTM_index])
こちらのようにエラーなく表示できていたら成功です。この図は、立体構造における各残基の信頼度スコアであるpLDDTを示したものです。X軸はアミノ酸残基のインデックス、Y軸は対応するpLDDTスコアを表しています。多くの残基でpLDDTが70以上となっており、全体として高い信頼度が得られていることが分かります。また、図中に引かれている黒線は、複数のサブユニット間の境界を示しており、それぞれA、B、C、Dの4つのチェーンに対応しています。

以下のコードを実行すると、ipTMスコアが最も良い構造と最も悪い構造を比較して、pLDDTをプロットすることができます。
my_data.plot_plddt([best_ipTM_index, worst_ipTM_index])
こちらのようにエラーなく表示できていたら成功です。この図は、青とオレンジの線は、それぞれipTMスコアが最も高い構造と最も低い構造におけるpLDDTの分布を示しています。この図から、pLDDTスコアの差はそれほど大きくないことが分かります。

以下のコードを実行すると、ipTMが最も良いもののPAE(予測整列誤差)をプロットすることができます。
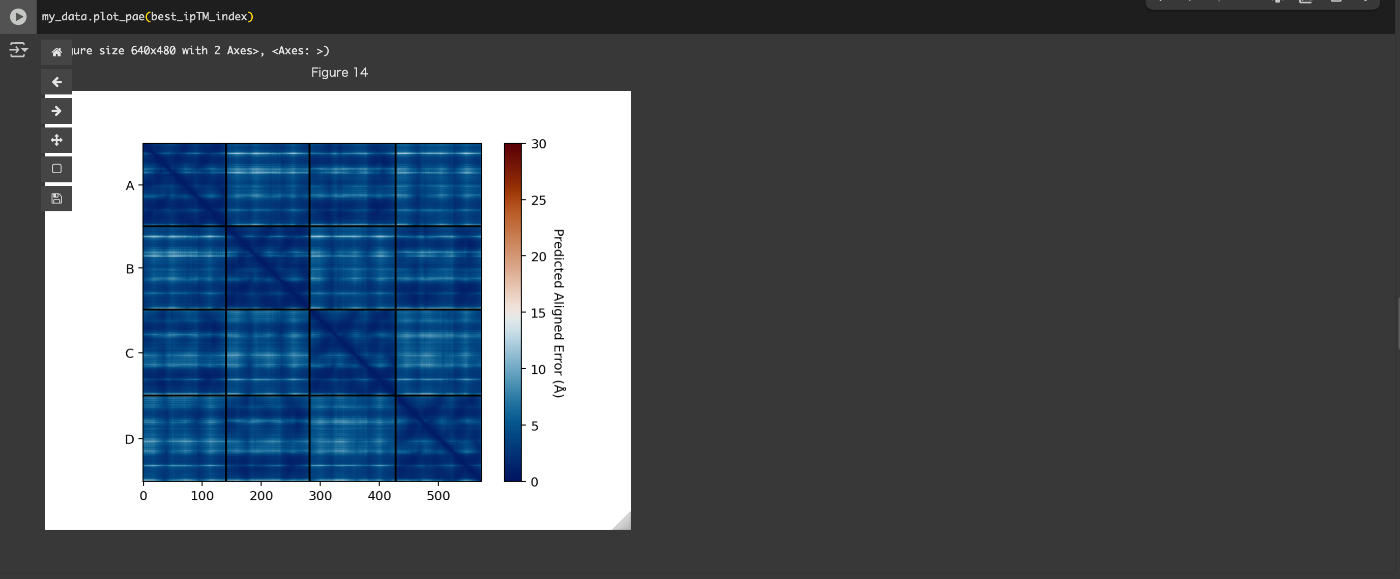
my_data.plot_pae(best_ipTM_index)
こちらのようにエラーなく表示できていたら成功です。この図は、立体構造における各残基間の予測誤差を示すPAEのヒートマップです。X軸およびY軸はアミノ酸残基の番号を表しており、各セルの色は対応する2残基間の予測誤差(Å単位)を示しています。色が青に近いほど誤差が小さく(0に近い)、赤に近いほど誤差が大きいことを意味します。また、図中に引かれた黒線は、複数のサブユニット間の境界を示しており、先ほど説明したA、B、C、Dの4つのチェーンに対応しています。
以下のコードを実行すると、全データに対してplddtとPAEをプロットできます。
スライドバーを用いて、予測された複数の立体構造データのどの結果を見るか選択できます。
スライドバーによる選択はGoogle Colaboratoryで上手く機能しない可能性があります。
from af_analysis import plot
plot.show_info(my_data)
こちらのようにエラーなく表示できていたら成功です。この図は、先ほどのpLDDTとPAEの結果を並べて表示したものです。左側の図は各残基ごとの信頼度スコアであるpLDDT、右側の図は残基間の予測誤差を示すPAEマトリクスを表しています。これにより、各残基の予測精度と残基間の位置関係の信頼度を同時に確認することができます。また、スライダーを使うことで、表示する構造をインタラクティブに切り替えることができます。

スコアの計算
このツールでは、Alphafoldの出力結果などに対して、追加でいくつかの指標を計算することができます。
以下のコードを実行すると、pdockqとpdockq2のスコアを計算できます。
- pdockq:主に2量体における、立体構造の複合体としての相互作用の強さを評価します。
- pdockq2:pDockQの改良版であり、特に多量体の相互作用の強さを評価します。
from af_analysis import analysis
#compute_pdockq
analysis.pdockq(my_data)
#compute_pdockq2
analysis.pdockq2(my_data)
以下のコードを実行すると、pae_pep、pdockq2_lig、LIS_pep、plddt_pepのスコアを計算できます。これらの指標は、ペプチド同士がドッキングして形成した複合体の構造的信頼性や結合強度を評価するために使用されます。
- pae_pep:ペプチド同士の接触部位における平均PAE値で、複合体としての相対的な位置関係の信頼性を示します。
- pdockq2_lig:ペプチド同士の接触部位間におけるpDockQ2スコアで、複合体としての結合信頼性を評価します。
- LIS_pep:ペプチド同士の接触部位間におけるLISスコアで、立体構造の複合体としての相互作用の強さを評価します。
- plddt_pep:ペプチド同士の接触部位におけるpLDDTスコアで、複合体としての局所構造の信頼性を評価します。
from af_analysis import docking
#extract_pae_pep
docking.pae_pep(my_data)
#compute_pdockq2_lig
docking.pdockq2_lig(my_data)
#compute_LIS_pep
docking.LIS_pep(my_data)
#extract_plddt_pep
docking.plddt_pep(my_data)
以下のコードを実行すると、ロードしたDataframeの列名が見れます。
my_data.df.columns
こちらのようにエラーなく表示できていたら成功です。
'pdockq', 'pdockq2_A', 'pdockq2_B', 'pdockq2_C', 'pdockq2_D', 'PAE_pep_rec', 'PAE_rec_pep', 'pdockq2_lig', 'LIS', 'LIS_rec_pep', 'LIS_pep_rec', 'plddt_pep' などの指標がDataframeの列として増えていることがわかります。

クラスタリングによる分析
このツールでは、Alphafoldの出力結果などに対して、立体構造データ間の類似度に基づいて階層的クラスタリングを実施できます。
以下のコードを実行すると、4Åの精度で階層的クラスタリングを実施できます。
from af_analysis import clustering
clustering.hierarchical(my_data.df, threshold=4)
こちらのようにエラーなく表示できていたら成功です。この図は、立体構造間の類似度に基づいて階層的クラスタリングを行った結果を示す樹形図です。X軸は構造モデルのインデックス、Y軸はクラスタ間の距離を示しており、クラスタの結合は下から上へ向かって進みます。類似度が高い構造ほど、より低い距離で結合されます。今回使用した5つの立体構造予測結果は、全体として非常に類似していることがこの図から読み取れます。

以下のコードを実行すると、クラスタリング結果をMDS(多次元尺度構成法)で2次元にプロットすることができます。
plt.figure()
sns.scatterplot(data=my_data.df, x='MDS 1', y='MDS 2', hue='cluster')
こちらのようにエラーなく表示できていたら成功です。この図は、立体構造間の距離を2次元空間上に射影し、空間的な関係を視覚的に表現したものです。X軸(MDS 1)とY軸(MDS 2)は、構造間距離行列を基にした主成分方向を示しています。すべての点がクラスタ1に属しており、他のクラスタは存在していないことが分かります。図を見ると、すべての点がクラスタ1に属しており、他のクラスタは存在していないことが分かります。これは、すべての構造モデルが非常に類似しており、同一クラスタに分類された可能性を示唆しています。また、各点同士の距離が非常に小さいことからも、構造間の違いがごくわずかであることが読み取れます。

コードの解説
1. af_analysis のインストール
git clone <https://github.com/samuelmurail/af_analysis>;
cd af_analysis;
pip install .;
pip install ipympl;
af_analysisは AlphaFold2 の出力を可視化・スコア化するライブラリです。ipymplは%matplotlib ipymplにより、インタラクティブなプロット表示を可能にします。
pip install notebook==6.5.7 jupyter-client<8.0
- Colab 環境でのバージョン競合(例: notebook 7系とColabの相性問題)を回避するために、旧バージョンに制限します。
2. ログと描画設定(Google Colab用)
import logging
from google.colab import output
output.enable_custom_widget_manager()
logging.basicConfig(filename='app.log',
level=logging.DEBUG,
force=True)
%matplotlib ipympl
loggingはライブラリの内部ログをapp.logに記録するための設定です。%matplotlib ipymplにより、グラフのズーム・パン操作がColab上で可能になります。
3. Google Drive のマウント
from google.colab import drive
drive.mount('/content/drive')
- Colab から Drive 内のファイルにアクセスするための初期設定です。
- Google Driveの中身をマウントしてくれます。
4. ライブラリとデータの読み込み
import af_analysis
import seaborn as sns
import matplotlib.pyplot as plt
seabornとmatplotlib.pyplotはクラスタリング結果などの可視化に使用します。
DATA_PATH = '/content/drive/My Drive/Hemoglobin'
my_data = af_analysis.Data(DATA_PATH)
af_analysis.Dataクラスは、フォルダ内の.pdb構造を自動的に読み込み、スコアデータを DataFrame として保持します。
5. データの基本情報の取得
my_data.df.head(2)
- AlphaFold が出力した構造ごとのスコア(例: pLDDT, ipTM, ranking_confidence など)をDataFrameとして確認します。
my_data.chain_length
- 鎖ごとのアミノ酸数が辞書形式で返ります(例:
{'A': 141, 'B': 141})。
my_data.chains
- 登録されたすべての鎖ID(例:
['A', 'B'])のリストが取得できます。
6. 信頼性スコア選択
best_ipTM_index = my_data.df['ipTM'].idxmax()
worst_ipTM_index = my_data.df['ipTM'].idxmin()
ipTMは鎖間の相互作用に関する信頼度(0〜1)で、複合体の品質判断に有用です。- 最も良い/悪い構造のインデックスを取得して、後続の可視化に使います。
my_data.df.iloc[best_ipTM_index]
- 最良構造の全スコア(pLDDT, ipTM, pTM, ranking_confidence など)を表示します。
7. pLDDT・PAE 可視化
my_data.plot_plddt([best_ipTM_index])
- pLDDT(位置ごとの信頼度スコア)を描画。信頼できるドメインや柔軟部位の判断に役立ちます。
my_data.plot_plddt([best_ipTM_index, worst_ipTM_index])
- 2つの構造を同時に描画して比較可能。
my_data.plot_pae(best_ipTM_index)
- PAE(予測整列誤差)をヒートマップ形式で表示。2残基間の相対配置に対する予測誤差(Å)を色で示します。
from af_analysis import plot
plot.show_info(my_data)
- pLDDT, PAE, 各スコアなどをGUIスライダーで切り替え表示すします。
8. pdockq・pdockq2 計算
from af_analysis import analysis
analysis.pdockq(my_data)
analysis.pdockq2(my_data)
pDockQは ipTM とインターフェースの pLDDT に基づいて、タンパク質複合体の結合信頼度をスコア化します。pDockQ2はさらに多様な複合体(例: 非対称・ペプチド含有型)に対応する拡張版です。
9. ペプチド・リガンド評価用スコア
from af_analysis import docking
docking.pae_pep(my_data)
docking.pdockq2_lig(my_data)
docking.LIS_pep(my_data)
docking.plddt_pep(my_data)
pae_pep:ペプチドと受容体間の平均PAE。相対配置の信頼度。pdockq2_lig:pDockQ2 をペプチド/リガンド評価用に拡張した指標。LIS_pep:局所インターフェース信頼度スコア。plddt_pep:ペプチド側の pLDDT 平均値。
my_data.df.columns
- DataFrame に追加された新たなスコア列(
pdockq,LIS_pep,plddt_pepなど)を確認できます。
10. クラスタリング・構造比較
from af_analysis import clustering
clustering.hierarchical(my_data.df, threshold=4)
- 構造間 RMSD を用いて階層的クラスタリングを実施。
threshold=4は 4Å を類似構造とみなす条件です。
plt.figure()
sns.scatterplot(data=my_data.df, x='MDS 1', y='MDS 2', hue='cluster')
- MDS(多次元尺度構成法)で構造間距離を2次元に縮約し、クラスタごとに色分け表示します。
最後に
AlphaFold 2を使った構造予測は、もはやバイオインフォマティクスの基本スキルと言える時代になってきました。しかし、予測された構造がどれほど信頼できるのか、どれが最も良いモデルなのかを評価するには、定量的な解析が欠かせません。
今回紹介した af_analysis を活用すれば、予測結果の品質をスコアで比較し、視覚的にも構造の特徴を把握できるようになります。
特に構造ベースの創薬やタンパク質設計を行っている研究者や学生にとって、非常に有用なツールです。ぜひ一度、ご自身のAlphaFold出力結果で試してみてください。
参考文献
自宅でできるin silico創薬の技術書を販売中
新薬探索を試したい方必読!
ITエンジニアである著者の視点から、wetな研究者からもdryの創薬研究をわかりやすく身近に感じられるように解説しています
自宅でできるin silico創薬の技術書を販売中
タンパク質デザイン・モデリングに焦点を当て、初めてこの分野に参入する方向けに、それぞれの手法の説明から、環境構築、実際の使い方まで網羅!










