前回の記事ではGoogle Earth Engine(以下GEE)のAPIを用いて、地球観測衛星(Sentinel-2)のデータから計算したNDVIのカラーマップを地図上に表示させる方法を紹介しました。あの方法では地域ごとの大まかな特徴をとらえるのに役立ちますが、数値を扱って何かを言えるわけではありません。
今回は実際の解析に繋がるような具体的な数値を扱い、時系列変化をグラフとしてプロットするところまでを紹介します。
この手法はNDVIの値から、季節と植物の状態や生活環の関係を知るのに役立ちます。
macOS Monterey(12.6), python3.10.7
Windows 11(21H2), python3.10.7, PowerShell(5.1)

Pythonを使った衛星データ解析の初心者向け技術書を販売中!
¥2,500 → ¥800 今なら約70%OFF!!
Google Earth Engineを使えば簡単に
自宅にいながら地球環境の変動を観察できます
地球観測衛星(Sentinel-2)について
時系列変化を扱う場合、主に回帰日数(観測頻度)について抑えておく必要があります。ここでは、前回も利用したSentinel-2を用いながら改めて地球観測衛星について説明します。
光学衛星の特徴
光学センサを搭載した地球観測衛星(光学衛星)は地表面もしくは海面の反射光(主に可視光、近赤外線)を測定しています。物質は固有の反射スペクトルを持つため、得られた反射光のデータによって観測地点に何が存在するかが分かります。しかし、反射光は太陽光由来であるため、曇りの日や夜間は観測できないという欠点もあります。
光学衛星の軌道について
Sentinel-2は太陽同期準回帰軌道(Sun-synchronous Sub-recurrent orbit)という軌道をしています。この軌道は太陽同期軌道と準回帰軌道を合わせた軌道です。
太陽同期軌道(Sun-synchronous orbit):
南北極付近を通過して地球を一周する極軌道の一種です。光学衛星は反射光を測定するため、太陽光が強くなる日中帯に観測します。そのため、常に同じ時刻(昼)の地球が観測できるように、図のような太陽との位置関係に同期した軌道をしています。この軌道の特徴として、図中の赤線のように季節を問わず衛星の軌道と太陽方向の角度が常に一定となります。
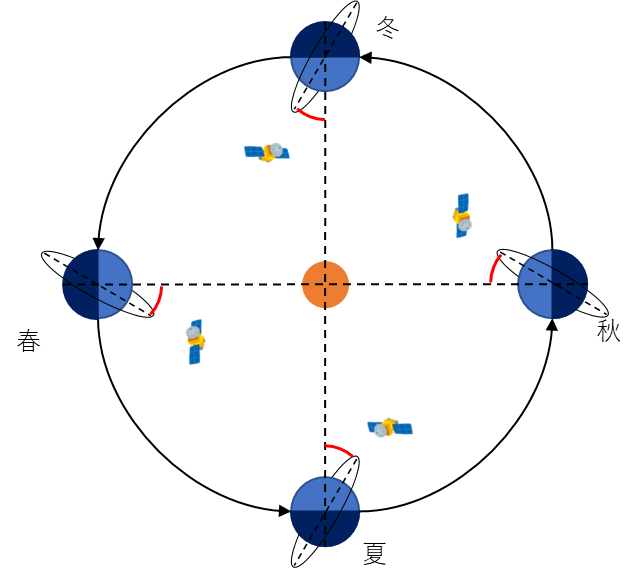
準回帰軌道(Sub-recurrent orbit):
地球がM周自転するごと、つまりM日ごとに同じ地点の上空に戻ってくる軌道です。このM日のことを回帰日数といい、衛星の高度と軌道傾斜角によって決まります。同じ地点を定期的に観測することができるため、比較解析が容易になるなどの利点があります。Sentinel-2は2Aと2Bの2機体制で、それぞれ10日の回帰日数を持ちます。そのため、Sentinel-2は2機合わせると5日の頻度で観測しています。この観測頻度は時間分解能とも言います。
測定波長について
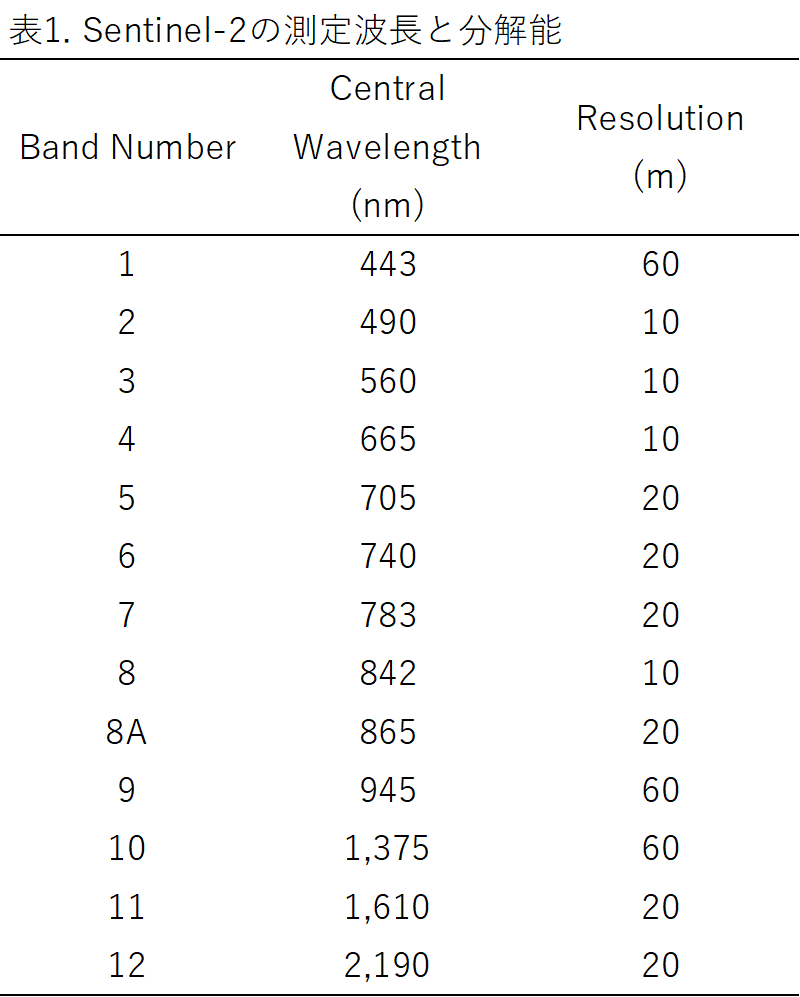
Sentinel-2はMSI(Multispectral Instrument)という名前のセンサを搭載し、表1のような13の異なる波長について分光測定しています。例えば、Sentinel-2でNDVIの解析を行う場合は、バンド4とバンド8のデータを扱います。
このような複数の波長が観測できるセンサのことをマルチスペクトルセンサといいます。例としてはLandsatやTerraなどの衛星が搭載しています。また、さらに細かく波長帯を分光測定できるハイパースペクトルセンサもあります。この分光測定できる波長帯の数についてはスペクトル分解能、波長分解能とも言います。
分解能について
ここまでで分解能について、時間分解能、波長分解能について紹介しました。最後に紹介するのは空間分解能です。単に分解能と言ったり、解像度と言う場合は、この空間分解能を指します。空間分解能は二つの点が識別可能な限界の距離で、どれくらいの大きさのものまで見れるかを示す指標となります。たとえば表1に示したSentinel-2の分解能は、青色光に当たるバンド2は10 mの分解能を示します。都市部では建物や施設の識別ができるくらいの解像度ですが、車や人の識別は出来ません。
光学衛星の画像データはラスターデータで、写真などと同様にピクセルの集まりです。NDVIに利用するSentinel-2のバンド4、バンド8は分解能10 mですので、ピクセルの1辺は10 mに相当します。そのため、解析に用いる値は、植生以外にも土壌や水面などが含まれていることは理解しておく必要があります。
NDVIの時系列変化のグラフを作成する
NDVIの変化は植物と植生がない時期のバックグラウンドに差がある場合が最も顕著になります。前回作成した地図で夏と冬のNDVIに差があった千葉県の印旛沼について、時系列変化を追ってみたいと思います。
ソースコード
import altair as alt
import ee
import pandas as pd
from altair_saver import save
# GEEの認証
# ee.Authenticate()
ee.Initialize()
# 緯度経度、データを扱う衛星の設定
location_lonlat = [140.219017, 35.757631]
stat_region = ee.Geometry.Point(location_lonlat)
sentinel2 = ee.ImageCollection('COPERNICUS/S2_HARMONIZED').filterBounds(stat_region).filter(
ee.Filter.lt('CLOUDY_PIXEL_PERCENTAGE',20))
# 関心領域を切り取る関数を定義
def reduce_region_function(img):
stat = img.reduceRegion(
reducer=ee.Reducer.mean(),
geometry=stat_region,
scale=10
)
return ee.Feature(stat_region, stat).set({'millis': img.date().millis()})
# GEEデータを辞書型に変換する関数を定義
def fc_to_dict(fc):
prop_names = fc.first().propertyNames()
prop_lists = fc.reduceColumns(
reducer=ee.Reducer.toList().repeat(prop_names.size()),
selectors=prop_names).get('list')
return ee.Dictionary.fromLists(prop_names, prop_lists)
# NDVI計算用関数を定義
def calc_ndvi(image):
ndvi = image.normalizedDifference(['B8', 'B4']).rename('NDVI')
return image.addBands(ndvi)
# データフレーム成型用関数を定義
def add_date_info(df):
df['Timestamp'] = pd.to_datetime(df['millis'], unit='ms')
df['Year'] = pd.DatetimeIndex(df['Timestamp']).year
df['Month'] = pd.DatetimeIndex(df['Timestamp']).month
return df
# グラフ描画用関数を定義
def ndvi_season(data):
highlight = alt.selection(
type='single', on='mouseover', fields=['Year'], nearest=True)
base = alt.Chart(data).encode(
x=alt.X('Month:Q', scale=alt.Scale(domain=[1, 12])),
y=alt.Y('NDVI:Q', scale=alt.Scale(domain=[-1.0, 1.0])),
color=alt.Color('Year:O', scale=alt.Scale(scheme='turbo')))
points = base.mark_circle().encode(
opacity=alt.value(1),
tooltip=[
alt.Tooltip('Year:Q', title='Year'),
alt.Tooltip('Month:Q', title='Month'),
alt.Tooltip('NDVI:Q', title='NDVI')
]).add_selection(highlight)
lines = base.mark_line().encode(
size=alt.condition(~highlight, alt.value(1), alt.value(5)))
return (points + lines).properties(width=600, height=500).interactive()
# NDVIを計算
ndvi = sentinel2.map(calc_ndvi)
ndvi_stat_fc = ee.FeatureCollection(ndvi.map(reduce_region_function)).filter(
ee.Filter.notNull(ndvi.first().bandNames()))
# 衛星データを辞書型に変換
ndvi_dict = fc_to_dict(ndvi_stat_fc).getInfo()
ndvi_df = pd.DataFrame(ndvi_dict)
ndvi_df.to_csv('sentinel2_original.csv')
# 月ごとの中央値を計算
ndvi_df['timestamp'] = pd.to_datetime(ndvi_df['millis'], unit='ms')
ndvi_df.set_index('timestamp', inplace=True)
ndvi_df = ndvi_df.resample('M').median()
ndvi_df.to_csv('NDVI_season.csv')
# データフレームの成形
ndvi_df = add_date_info(ndvi_df)
# グラフの出力
save(ndvi_season(ndvi_df), 'NDVI_season.html')上記のコードをndvi_season.pyという名前でDesktop/LabCode/python/satellite-analysisディレクトリに保存します。
プログラムの実行
Macではターミナル、WindowsではPowerShellを開き
$ cd Desktop/labcode/python/satellite-analysisと入力し、ディレクトリを移動します。あとは以下のコマンドを実行します。
$ python ndvi_season.pycsvファイルが2つとhtmlファイルが1つ作成されていると思います。
実行結果
上記のプログラムで作成したNDVI_season.htmlファイルを開くと、以下のようなグラフが表示されます。横軸が月で縦軸がNDVIの値になります。
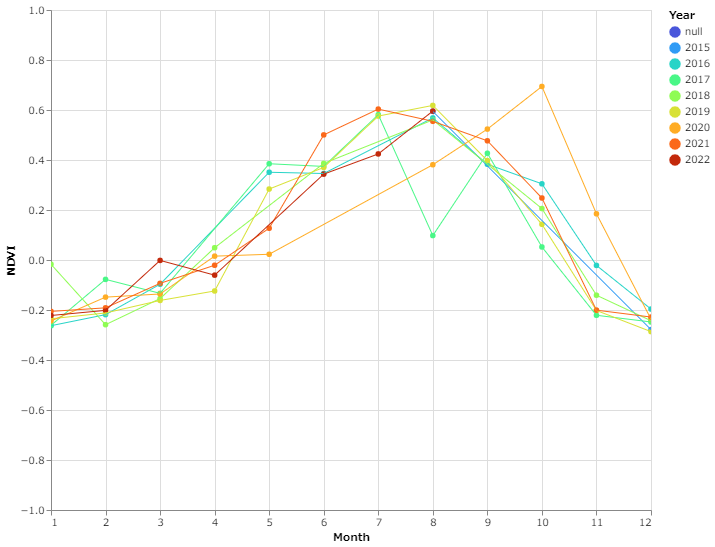
Sentinel-2が観測を始めた2015年からのデータしかありませんが、いずれの年も12~2月に負の値をとり、6~9月あたりにピークを迎えています。印旛沼に生息する水生植物のヒシは一年草で7~9月ごろに開花し最盛期を迎えることが知られています。今回作成したグラフ上でもその傾向が見て取れるため、ヒシを捉えたデータであるといえそうです。
今回のデータでは例えば2017年の8月や2020年の6-8月あたりがうまくデータがプロットされていません。原因は定かではありませんが、被雲率が20%以下のデータを用いているためフィルターされている可能性がありそうです。気象庁のHPで千葉県の日照時間を調べたところ、2017年8月の日照時間は101.8時間となっています。前後の年では、2016年で168.8時間、2018年で231.1時間の日照時間でした。そのため、2017年は曇りの日が多かった可能性があります。
先ほども説明しましたが、光学衛星は雲の影響を強く受けるため、気象状態によってはデータが取得できない期間があることが分かります。
コードの解説
import altair as alt
import ee
import pandas as pd
from altair_saver import savealtair、GEE、pandas、altair_saveのライブラリを使用します。ライブラリがない場合はpip installでインストールしましょう。
location_lonlat = [140.219017, 35.757631]
stat_region = ee.Geometry.Point(location_lonlat)
sentinel2 = ee.ImageCollection('COPERNICUS/S2_HARMONIZED').filterBounds(stat_region).filter(
ee.Filter.lt('CLOUDY_PIXEL_PERCENTAGE',20))ee.Geometry.Point()に経度、緯度を定義したlocation_lonlatを渡し、ee.ImageCollection.filterBounds()で観測地点の設定を行います。
これにより設定した地点が含まれるラスターデータの集まり(ImageCollection)が抽出できます。今回は千葉県の印旛沼を設定しています。
次にreduce_region_function()とfc_to_dict()について説明します。
# 関心領域を切り取る関数を定義
def reduce_region_function(img):
stat = img.reduceRegion(
reducer=ee.Reducer.mean(),
geometry=stat_region,
scale=10
)
return ee.Feature(stat_region, stat).set({'millis': img.date().millis()})
# GEEデータを辞書型に変換する関数を定義
def fc_to_dict(fc):
prop_names = fc.first().propertyNames()
prop_lists = fc.reduceColumns(
reducer=ee.Reducer.toList().repeat(prop_names.size()),
selectors=prop_names).get('list')
return ee.Dictionary.fromLists(prop_names, prop_lists)ImageCollection()で抽出したラスターデータは、解析したい地点以外にも多くの範囲を持っているため、解析には回しずらいです。そのため、reduce_region_function()で解析したい地点(関心領域と言います)をベクターデータ(Feature)を用いて切り取ります。
fc_to_dict()ではGEEのデータをprop_listsでリスト型に成形し、ee.Dictionaryで辞書型に変換しています。この操作でpandasの処理に回せるようになります。
# データフレーム成形用関数を定義
def add_date_info(df):
df['Timestamp'] = pd.to_datetime(df['millis'], unit='ms')
df['Year'] = pd.DatetimeIndex(df['Timestamp']).year
df['Month'] = pd.DatetimeIndex(df['Timestamp']).month
return df
# グラフ描画用関数を定義
def ndvi_season(data):
highlight = alt.selection(
type='single', on='mouseover', fields=['Year'], nearest=True)
base = alt.Chart(data).encode(
x=alt.X('Month:Q', scale=alt.Scale(domain=[1, 12])),
y=alt.Y('NDVI:Q', scale=alt.Scale(domain=[-1.0, 1.0])),
color=alt.Color('Year:O', scale=alt.Scale(scheme='turbo')))
points = base.mark_circle().encode(
opacity=alt.value(1),
tooltip=[
alt.Tooltip('Year:Q', title='Year'),
alt.Tooltip('Month:Q', title='Month'),
alt.Tooltip('NDVI:Q', title='NDVI')
]).add_selection(highlight)
lines = base.mark_line().encode(
size=alt.condition(~highlight, alt.value(1), alt.value(5)))
return (points + lines).properties(width=600, height=500).interactive()add_date_info()で衛星データの撮影時間がmillisとなっているため、年月日が入ったTimestanp型に変換します。さらにTimestanp型からグラフ作成用に年と月を抽出しています。
ndvi_season()ではaltairのライブラリを用いたグラフ描画用の設定を定義しています。baseでグラフの縦軸横軸、色合いを設定しています。pointsとlinesでグラフにカーソルを合わせた際の記述方法を設定しています。
# NDVIを計算
ndvi = sentinel2.map(calc_ndvi)
ndvi_stat_fc = ee.FeatureCollection(ndvi.map(reduce_region_function)).filter(
ee.Filter.notNull(ndvi.first().bandNames()))
# 衛星データを辞書型に変換
ndvi_dict = fc_to_dict(ndvi_stat_fc).getInfo()
ndvi_df = pd.DataFrame(ndvi_dict)
ndvi_df.to_csv('sentinel2_original.csv')
# 月ごとの中央値を計算
ndvi_df['timestamp'] = pd.to_datetime(ndvi_df['millis'], unit='ms')
ndvi_df.set_index('timestamp', inplace=True)
ndvi_df = ndvi_df.resample('M').median()
ndvi_df.to_csv('NDVI_season.csv')
# データフレームの成形
ndvi_df = add_date_info(ndvi_df)
# グラフの出力
save(ndvi_season(ndvi_df), 'NDVI_season.html')最後にNDVI計算結果をcsvとして出力し、グラフの描画結果をhtmlとして出力しています。
最後に
今回はある地点におけるNDVIの時系列変化をグラフ化しました。時系列変化は衛星データを用いたリモートセンシングの分野では基本的な解析ですが、読み取れる情報も多いためいろいろなことに応用できると思います。また、光学衛星ではNDVIだけではく、土壌や鉱物資源、水質の年別、季節別の変化もモニターできます。GEEはSentinel-2以外にも多くの衛星データが無料で取得できるので、いろいろと試していくと面白いと思います。

Pythonを使った衛星データ解析の初心者向け技術書を販売中!
¥2,500 → ¥800 今なら約70%OFF!!
Google Earth Engineを使えば簡単に
自宅にいながら地球環境の変動を観察できます



