本記事は創薬分野における低分子のin silico screeningについての記事です。標的化合物に対する薬候補化合物を見つけるときに使います。こちらの記事の内容ができるようになると、タンパク質の準備、低分子化合物ライブラリの準備、in silico screeningを通して一連のin silico創薬を体験できます。
ぜひ、トライしてみてください!
windows 64 bit, PyMOL 2.5.4. PyRx 0.8
自宅でできるin silico創薬の技術書を販売中
¥3,200 → ¥1500 今なら50%OFF!!
新薬探索を試したい方必読!
ITエンジニアである著者の視点から、wetな研究者からもdryの創薬研究をわかりやすく身近に感じられるように解説しています
自宅でできるin silico創薬の技術書を販売中
¥3,200 → ¥1,500 今なら50%OFF!!
分子ドッキングやMDシミュレーションなど、
自宅でできるin silico創薬の解析方法を解説したものになります!
In silico screeningとは?
In silico screening(インシリコスクリーニング)とは、バーチャルスクリーニングの一種で、コンピューターシミュレーションを使用して、大規模な分子ライブラリーから、特定の生物学的標的に対して有望な化合物を選別するプロセスです。
“In silico”とは、ラテン語の「in silicium(シリコン中)」に由来し、コンピューター上でのシミュレーションを意味しています。
In silico screeningでは、コンピューターモデルを用いて、大量の化合物の構造情報や生物学的活性を予測し、検討対象とするターゲットに対して最適な化合物を探索することができます。
このプロセスは、実験的に合成する前に、有望な化合物を選別し、合成の労力や費用を節約することができます。また、実験的なスクリーニングよりも高速であり、より多くの化合物を同時に検討できるため、薬物開発の効率を高めることができます。
In silico screeningは、薬剤の探索や、化学物質の特性予測、新しい材料の設計など、様々な分野で活用されています。
今回はパソコンのスペック上、大規模ライブラリーを構築することは難しいので、3つの化合物からなる小規模ライブラリを使ってin silico screeningを行います。
PyRxとは?
PyRxは、無料の分子ドッキングソフトウェアで、バーチャルスクリーニングを行うためのツールです。
分子ドッキングとは、薬剤とターゲット分子の相互作用をシミュレーションすることで、最も適した結合様式を予測することを指します。これにより、薬剤の設計や最適化に役立ちます。
PyRxでは、グラフィカルユーザーインターフェース(GUI)を介して、分子ドッキングの準備や実行、結果の解析を簡単に行うことができます。また、標準的なフォーマットの分子構造データを使用できるため、多くのデータベースから簡単に化合物を読み込むことができます。
PyRxは、無料で入手でき、Windows、macOS、Linuxなどの多くのオペレーティングシステムに対応しています。これにより、広く利用されるバーチャルスクリーニングツールとなっています。
本記事を進むにあたって、PyMOLのダウンロードをお願いします。
大阪大学の蛋白研究所からインストールの仕方が解説されています。
PyRxのinstall
まずPyRXのダウンロードページからwindows版のFree 0.8をダウンロードします。Mac版もありますが、現在のversionは対応していないので、注意してください。
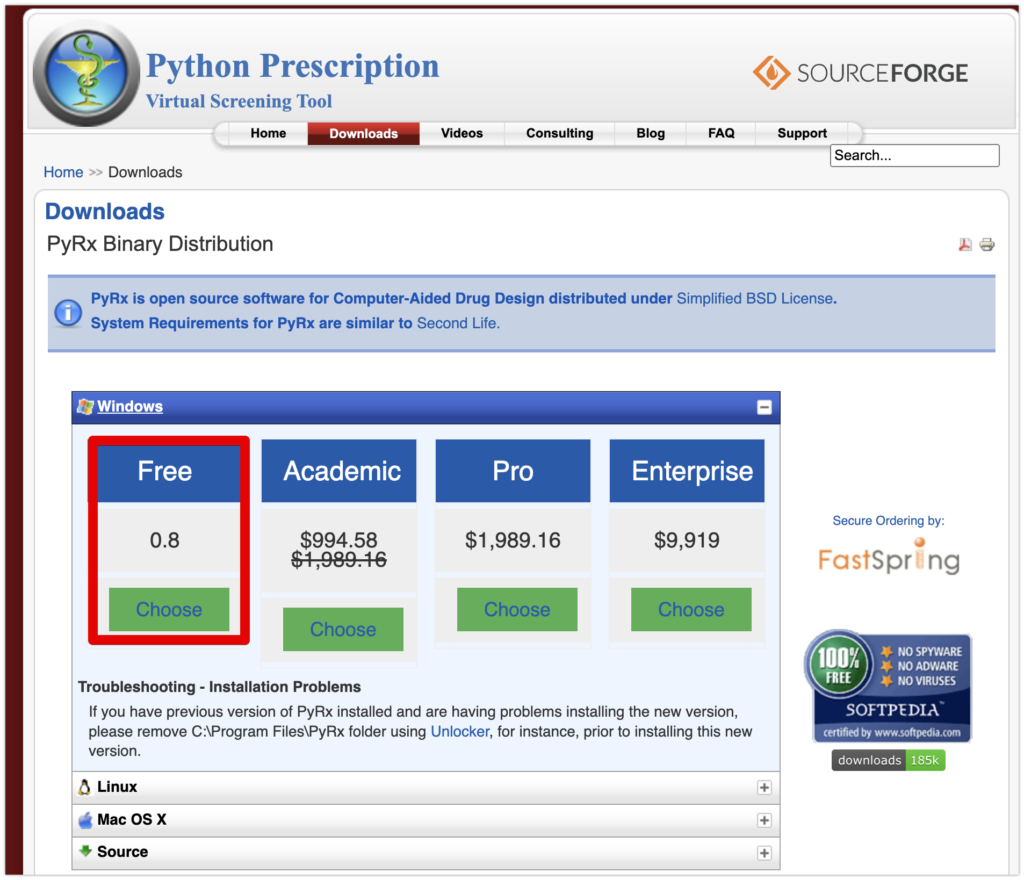
ダウンロードしたPyRx-0.8-Setup.exeからPyRxをインストールしてください。特に特別なことはせず、Nextを押してインストールしてください。
タンパク質の下準備
続いて、in silicoスクリーニングの標的の下準備をしていきます。Protein Data Bankのタンパク質の多くはそのバインダーとの複合体なので、それを取り除いていきます。今回は糖尿病の標的である、DPP4(pdb:2OQV)を例にして解説します。
まずpymolを開き、
FIle→GetPDB…をクリックし、PDB IDに2OQVを入れ、downloadを押し、pymol上にDPP4を表示させます。
続いてDPP4は二つの同じユニットの構成されるタンパク質なので、片方のユニットだけを指定します。
pymolのコマンドラインに
sele chain A
と記入し実行してください。sele chain Aとは、鎖IDがAである分子を選択するコマンドです。
また
color red, sele
と実行して、分かりやすいようにchain Aの色を赤くします。
Chain Aにはバインダーが含まれているので、これを消去します。
上のDisplay→Sequenceから配列を表示させ、一番右にあるMA9を選択し、
sele→remove atomsからバインダーを消去します。
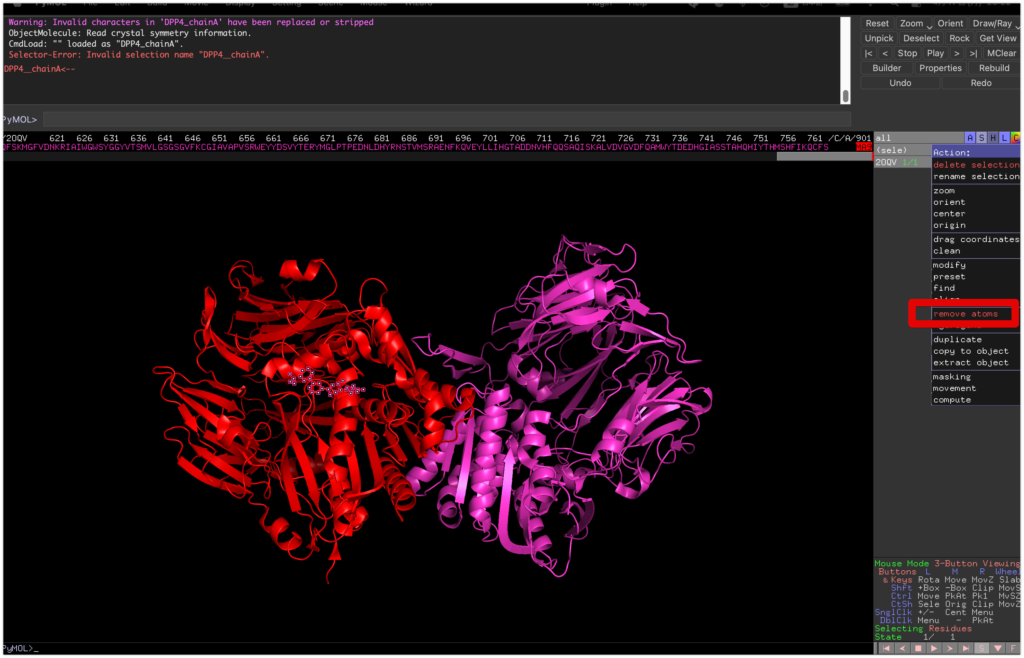
そして、再度sele chain Aからchain Aを選択し、
save DPP4_prep.pdb, sele
からDPP4_prep.pdbという名前でchain AをPDBファイルとして保存します。
これにてタンパク質の準備は終了です。
低分子化合物ライブラリの下準備
続いて、スクリーニング対象の低分子化合物ライブラリを作ります。やり方としては
- PubChemまたはZINCデータベースからの化合物データをSDFファイルの取得
- 構造データをpdbファイルに変換
となります。2のpdbファイルへの変換は本来やらなくてもよさそうですが、私がやった時にはうまくいかなかったので、pdbファイルに変換ををしています
1.PubChemまたはZINCデータベースからの化合物データをSDFファイルの取得
PubChemは、アメリカ合衆国の国立衛生研究所(NIH)の国立生物工学情報センター(NCBI)が管理する公開された化学データベースです。PubChemは、数百万の化学化合物の生物活性、物性、構造などの情報を提供する包括的なリソースです。
PubChemには、化学構造、化学物性、生物活性、安全性データ、科学文献の参照など、多岐にわたる化学データが含まれています。これは、化学、生化学、薬理学、毒性学などの分野での研究者、科学者、他の専門家によって幅広い用途で使用されています。例えば、新薬探索や化学研究、毒性評価などがあります。
PubChemには、化学名、化学構造、化学物性、生物活性などを使った検索など、複数の検索オプションがあります。また、化学データや生物データの解析、可視化、ダウンロードなどを行うためのツールも提供されています。
まずPubChemのサイトにいき、ライブラリの中に入れたい低分子化合物を取得します。
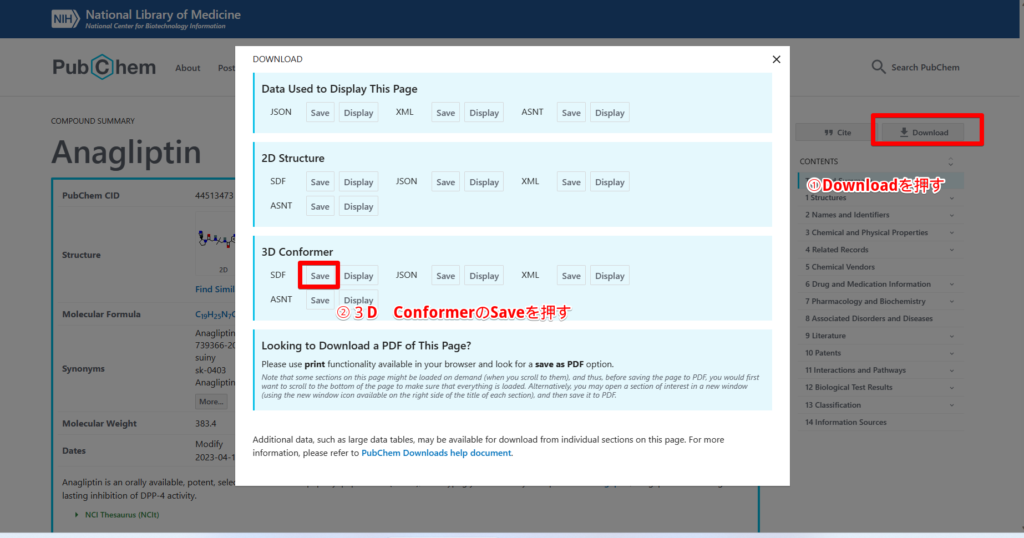
ここではすでにDPP4に対して作用があることが知られているAnagliptinを例に解説します。
Anagliptinを示すサイトに行き、右側のDownload→3D conformerのSDFを押し、SDFファイルの取得してください。
一方でZINCデータベースを用いても、PubChemのように化合物情報は取得できます。
ZINCデータベースは、化学化合物の仮想スクリーニングに使用される公開された化学データベースの一つです。化学化合物のデータや情報を提供し、新薬の探索や設計に役立ちます。
ZINCデータベースは、様々な化学化合物のデータを含んでおり、化学構造、物性、生物活性、参考文献などが含まれています。これにより、研究者や薬剤師は、様々な化学化合物の情報を簡単に検索し、必要なデータを入手することができます。
ZINCデータベースは、主に仮想スクリーニングという手法に使用されます。仮想スクリーニングは、コンピュータ上で膨大な数の化学化合物を高速にスクリーニングし、有望な化合物を選択する手法です。ZINCデータベースは、このような仮想スクリーニングに利用されることが多く、新薬の探索や設計を支援するための有用なツールとして広く使用されています。
ZINCデータベースのサイトにアクセスし、上記タブのSubstancesを押してください。
ここから望みの低分子化合物を取得できます。
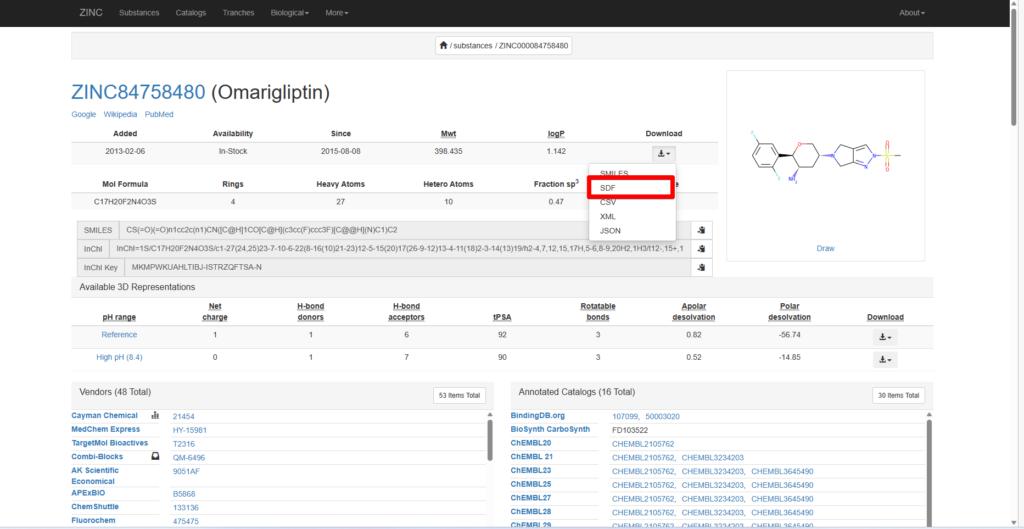
ここではすでにDPP4に対して作用があることが知られているOmarigliptinを例にして説明します。
SearchバーよりOmarigliptinを検索し、Omarigliptinを選択すると以下のページが出てきます。
ここからダウンロードを押し、SDFファイルを保存してください。
2.構造データをpdbファイルに変換
続いて、SDFファイルをpdbファイルに変換します。ダウンロードしたファイルをPyMOLで開きます。
File→Export Molecule…を押すと次のような画面が出てきます。

このままSaveを押し、PDB(**.*pdb **.*pdb.gz)を選択して、PDBファイルとしてデータを保存します。
これをライブラリのすべての化合物に対して行います。
In silico screening
今回はパソコンのスペック上、Anagliptin、Omarigliptin、Voglibose(糖尿病の別の標的であるα-グルコシダーゼ阻害薬)の3化合物でスクリーニングを行います。
まずProgram Files(×86)のPyRxからVBScriptファイルのPyRxを選択し、起動します。
起動すると以下のような画面が出てくると思います。
Moleculesの場所で右クリックを押し、Load Moleculeからタンパク質と低分子のPDBファイルをすべてloadします。
続いてDPP4_prepを右クリックし、AutoDock→Make Macromoleculeを押します。
続いて低分子のそれぞれをを右クリックし、AutoDock→Make ligandを押します。
AutoDockバーを見ると、それぞれがMacromoleculesと ligandsに入っていることがわかります。
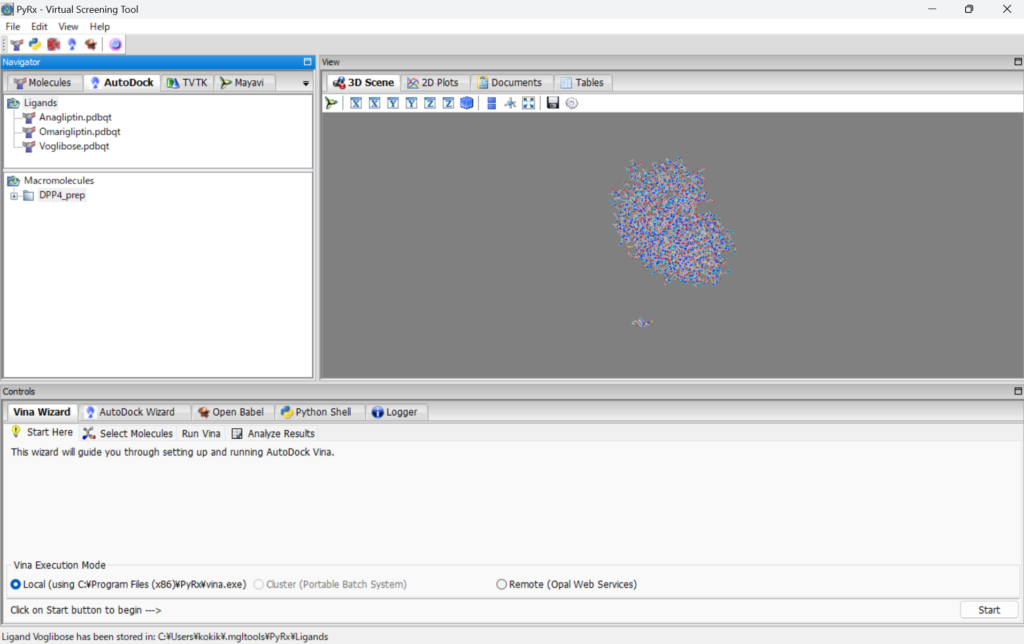
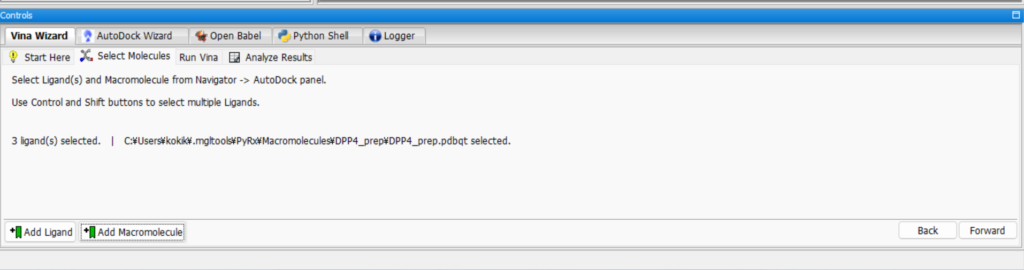
続いて、右下のstartを押し、低分子とタンパク質を選択します。すると以下のような表示になります。
このままForwardを押し、3D Sceneの四角を調節し、低分子の結合箇所を選びます。四角の中にタンパク質が入るようにすると、タンパク質の全場所から低分子が結合できる箇所を探し出すことができます。
その後、Forwardを押していくと、in silico screeningが開始されます。
以下のような画面になるので10分ほど待ちます。
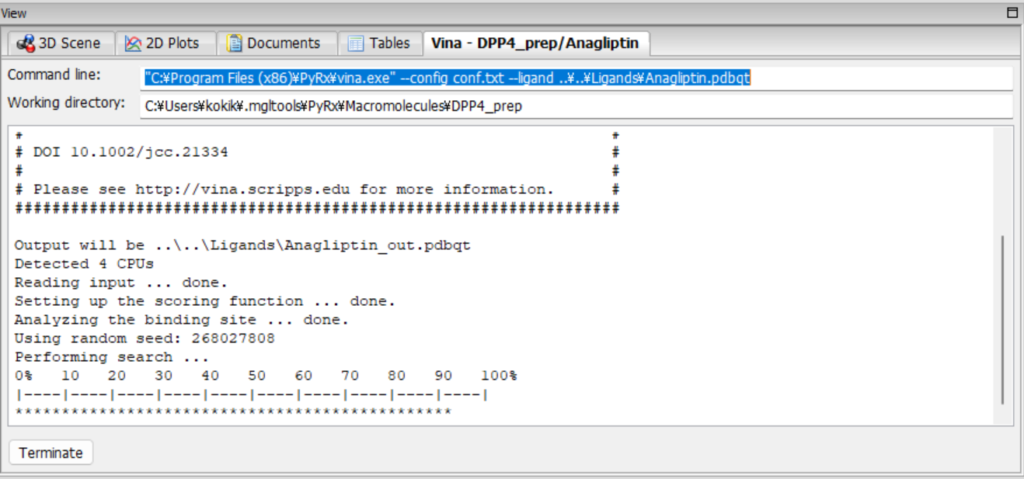
結果が出てきました。以下はDPP4とAnagliptinの結果を示しています。
この中では一番上のBinding Affinity(kcal/mol)が一番低いものが最も強く結合するものとして
表示されています。
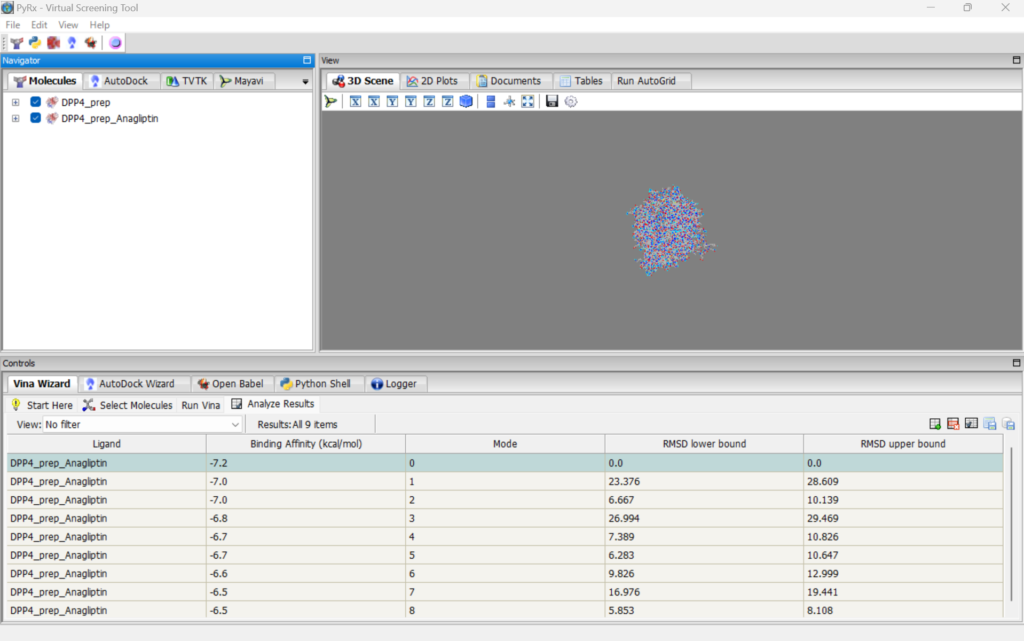
またライブラリの低分子の中で、Binding Affinity(kcal/mol)が最も低いものはAnagliptin:-7.2、Omarigliptin:-7.6、Voglibose:-5.4 kcal/molという結果だったので、結合力はOmarigliptin>Anagliptin>Vogliboseとなり、確かにすでに効果のある低分子化合物であるOmarigliptinとAnagliptinが高い結合力を有していることがわかります。
Pymolによる結合場所の可視化
では最後にスクリーニングした化合物がどこに結合しているかPymolで確認しましょう。
PyRxのEdit→preferencesのworkspaceを確認し、結果が出力されているか確認します。
C:\Users\kokik\.mgltools\PyRxにいき、Macromolecules→DPP4_prepに行くと、DPP4_prepやOmarigliptin_out、Anagliptin_out、Voglibose_outが生成しています。これらは最も結合力が高い箇所のそれぞれの低分子を表しています。これらをPyMolのFile→Openから選択し、表示させます。
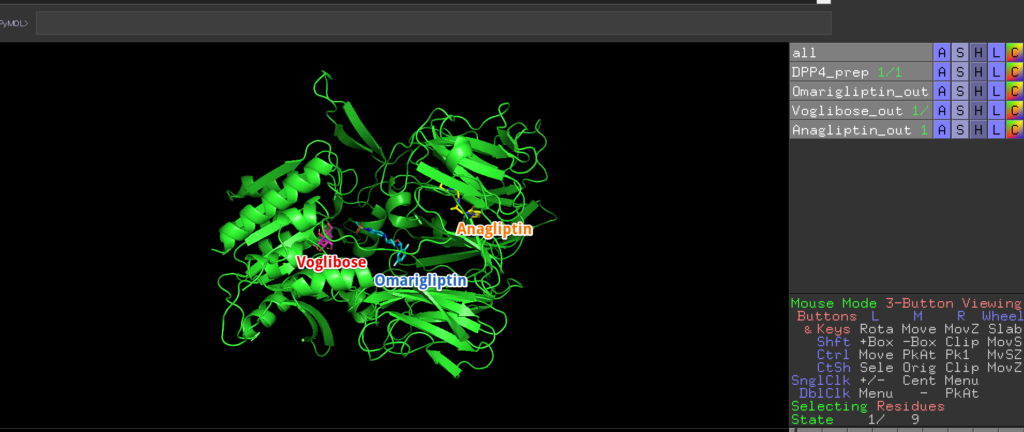
それぞれの結合サイトが確認できました!どれもDPP4内部に入っていてとても興味深いですね!
最後に
いかができたでしょうか? 今回は創薬手法の一つであるin silicoスクリーニングをご紹介しました。みなさんもお好きなタンパク質にお好きなライブラリを当てて、薬の候補化合物を探してみてください!
また今回利用しませんでしたが、ZINCデータベースの良いところは上記バーのCatalog→subsetsからすでにある化合物ライブラリが検索できることです。これによりFDAに認可された薬物などを一挙に検索できます。
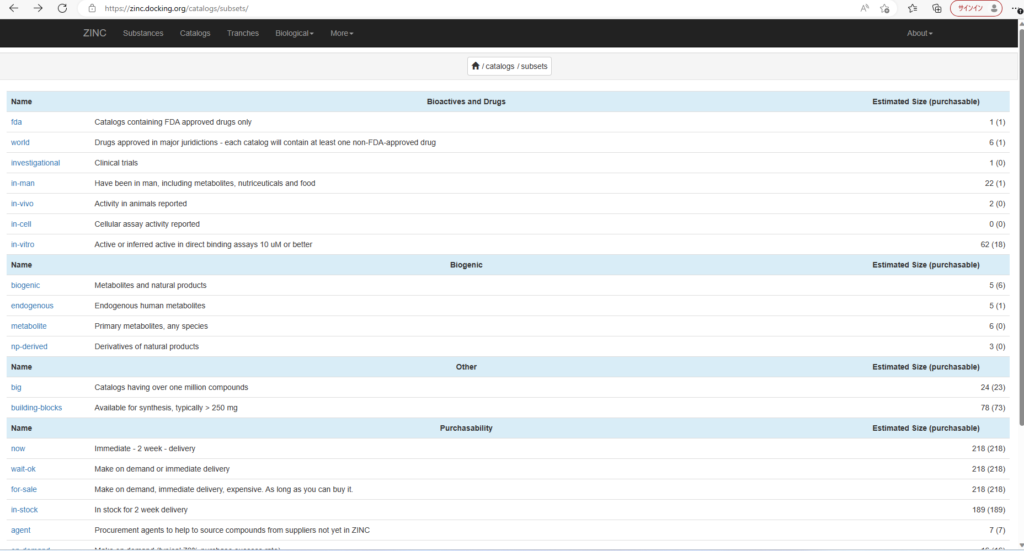
ここから得られた化合物により、一挙に大規模スクリーニングをすることも可能となります。パソコンのスペックが高い方はぜひ試してみてください!
参考動画
Protein-ligand virtual docking using PyRx | Computational biology | Bioinformatics | Akash Mitra
PyRx Tutorial || Multiple Ligand Docking || From Download to Result Analysis || All in One
PyRx – Virtual Screening Tool | Multiple Ligand Docking | Lecture 42 | Dr. Muhammad Naveed





